+検索条件
-Structure paper
| タイトル | Octomeric pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris. |
|---|---|
| ジャーナル・号・ページ | J Struct Biol, Vol. 159, Issue 1, Page 9-18, Year 2007 |
| 掲載日 | 2007年2月17日 |
 著者 著者 | Florian Garczarek / Ming Dong / Dieter Typke / H Ewa Witkowska / Terry C Hazen / Eva Nogales / Mark D Biggin / Robert M Glaeser /  |
| PubMed 要旨 | Pyruvate-ferredoxin oxidoreductatse (PFOR) carries out the central step in oxidative decarboxylation of pyruvate to acetyl-CoA. We have purified this enzyme from Desulfovibrio vulgaris Hildenborough ...Pyruvate-ferredoxin oxidoreductatse (PFOR) carries out the central step in oxidative decarboxylation of pyruvate to acetyl-CoA. We have purified this enzyme from Desulfovibrio vulgaris Hildenborough (DvH) as part of a systematic characterization of as many multiprotein complexes as possible for this organism, and the three-dimensional structure of this enzyme has been determined by a combination of electron microscopy (EM), single particle image analysis, homology modeling and computational molecular docking. Our results show that the 1MDa DvH PFOR complex is a homo-octomer, or more precisely, a tetramer of the dimeric form of the related enzyme found in Desulfovibrio africanus (Da), with which it shares a sequence identity of 69%. Our homology model of the DvH PFOR dimer is based on the Da PFOR X-ray structure. Docking of this model into our 17A resolution EM-reconstruction of negatively stained DvH PFOR octomers strongly suggests that the difference in oligomerization state for the two species is due to the insertion of a single valine residue (Val383) within a surface loop of the DvH enzyme. This study demonstrates that the strategy of intermediate resolution EM reconstruction coupled to homology modeling and docking can be powerful enough to infer the functionality of single amino acid residues. |
 リンク リンク |  J Struct Biol / J Struct Biol /  PubMed:17400475 PubMed:17400475 |
| 手法 | EM (単粒子) |
| 解像度 | 17.0 Å |
| 構造データ | 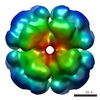 EMDB-1319: |
| 由来 |
|
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 Desulfovibrio vulgaris str. Hildenborough (バクテリア)
Desulfovibrio vulgaris str. Hildenborough (バクテリア)