+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8072 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Alcohol oxidase from Pichia pastoris | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | alcohol oxidase peroxisome / oxidoreductase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methane catabolic process / alcohol oxidase activity / alcohol oxidase / methanol metabolic process / peroxisomal matrix / flavin adenine dinucleotide binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Komagataella pastoris (菌類) Komagataella pastoris (菌類) | |||||||||
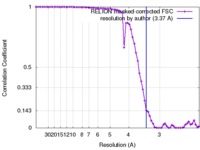
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.37 Å | |||||||||
 データ登録者 データ登録者 | Vonck J / Mills DJ | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2016 ジャーナル: PLoS One / 年: 2016タイトル: Structure of Alcohol Oxidase from Pichia pastoris by Cryo-Electron Microscopy. 著者: Janet Vonck / David N Parcej / Deryck J Mills /  要旨: The first step in methanol metabolism in methylotrophic yeasts, the oxidation of methanol and higher alcohols with molecular oxygen to formaldehyde and hydrogen peroxide, is catalysed by alcohol ...The first step in methanol metabolism in methylotrophic yeasts, the oxidation of methanol and higher alcohols with molecular oxygen to formaldehyde and hydrogen peroxide, is catalysed by alcohol oxidase (AOX), a 600-kDa homo-octamer containing eight FAD cofactors. When these yeasts are grown with methanol as the carbon source, AOX forms large crystalline arrays in peroxisomes. We determined the structure of AOX by cryo-electron microscopy at a resolution of 3.4 Å. All residues of the 662-amino acid polypeptide as well as the FAD are well resolved. AOX shows high structural homology to other members of the GMC family of oxidoreductases, which share a conserved FAD binding domain, but have different substrate specificities. The preference of AOX for small alcohols is explained by the presence of conserved bulky aromatic residues near the active site. Compared to the other GMC enzymes, AOX contains a large number of amino acid inserts, the longest being 75 residues. These segments are found at the periphery of the monomer and make extensive inter-subunit contacts which are responsible for the very stable octamer. A short surface helix forms contacts between two octamers, explaining the tendency of AOX to form crystals in the peroxisomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8072.map.gz emd_8072.map.gz | 25 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8072-v30.xml emd-8072-v30.xml emd-8072.xml emd-8072.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8072_fsc.xml emd_8072_fsc.xml | 6.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8072.png emd_8072.png | 259.2 KB | ||
| Filedesc metadata |  emd-8072.cif.gz emd-8072.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8072 http://ftp.pdbj.org/pub/emdb/structures/EMD-8072 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8072 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8072 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8072_validation.pdf.gz emd_8072_validation.pdf.gz | 316.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8072_full_validation.pdf.gz emd_8072_full_validation.pdf.gz | 315.6 KB | 表示 | |
| XML形式データ |  emd_8072_validation.xml.gz emd_8072_validation.xml.gz | 9.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8072 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8072 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8072 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8072 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8072.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8072.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.14 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : alcohol oxidase
| 全体 | 名称: alcohol oxidase |
|---|---|
| 要素 |
|
-超分子 #1: alcohol oxidase
| 超分子 | 名称: alcohol oxidase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 分子量 | 理論値: 600 KDa |
-分子 #1: Alcohol oxidase 1
| 分子 | 名称: Alcohol oxidase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: alcohol oxidase |
|---|---|
| 由来(天然) | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 分子量 | 理論値: 73.992195 KDa |
| 配列 | 文字列: MAIPEEFDIL VLGGGSSGSC IAGRLANLDH SLKVGLIEAG ENNLNNPWVY LPGIYPRNMK LDSKTASFYT SNPSPHLNGR RAIVPCANV LGGGSSINFM MYTRGSASDY DDFQAEGWKT KDLLPLMKKT ETYQRACNNP DIHGFEGPIK VSFGNYTYPV C QDFLRASE ...文字列: MAIPEEFDIL VLGGGSSGSC IAGRLANLDH SLKVGLIEAG ENNLNNPWVY LPGIYPRNMK LDSKTASFYT SNPSPHLNGR RAIVPCANV LGGGSSINFM MYTRGSASDY DDFQAEGWKT KDLLPLMKKT ETYQRACNNP DIHGFEGPIK VSFGNYTYPV C QDFLRASE SQGIPYVDDL EDLVTAHGAE HWLKWINRDT GRRSDSAHAF VHSTMRNHDN LYLICNTKVD KIIVEDGRAA AV RTVPSKP LNPKKPSHKI YRARKQIVLS CGTISSPLVL QRSGFGDPIK LRAAGVKPLV NLPGVGRNFQ DHYCFFSPYR IKP QYESFD DFVRGDAEIQ KRVFDQWYAN GTGPLATNGI EAGVKIRPTP EELSQMDESF QEGYREYFED KPDKPVMHYS IIAG FFGDH TKIPPGKYMT MFHFLEYPFS RGSIHITSPD PYAAPDFDPG FMNDERDMAP MVWAYKKSRE TARRMDHFAG EVTSH HPLF PYSSEARALE MDLETSNAYG GPLNLSAGLA HGSWTQPLKK PTAKNEGHVT SNQVELHPDI EYDEEDDKAI ENYIRE HTE TTWHCLGTCS IGPREGSKIV KWGGVLDHRS NVYGVKGLKV GDLSVCPDNV GCNTYTTALL IGEKTATLVG EDLGYSG EA LDMTVPQFKL GTYEKTGLAR F UniProtKB: Alcohol oxidase 1 |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: Potassium phosphate buffer, 50 mM |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 詳細: The grids had been cleaned in chloroform for 2 hrs. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK I / 詳細: blot for 11 seconds before plunging. |
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 詳細 | Data was collected manually |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 2-21 / 平均露光時間: 6.0 sec. / 平均電子線量: 51.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.6 µm / 倍率(補正後): 43860 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 4.2 mm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 147 | ||||||||
| 得られたモデル |  PDB-5i68: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)