+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | p97 (VCP) mutant - F539A with its adaptor | |||||||||
 マップデータ マップデータ | Post-processed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Hexameric complex / ATPase / Unfoldase / Protein Quality Control / Segregase / CHAPERONE | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Arie M / Matzov D / Karmona R / Szenkier N / Stanhill A / Navon A | |||||||||
| 資金援助 |  イスラエル, 1件 イスラエル, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: p97 (VCP) mutant - F539A with its adaptor 著者: Arie M / Matzov D / Karmona R / Szenkier N / Stanhill A / Navon A | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
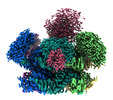
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18517.map.gz emd_18517.map.gz | 23.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18517-v30.xml emd-18517-v30.xml emd-18517.xml emd-18517.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18517.png emd_18517.png | 166.7 KB | ||
| Filedesc metadata |  emd-18517.cif.gz emd-18517.cif.gz | 6.2 KB | ||
| その他 |  emd_18517_additional_1.map.gz emd_18517_additional_1.map.gz emd_18517_half_map_1.map.gz emd_18517_half_map_1.map.gz emd_18517_half_map_2.map.gz emd_18517_half_map_2.map.gz | 139.9 MB 140.7 MB 140.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18517 http://ftp.pdbj.org/pub/emdb/structures/EMD-18517 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18517 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18517 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18517_validation.pdf.gz emd_18517_validation.pdf.gz | 841.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18517_full_validation.pdf.gz emd_18517_full_validation.pdf.gz | 840.8 KB | 表示 | |
| XML形式データ |  emd_18517_validation.xml.gz emd_18517_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_18517_validation.cif.gz emd_18517_validation.cif.gz | 17.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18517 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18517 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18517 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18517 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18517.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18517.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.793 Å | ||||||||||||||||||||||||||||||||||||
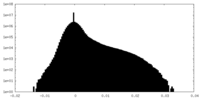
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_18517_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
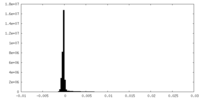
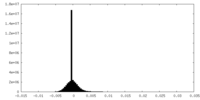
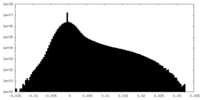
| 密度ヒストグラム |
-ハーフマップ: Half-map B
| ファイル | emd_18517_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
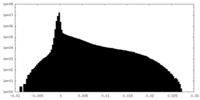
| 密度ヒストグラム |
-ハーフマップ: Half-map A
| ファイル | emd_18517_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
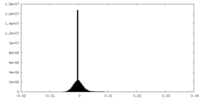
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tertiary complex of the hexameric p97 F539A mutant with its adaptor
| 全体 | 名称: Tertiary complex of the hexameric p97 F539A mutant with its adaptor |
|---|---|
| 要素 |
|
-超分子 #1: Tertiary complex of the hexameric p97 F539A mutant with its adaptor
| 超分子 | 名称: Tertiary complex of the hexameric p97 F539A mutant with its adaptor タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The mutant generated by the replacement of phenylalanine to alanine at position 539 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Transitional endoplasmic reticulum ATPase
| 分子 | 名称: Transitional endoplasmic reticulum ATPase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASGADSKGD DLSTAILKQK NRPNRLIVDE AINEDNSVVS LSQPKMDELQ LFRGDTVLLK GKKRREAVCI VLSDDTCSDE KIRMNRVVR NNLRVRLGDV ISIQPCPDVK YGKRIHVLPI DDTVEGITGN LFEVYLKPYF LEAYRPIRKG DIFLVRGGMR A VEFKVVET ...文字列: MASGADSKGD DLSTAILKQK NRPNRLIVDE AINEDNSVVS LSQPKMDELQ LFRGDTVLLK GKKRREAVCI VLSDDTCSDE KIRMNRVVR NNLRVRLGDV ISIQPCPDVK YGKRIHVLPI DDTVEGITGN LFEVYLKPYF LEAYRPIRKG DIFLVRGGMR A VEFKVVET DPSPYCIVAP DTVIHCEGEP IKREDEEESL NEVGYDDIGG CRKQLAQIKE MVELPLRHPA LFKAIGVKPP RG ILLYGPP GTGKTLIARA VANETGAFFF LINGPEIMSK LAGESESNLR KAFEEAEKNA PAIIFIDELD AIAPKREKTH GEV ERRIVS QLLTLMDGLK QRAHVIVMAA TNRPNSIDPA LRRFGRFDRE VDIGIPDATG RLEILQIHTK NMKLADDVDL EQVA NETHG HVGADLAALC SEAALQAIRK KMDLIDLEDE TIDAEVMNSL AVTMDDFRWA LSQSNPSALR ETVVEVPQVT WEDIG GLED VKRELQELVQ YPVEHPDKFL KFGMTPSKGV LFYGPPGCGK TLLAKAIANE CQANAISIKG PELLTMWFGE SEANVR EIF DKARQAAPCV LFFDELDSIA KARGGNIGDG GGAADRVINQ ILTEMDGMST KKNVFIIGAT NRPDIIDPAI LRPGRLD QL IYIPLPDEKS RVAILKANLR KSPVAKDVDL EFLAKMTNGF SGADLTEICQ RACKLAIRES IESEIRRERE RQTNPSAM E VEEDDPVPEI RRDHFEEAMR FARRSVSDND IRKYEMFAQT LQQSRGFGSF RFPSGNQGGA GPSQGSGGGT GGSVYTEDN DDDLYG |
-分子 #2: Nuclear protein localization protein 4 homolog
| 分子 | 名称: Nuclear protein localization protein 4 homolog / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAEGTIIRVQ SPDGVKRITA TKRETAATFL KKVAKEFGFQ NNGFSVYINR NKTGEITASS SKSLHLLKI KHGDLLFLFP SSLAGPSSEM ETSTSVGLKA FGAPHVVEDE IDQYLSKQDG K IYRSRDPQ LCRHGPLGKC VHCVPLEPFD EDYLNHLEPP VKHMSFHAYI ...文字列: MAEGTIIRVQ SPDGVKRITA TKRETAATFL KKVAKEFGFQ NNGFSVYINR NKTGEITASS SKSLHLLKI KHGDLLFLFP SSLAGPSSEM ETSTSVGLKA FGAPHVVEDE IDQYLSKQDG K IYRSRDPQ LCRHGPLGKC VHCVPLEPFD EDYLNHLEPP VKHMSFHAYI RKLTGGADKG KF VALENIS CKIKSGCEGH LPWPNGICTK CQPSAITLNR QKYRHVDNIM FENHTVADRF LDF WRKTGN QHFGYLYGRY TEHKDIPLGI RAEVAAIYEP PQIGTQNSLE LLEDPKAEVV DEIA SKLGL RKVGWIFTDL VSEDTRKGTV RYSRNKDTYF LSSEECITAG DFQNKHPNIC RLSPD GHFG SKFVTAVATG GPDNQVHFEG YQVSNQCMAL VRDECLLPCK DAPELGYAKE SSSEQY VPD VFYKDIDKFG NEITQLARPL PVEYLIIDIT TTFPKDPVYT FSISQNPFPI ENRDVLG ET QDFHSLATYL SQNTSSVFLD TISDFHLLLF LVTNEVMPLQ DSISLLLEAV RTRNEELA Q TWKKSEQWAT IEQLCSTVGV QLPGLHEFGA VGGSARAATS AMWACQHCTF MNQPGTGHC EMCSLPRT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: pre-blotting incubation time of 20 seconds and blot for 3.5 seconds with -1 blot force. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 11807 / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: FROM EMDB |
|---|---|
| 精密化 | 温度因子: 104 |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)