+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
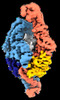
| タイトル | MutSbeta bound to 61bp homoduplex DNA | |||||||||
 マップデータ マップデータ | MutSbeta bound to 61bp homoduplex DNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Protein-DNA complex / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報somatic recombination of immunoglobulin genes involved in immune response / MutSbeta complex / Defective Mismatch Repair Associated With MSH3 / MutSalpha complex / Defective Mismatch Repair Associated With MSH2 / Defective Mismatch Repair Associated With MSH6 / guanine/thymine mispair binding / somatic recombination of immunoglobulin gene segments / B cell mediated immunity / maintenance of DNA repeat elements ...somatic recombination of immunoglobulin genes involved in immune response / MutSbeta complex / Defective Mismatch Repair Associated With MSH3 / MutSalpha complex / Defective Mismatch Repair Associated With MSH2 / Defective Mismatch Repair Associated With MSH6 / guanine/thymine mispair binding / somatic recombination of immunoglobulin gene segments / B cell mediated immunity / maintenance of DNA repeat elements / positive regulation of helicase activity / positive regulation of isotype switching to IgA isotypes / centromeric DNA binding / mitotic recombination / positive regulation of isotype switching to IgG isotypes / mismatched DNA binding / negative regulation of DNA recombination / Mismatch repair (MMR) directed by MSH2:MSH3 (MutSbeta) / Mismatch repair (MMR) directed by MSH2:MSH6 (MutSalpha) / isotype switching / oxidative phosphorylation / response to UV-B / postreplication repair / mitotic intra-S DNA damage checkpoint signaling / ATP-dependent DNA damage sensor activity / germ cell development / response to X-ray / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / somatic hypermutation of immunoglobulin genes / mismatch repair / ATP-dependent activity, acting on DNA / protein localization to chromatin / B cell differentiation / determination of adult lifespan / TP53 Regulates Transcription of DNA Repair Genes / male gonad development / double-strand break repair / double-stranded DNA binding / in utero embryonic development / negative regulation of neuron apoptotic process / damaged DNA binding / chromosome, telomeric region / DNA repair / chromatin binding / enzyme binding / protein homodimerization activity / ATP hydrolysis activity / DNA binding / nucleoplasm / ATP binding / membrane / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.29 Å | |||||||||
 データ登録者 データ登録者 | Lee J-H / Thomsen M / Daub H / Steinbacher S / Sztyler A / Thieulin-Pardo G / Neudegger T / Plotnikov N / Iyer RR / Wilkinson H ...Lee J-H / Thomsen M / Daub H / Steinbacher S / Sztyler A / Thieulin-Pardo G / Neudegger T / Plotnikov N / Iyer RR / Wilkinson H / Monteagudo E / Felsenfeld DP / Haque T / Finley M / Dominguez C / Vogt TF / Prasad BC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: MutSbeta bound to 61bp homoduplex DNA 著者: Lee J-H / Thomsen M / Daub H / Steinbacher S / Sztyler A / Thieulin-Pardo G / Neudegger T / Plotnikov N / Iyer RR / Wilkinson H / Monteagudo E / Felsenfeld DP / Haque T / Finley M / Dominguez ...著者: Lee J-H / Thomsen M / Daub H / Steinbacher S / Sztyler A / Thieulin-Pardo G / Neudegger T / Plotnikov N / Iyer RR / Wilkinson H / Monteagudo E / Felsenfeld DP / Haque T / Finley M / Dominguez C / Vogt TF / Prasad BC | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16972.map.gz emd_16972.map.gz | 111.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16972-v30.xml emd-16972-v30.xml emd-16972.xml emd-16972.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16972.png emd_16972.png | 19.2 KB | ||
| Filedesc metadata |  emd-16972.cif.gz emd-16972.cif.gz | 6.9 KB | ||
| その他 |  emd_16972_half_map_1.map.gz emd_16972_half_map_1.map.gz emd_16972_half_map_2.map.gz emd_16972_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16972 http://ftp.pdbj.org/pub/emdb/structures/EMD-16972 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16972 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16972 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16972_validation.pdf.gz emd_16972_validation.pdf.gz | 589.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16972_full_validation.pdf.gz emd_16972_full_validation.pdf.gz | 588.6 KB | 表示 | |
| XML形式データ |  emd_16972_validation.xml.gz emd_16972_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_16972_validation.cif.gz emd_16972_validation.cif.gz | 16.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16972 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16972 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16972 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16972 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8omaMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16972.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16972.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MutSbeta bound to 61bp homoduplex DNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9142 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: MutSbeta bound to 61bp homoduplex DNA
| ファイル | emd_16972_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MutSbeta bound to 61bp homoduplex DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
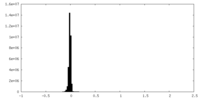
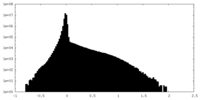
| 密度ヒストグラム |
-ハーフマップ: MutSbeta bound to 61bp homoduplex DNA
| ファイル | emd_16972_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MutSbeta bound to 61bp homoduplex DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
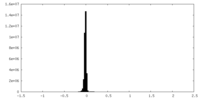
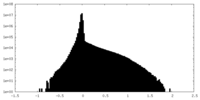
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MutSbeta bound to 61bp homoduplex DNA
| 全体 | 名称: MutSbeta bound to 61bp homoduplex DNA |
|---|---|
| 要素 |
|
-超分子 #1: MutSbeta bound to 61bp homoduplex DNA
| 超分子 | 名称: MutSbeta bound to 61bp homoduplex DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DNA1
| 分子 | 名称: DNA1 / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5.242409 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(DA)(DA)(DG)(DC)(DT)(DT)(DA) (DG)(DC)(DT)(DT)(DA)(DG)(DG) |
-分子 #2: DNA2
| 分子 | 名称: DNA2 / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5.171374 KDa |
| 配列 | 文字列: (DC)(DC)(DT)(DA)(DA)(DG)(DC)(DT)(DA)(DA) (DG)(DC)(DT)(DT)(DC)(DA)(DG) |
-分子 #3: DNA mismatch repair protein Msh2
| 分子 | 名称: DNA mismatch repair protein Msh2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 104.861875 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVQPKETLQ LESAAEVGFV RFFQGMPEKP TTTVRLFDRG DFYTAHGEDA LLAAREVFKT QGVIKYMGPA GAKNLQSVVL SKMNFESFV KDLLLVRQYR VEVYKNRAGN KASKENDWYL AYKASPGNLS QFEDILFGNN DMSASIGVVG VKMSAVDGQR Q VGVGYVDS ...文字列: MAVQPKETLQ LESAAEVGFV RFFQGMPEKP TTTVRLFDRG DFYTAHGEDA LLAAREVFKT QGVIKYMGPA GAKNLQSVVL SKMNFESFV KDLLLVRQYR VEVYKNRAGN KASKENDWYL AYKASPGNLS QFEDILFGNN DMSASIGVVG VKMSAVDGQR Q VGVGYVDS IQRKLGLCEF PDNDQFSNLE ALLIQIGPKE CVLPGGETAG DMGKLRQIIQ RGGILITERK KADFSTKDIY QD LNRLLKG KKGEQMNSAV LPEMENQVAV SSLSAVIKFL ELLSDDSNFG QFELTTFDFS QYMKLDIAAV RALNLFQGSV EDT TGSQSL AALLNKCKTP QGQRLVNQWI KQPLMDKNRI EERLNLVEAF VEDAELRQTL QEDLLRRFPD LNRLAKKFQR QAAN LQDCY RLYQGINQLP NVIQALEKHE GKHQKLLLAV FVTPLTDLRS DFSKFQEMIE TTLDMDQVEN HEFLVKPSFD PNLSE LREI MNDLEKKMQS TLISAARDLG LDPGKQIKLD SSAQFGYYFR VTCKEEKVLR NNKNFSTVDI QKNGVKFTNS KLTSLN EEY TKNKTEYEEA QDAIVKEIVN ISSGYVEPMQ TLNDVLAQLD AVVSFAHVSN GAPVPYVRPA ILEKGQGRII LKASRHA CV EVQDEIAFIP NDVYFEKDKQ MFHIITGPNM GGKSTYIRQT GVIVLMAQIG CFVPCESAEV SIVDCILARV GAGDSQLK G VSTFMAEMLE TASILRSATK DSLIIIDELG RGTSTYDGFG LAWAISEYIA TKIGAFCMFA THFHELTALA NQIPTVNNL HVTALTTEET LTMLYQVKKG VCDQSFGIHV AELANFPKHV IECAKQKALE LEEFQYIGES QGYDIMEPAA KKCYLEREQG EKIIQEFLS KVKQMPFTEM SEENITIKLK QLKAEVIAKN NSFVNEIISR IKVTT UniProtKB: DNA mismatch repair protein Msh2 |
-分子 #4: DNA mismatch repair protein Msh3
| 分子 | 名称: DNA mismatch repair protein Msh3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 127.618703 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSRRKPASGG LAASSSAPAR QAVLSRFFQS TGSLKSTSSS TGAADQVDPG AAAAAAAAAA AAPPAPPAPA FPPQLPPHIA TEIDRRKKR PLENDGPVKK KVKKVQQKEG GSDLGMSGNS EPKKCLRTRN VSKSLEKLKE FCCDSALPQS RVQTESLQER F AVLPKCTD ...文字列: MSRRKPASGG LAASSSAPAR QAVLSRFFQS TGSLKSTSSS TGAADQVDPG AAAAAAAAAA AAPPAPPAPA FPPQLPPHIA TEIDRRKKR PLENDGPVKK KVKKVQQKEG GSDLGMSGNS EPKKCLRTRN VSKSLEKLKE FCCDSALPQS RVQTESLQER F AVLPKCTD FDDISLLHAK NAVSSEDSKR QINQKDTTLF DLSQFGSSNT SHENLQKTAS KSANKRSKSI YTPLELQYIE MK QQHKDAV LCVECGYKYR FFGEDAEIAA RELNIYCHLD HNFMTASIPT HRLFVHVRRL VAKGYKVGVV KQTETAALKA IGD NRSSLF SRKLTALYTK STLIGEDVNP LIKLDDAVNV DEIMTDTSTS YLLCISENKE NVRDKKKGNI FIGIVGVQPA TGEV VFDSF QDSASRSELE TRMSSLQPVE LLLPSALSEQ TEALIHRATS VSVQDDRIRV ERMDNIYFEY SHAFQAVTEF YAKDT VDIK GSQIISGIVN LEKPVICSLA AIIKYLKEFN LEKMLSKPEN FKQLSSKMEF MTINGTTLRN LEILQNQTDM KTKGSL LWV LDHTKTSFGR RKLKKWVTQP LLKLREINAR LDAVSEVLHS ESSVFGQIEN HLRKLPDIER GLCSIYHKKC STQEFFL IV KTLYHLKSEF QAIIPAVNSH IQSDLLRTVI LEIPELLSPV EHYLKILNEQ AAKVGDKTEL FKDLSDFPLI KKRKDEIQ G VIDEIRMHLQ EIRKILKNPS AQYVTVSGQE FMIEIKNSAV SCIPTDWVKV GSTKAVSRFH SPFIVENYRH LNQLREQLV LDCSAEWLDF LEKFSEHYHS LCKAVHHLAT VDCIFSLAKV AKQGDYCRPT VQEERKIVIK NGRHPVIDVL LGEQDQYVPN NTDLSEDSE RVMIITGPNM GGKSSYIKQV ALITIMAQIG SYVPAEEATI GIVDGIFTRM GAADNIYKGR STFMEELTDT A EIIRKATS QSLVILDELG RGTSTHDGIA IAYATLEYFI RDVKSLTLFV THYPPVCELE KNYSHQVGNY HMGFLVSEDE SK LDPGAAE QVPDFVTFLY QITRGIAARS YGLNVAKLAD VPGEILKKAA HKSKELEGLI NTKRKRLKYF AKLWTMHNAQ DLQ KWTEEF NMEETQTSLL H UniProtKB: DNA mismatch repair protein Msh3 |
-分子 #5: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 56.8 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.29 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 226539 |
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)