+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | rotor of the Trypanosoma brucei mitochondrial ATP synthase dimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATP synthase / mitochondria / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報H+-transporting two-sector ATPase / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する / : / : / : / proton motive force-driven ATP synthesis / proton-transporting ATP synthase complex, catalytic core F(1) / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / mitochondrial inner membrane ...H+-transporting two-sector ATPase / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する / : / : / : / proton motive force-driven ATP synthesis / proton-transporting ATP synthase complex, catalytic core F(1) / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / mitochondrial inner membrane / hydrolase activity / lipid binding / mitochondrion 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Muehleip A / Gahura O / Zikova A / Amunts A | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: An ancestral interaction module promotes oligomerization in divergent mitochondrial ATP synthases. 著者: Ondřej Gahura / Alexander Mühleip / Carolina Hierro-Yap / Brian Panicucci / Minal Jain / David Hollaus / Martina Slapničková / Alena Zíková / Alexey Amunts /   要旨: Mitochondrial ATP synthase forms stable dimers arranged into oligomeric assemblies that generate the inner-membrane curvature essential for efficient energy conversion. Here, we report cryo-EM ...Mitochondrial ATP synthase forms stable dimers arranged into oligomeric assemblies that generate the inner-membrane curvature essential for efficient energy conversion. Here, we report cryo-EM structures of the intact ATP synthase dimer from Trypanosoma brucei in ten different rotational states. The model consists of 25 subunits, including nine lineage-specific, as well as 36 lipids. The rotary mechanism is influenced by the divergent peripheral stalk, conferring a greater conformational flexibility. Proton transfer in the lumenal half-channel occurs via a chain of five ordered water molecules. The dimerization interface is formed by subunit-g that is critical for interactions but not for the catalytic activity. Although overall dimer architecture varies among eukaryotes, we find that subunit-g together with subunit-e form an ancestral oligomerization motif, which is shared between the trypanosomal and mammalian lineages. Therefore, our data defines the subunit-g/e module as a structural component determining ATP synthase oligomeric assemblies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15562.map.gz emd_15562.map.gz | 543.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15562-v30.xml emd-15562-v30.xml emd-15562.xml emd-15562.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15562_fsc.xml emd_15562_fsc.xml | 19.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15562.png emd_15562.png | 53.8 KB | ||
| マスクデータ |  emd_15562_msk_1.map emd_15562_msk_1.map | 669.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15562.cif.gz emd-15562.cif.gz | 5.7 KB | ||
| その他 |  emd_15562_half_map_1.map.gz emd_15562_half_map_1.map.gz emd_15562_half_map_2.map.gz emd_15562_half_map_2.map.gz | 537.6 MB 537.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15562 http://ftp.pdbj.org/pub/emdb/structures/EMD-15562 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15562 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15562 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15562_validation.pdf.gz emd_15562_validation.pdf.gz | 892.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15562_full_validation.pdf.gz emd_15562_full_validation.pdf.gz | 892.1 KB | 表示 | |
| XML形式データ |  emd_15562_validation.xml.gz emd_15562_validation.xml.gz | 27.6 KB | 表示 | |
| CIF形式データ |  emd_15562_validation.cif.gz emd_15562_validation.cif.gz | 37 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15562 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15562 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15562 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15562 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ap9MC  8ap6C  8ap7C  8ap8C  8apaC  8apbC  8apcC  8apdC  8apeC  8apfC  8apgC  8aphC  8apjC  8apkC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15562.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15562.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
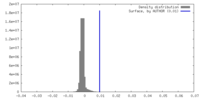
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15562_msk_1.map emd_15562_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
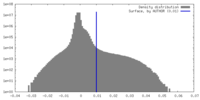
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15562_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
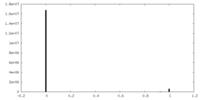
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15562_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
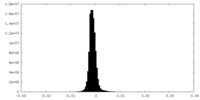
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : mitochondrial ATP synthase dimer from Trypanosoma brucei
| 全体 | 名称: mitochondrial ATP synthase dimer from Trypanosoma brucei |
|---|---|
| 要素 |
|
-超分子 #1: mitochondrial ATP synthase dimer from Trypanosoma brucei
| 超分子 | 名称: mitochondrial ATP synthase dimer from Trypanosoma brucei タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  株: Lister 427 / Organelle: Mitochondrion |
-分子 #1: ATP synthase gamma subunit
| 分子 | 名称: ATP synthase gamma subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: H+-transporting two-sector ATPase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.472254 KDa |
| 配列 | 文字列: MSGKLRLYKE KLEGYNRFYS IVKTIKMVTL AKYRAAQGRI RTRDFSLRYT ELAFSKPQAS RDAVVAAKNA LVYIPITTNR GSCGALNSN IVRCIDSVVS SKMVLMPVGK RGIDSFSKLY PDEFRYGIIN DMKESMHFGY ATFVIENAYE VSKDADRYQV I FNRFVSAG ...文字列: MSGKLRLYKE KLEGYNRFYS IVKTIKMVTL AKYRAAQGRI RTRDFSLRYT ELAFSKPQAS RDAVVAAKNA LVYIPITTNR GSCGALNSN IVRCIDSVVS SKMVLMPVGK RGIDSFSKLY PDEFRYGIIN DMKESMHFGY ATFVIENAYE VSKDADRYQV I FNRFVSAG VQRNAVYNIP SYEKWKEDLA DAASSDNQKN RYLFANALQN EEEQLIRDFF DFHAALAVLN AVGENELSEQ AA RLVAVEG QLTNISSLQQ RTSSLYNKTR QFGITAALIE ILSAMSSLEG NAMKGVRRNK FWEGAVTK UniProtKB: ATP synthase subunit gamma, mitochondrial |
-分子 #2: ATP synthase, epsilon chain, putative
| 分子 | 名称: ATP synthase, epsilon chain, putative / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する |
|---|---|
| 由来(天然) | 生物種:  株: 927/4 GUTat10.1 |
| 分子量 | 理論値: 20.172938 KDa |
| 配列 | 文字列: MFRTFGRRLV SCTLPLLQSA PHDLPEGFEF MEHKVVNKDI HAPHENLETL RLTLTRQDEF LLREEPVKCV TVTGTNGEYG IYPGHAYKI VQLNPSPLTV EYTDGTTKKY FVSGGFAHIN NEGSCDVNTV ECTLLDDLDL AIAEKELAAQ QAALGSAKDD K AKSVVEIR ISVIEAVIAA LKHH UniProtKB: ATP synthase, epsilon chain, putative |
-分子 #3: ATP synthase subunit epsilon, mitochondrial
| 分子 | 名称: ATP synthase subunit epsilon, mitochondrial / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.70689 KDa |
| 配列 | 文字列: MIRRSCALLS SSWRDHGISY LKYLNVCTET LHSTVKESRR AKYERWSKPC YTAQRPDGAG GQETIDKVPI HTKDY UniProtKB: ATP synthase subunit epsilon, mitochondrial |
-分子 #4: ATPase subunit 9, putative
| 分子 | 名称: ATPase subunit 9, putative / タイプ: protein_or_peptide / ID: 4 / コピー数: 10 / 光学異性体: LEVO / EC番号: H+-transporting two-sector ATPase |
|---|---|
| 由来(天然) | 生物種:  株: 927/4 GUTat10.1 |
| 分子量 | 理論値: 12.39887 KDa |
| 配列 | 文字列: MMRRLALQSS IRRATPFATP LVASTKALNP MCSAITIREA STVAISVQGL HYVGTGLAAI ALAGVGLGIG TIFGNLLVAC ARQPNLTKM LFNYAILGFA LTEAIGLFAL MLAFLMLFS UniProtKB: ATPase subunit 9, putative |
-分子 #5: URIDINE 5'-TRIPHOSPHATE
| 分子 | 名称: URIDINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: UTP |
|---|---|
| 分子量 | 理論値: 484.141 Da |
| Chemical component information |  ChemComp-UTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 33.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 1.6 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)