+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Saccharomyces cerevisiae TOROID (TORC1 Organized in Inhibited Domains). | |||||||||||||||
 マップデータ マップデータ | Main EM map of TORC1 in TOROID (TORC1 Organized in Inhibited Domains), obtained by Single Particle Analysis (SPA) after signal subtraction performed on TOROID filaments. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | TARGET OF RAPAMYCIN / TORC1 / KOG1 / LST8 / TOR2 / FRB DOMAIN / SIGNALING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報PIP3 activates AKT signaling / CD28 dependent PI3K/Akt signaling / regulation of snRNA pseudouridine synthesis / mitochondria-nucleus signaling pathway / Regulation of TP53 Degradation / 1-phosphatidylinositol 4-kinase / 1-phosphatidylinositol 4-kinase activity / establishment or maintenance of actin cytoskeleton polarity / HSF1-dependent transactivation / VEGFR2 mediated vascular permeability ...PIP3 activates AKT signaling / CD28 dependent PI3K/Akt signaling / regulation of snRNA pseudouridine synthesis / mitochondria-nucleus signaling pathway / Regulation of TP53 Degradation / 1-phosphatidylinositol 4-kinase / 1-phosphatidylinositol 4-kinase activity / establishment or maintenance of actin cytoskeleton polarity / HSF1-dependent transactivation / VEGFR2 mediated vascular permeability / fungal-type cell wall organization / Amino acids regulate mTORC1 / TORC2 complex / TORC1 complex / TORC1 signaling / fungal-type vacuole membrane / cellular response to nitrogen starvation / vacuolar membrane / positive regulation of Rho protein signal transduction / negative regulation of macroautophagy / positive regulation of endocytosis / TOR signaling / cytoskeleton organization / cellular response to starvation / protein serine/threonine kinase activator activity / nuclear periphery / negative regulation of autophagy / response to nutrient / ubiquitin binding / regulation of autophagy / regulation of cell growth / regulation of actin cytoskeleton organization / cellular response to amino acid stimulus / cytoplasmic stress granule / ribosome biogenesis / protein-macromolecule adaptor activity / positive regulation of cell growth / non-specific serine/threonine protein kinase / regulation of cell cycle / endosome membrane / Golgi membrane / protein serine kinase activity / protein serine/threonine kinase activity / protein-containing complex binding / signal transduction / mitochondrion / ATP binding / nucleus / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
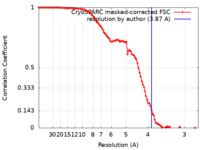
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.87 Å | |||||||||||||||
 データ登録者 データ登録者 | Felix J / Prouteau M / Bourgoint C / Bonadei L / Desfosses A / Gabus C / Sadian Y / Savvides SN / Gutsche I / Loewith R | |||||||||||||||
| 資金援助 | European Union, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: EGOC inhibits TOROID polymerization by structurally activating TORC1. 著者: Manoël Prouteau / Clélia Bourgoint / Jan Felix / Lenny Bonadei / Yashar Sadian / Caroline Gabus / Savvas N Savvides / Irina Gutsche / Ambroise Desfosses / Robbie Loewith /    要旨: Target of rapamycin complex 1 (TORC1) is a protein kinase controlling cell homeostasis and growth in response to nutrients and stresses. In Saccharomyces cerevisiae, glucose depletion triggers a ...Target of rapamycin complex 1 (TORC1) is a protein kinase controlling cell homeostasis and growth in response to nutrients and stresses. In Saccharomyces cerevisiae, glucose depletion triggers a redistribution of TORC1 from a dispersed localization over the vacuole surface into a large, inactive condensate called TOROID (TORC1 organized in inhibited domains). However, the mechanisms governing this transition have been unclear. Here, we show that acute depletion and repletion of EGO complex (EGOC) activity is sufficient to control TOROID distribution, independently of other nutrient-signaling pathways. The 3.9-Å-resolution structure of TORC1 from TOROID cryo-EM data together with interrogation of key interactions in vivo provide structural insights into TORC1-TORC1' and TORC1-EGOC interaction interfaces. These data support a model in which glucose-dependent activation of EGOC triggers binding to TORC1 at an interface required for TOROID assembly, preventing TORC1 polymerization and promoting release of active TORC1. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13594.map.gz emd_13594.map.gz | 91.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13594-v30.xml emd-13594-v30.xml emd-13594.xml emd-13594.xml | 28 KB 28 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13594_fsc.xml emd_13594_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13594.jpg emd_13594.jpg emd_13594.png emd_13594.png | 2.9 MB 157.9 KB | ||
| Filedesc metadata |  emd-13594.cif.gz emd-13594.cif.gz | 10.1 KB | ||
| その他 |  emd_13594_half_map_1.map.gz emd_13594_half_map_1.map.gz emd_13594_half_map_2.map.gz emd_13594_half_map_2.map.gz | 95.4 MB 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13594 http://ftp.pdbj.org/pub/emdb/structures/EMD-13594 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13594 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13594 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13594_validation.pdf.gz emd_13594_validation.pdf.gz | 945.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13594_full_validation.pdf.gz emd_13594_full_validation.pdf.gz | 944.9 KB | 表示 | |
| XML形式データ |  emd_13594_validation.xml.gz emd_13594_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_13594_validation.cif.gz emd_13594_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13594 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13594 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13594 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13594 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7pqhMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13594.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13594.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main EM map of TORC1 in TOROID (TORC1 Organized in Inhibited Domains), obtained by Single Particle Analysis (SPA) after signal subtraction performed on TOROID filaments. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: EM half map 1 of TORC1 in TOROID...
| ファイル | emd_13594_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 1 of TORC1 in TOROID (TORC1 Organized in Inhibited Domains), obtained by Single Particle Analysis (SPA) after signal subtraction performed on TOROID filaments. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EM half map 2 of TORC1 in TOROID...
| ファイル | emd_13594_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 2 of TORC1 in TOROID (TORC1 Organized in Inhibited Domains), obtained by Single Particle Analysis (SPA) after signal subtraction performed on TOROID filaments. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TORC1 Organized in Inhibited Domains (TOROID)
| 全体 | 名称: TORC1 Organized in Inhibited Domains (TOROID) |
|---|---|
| 要素 |
|
-超分子 #1: TORC1 Organized in Inhibited Domains (TOROID)
| 超分子 | 名称: TORC1 Organized in Inhibited Domains (TOROID) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Target of rapamycin complex 1 subunit KOG1,Target of rapamycin co...
| 分子 | 名称: Target of rapamycin complex 1 subunit KOG1,Target of rapamycin complex 1 subunit Kog1 タイプ: protein_or_peptide / ID: 1 詳細: S. cerevisiae TOROIDS (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873) Lst8, P41318) and Tor2 (P32600),S. cerevisiae TOROIDS (TORC1 Organized in Inhibited ...詳細: S. cerevisiae TOROIDS (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873) Lst8, P41318) and Tor2 (P32600),S. cerevisiae TOROIDS (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873) Lst8, P41318) and Tor2 (P32600) コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 183.594562 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPEIYGPQPL KPLNTVMRHG FEEQYQSDQL LQSLANDFIF YFDDKRHKTN GNPIPEEDKQ RDVNRYYQPI TDWKIMKDRQ KTVSAALLL CLNLGVDPPD VMKTHPCARV EAWVDPLNFQ DSKKAIEQIG KNLQAQYETL SLRTRYKQSL DPCVEDVKRF C NSLRRTSK ...文字列: MPEIYGPQPL KPLNTVMRHG FEEQYQSDQL LQSLANDFIF YFDDKRHKTN GNPIPEEDKQ RDVNRYYQPI TDWKIMKDRQ KTVSAALLL CLNLGVDPPD VMKTHPCARV EAWVDPLNFQ DSKKAIEQIG KNLQAQYETL SLRTRYKQSL DPCVEDVKRF C NSLRRTSK EDRILFHYNG HGVPKPTKSG EIWVFNRGYT QYIPVSLYDL QTWLGAPCIF VYDCNSAENI LINFQKFVQK RI KDDEEGN HDVAAPSPTS AYQDCFQLAS CTSDELLLMS PELPADLFSC CLTCPIEISI RIFLMQSPLK DSKYKIFFEN STS NQPFGD SKNSFKSKIP NVNIPGMLSD RRTPLGELNW IFTAITDTIA WTSLPRPLFK KLFRHDLMIA ALFRNFLLAK RIMP WYNCH PVSDPELPDS ITTHPMWKSW DLAMDEVLTK IVIDLKNAPP ATALESQMIL QQQETLQNGG SSKSNAQDTK AGSIQ TQSR FAVANLSTMS LVNNPALQSR KSISLQSSQQ QLQQQQQQQQ QFTGFFEQNL TAFELWLKYA SNVRHPPEQL PIVLQV LLS QVHRIRALVL LSRFLDLGPW AVYLSLSIGI FPYVLKLLQS PAPELKPILV FIWARIMSID YKNTQSELIK EKGYMYF VT VLVPDWGVNG MSATNGSAMI NSGNPLTMTA SQNINGPSSR YYERQQGNRT SNLGHNNLPF YHSNDTTDEQ KAMAVFVL A SFVRNFPLGQ KNCFSLELVN KLCFYIDNSE IPLLRQWCVI LLGLLFADNP LNRFVCMNTG AVEILLKSLK DPVPEVRTA SIFALKHFIS GFQDAEVILR LQQEFEEQYQ QLHSQLQHLQ NQSHLQQQQS QQQQQHLEQQ QMKIEKQIRH CQVMQNQLEV IDLRKLKRQ EIGNLISILP LINDGSSLVR KELVVYFSHI VSRYSNFFIV VVFNDLLEEI KLLEKSDINT RNTSDKYSVS Q GSIFYTVW KSLLILAEDP FLENKELSKQ VIDYILLELS AHKELGGPFA VMEKFLLKRS SKAHQTGKFG FNSSQVQFVK SS LRSFSPN ERVDNNAFKK EQQQHDPKIS HPMRTSLAKL FQSLGFSESN SDSDTQSSNT SMKSHTSKKG PSGLYLLNGN NNI YPTAET PRFRKHTEPL QLPLNSSFLD YSREYFQEPQ MKKQEADEPG SVEYNARLWR RNRNETIIQE TQGEKKLSIY GNWS KKLIS LNNKSQPKLM KFAQFEDQLI TADDRSTITV FDWEKGKTLS KFSNGTPFGT KVTDLKLINE DDSALLLTGS SDGVI KIYR DYQDVDTFKI VSAWRGLTDM LLTPRSTGLL TEWLQIRGSL LTTGDVKVIR VWDAHTETVE VDIPAKTSSL ITSLTA DQL AGNIFVAGFA DGSLRVYDRR LDPRDSMIRR WRAGNDKQGV WINNVHLQRG GYRELVSGAT NGVVELWDIR SEDPVES FV DQNVTSQYGS QQKPTTMTCM QVHEHAPIIA TGTKQIKIWT TSGDLLNSFK NSHNNGVTST LAATGIPKSL SYSSTSDA F LSSMAFHPHR MMIAATNSHD SIVNIYKCED ERIDYFRTLQ VDKRRWKKNF IAVSAANRFK KISSSGALDY DIPTTASVD GSENLYFQ UniProtKB: Target of rapamycin complex 1 subunit KOG1 |
-分子 #2: Target of rapamycin complex subunit LST8
| 分子 | 名称: Target of rapamycin complex subunit LST8 / タイプ: protein_or_peptide / ID: 2 詳細: S. cerevisiae TOROIDs (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873), Lst8 (P41318) and Tor2 (P32600) コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.077879 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVILVSAGY DHTIRFWEAL TGVCSRTIQH SDSQVNRLEI TNDKKLLATA GHQNVRLYDI RTTNPNPVAS FEGHRGNVTS VSFQQDNRW MVTSSEDGTI KVWDVRSPSI PRNYKHNAPV NEVVIHPNQG ELISCDRDGN IRIWDLGENQ CTHQLTPEDD T SLQSLSMA ...文字列: MSVILVSAGY DHTIRFWEAL TGVCSRTIQH SDSQVNRLEI TNDKKLLATA GHQNVRLYDI RTTNPNPVAS FEGHRGNVTS VSFQQDNRW MVTSSEDGTI KVWDVRSPSI PRNYKHNAPV NEVVIHPNQG ELISCDRDGN IRIWDLGENQ CTHQLTPEDD T SLQSLSMA SDGSMLAAAN TKGNCYVWEM PNHTDASHLK PVTKFRAHST YITRILLSSD VKHLATCSAD HTARVWSIDD DF KLETTLD GHQRWVWDCA FSADSAYLVT ASSDHYVRLW DLSTREIVRQ YGGHHKGAVC VALNDV UniProtKB: Target of rapamycin complex subunit LST8 |
-分子 #3: Serine/threonine-protein kinase TOR2
| 分子 | 名称: Serine/threonine-protein kinase TOR2 / タイプ: protein_or_peptide / ID: 3 詳細: S. cerevisiae TOROIDs (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873), Lst8 (P41318) and Tor2 (P32600) コピー数: 4 / 光学異性体: LEVO / EC番号: 1-phosphatidylinositol 4-kinase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 281.915438 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNKYINKYTT PPNLLSLRQR AEGKHRTRKK LTHKSHSHDD EMSTTSNTDS NHNGPNDSGR VITGSAGHIG KISFVDSELD TTFSTLNLI FDKLKSDVPQ ERASGANELS TTLTSLAREV SAEQFQRFSN SLNNKIFELI HGFTSSEKIG GILAVDTLIS F YLSTEELP ...文字列: MNKYINKYTT PPNLLSLRQR AEGKHRTRKK LTHKSHSHDD EMSTTSNTDS NHNGPNDSGR VITGSAGHIG KISFVDSELD TTFSTLNLI FDKLKSDVPQ ERASGANELS TTLTSLAREV SAEQFQRFSN SLNNKIFELI HGFTSSEKIG GILAVDTLIS F YLSTEELP NQTSRLANYL RVLIPSSDIE VMRLAANTLG RLTVPGGTLT SDFVEFEVRT CIDWLTLTAD NNSSSSKLEY RR HAALLII KALADNSPYL LYPYVNSILD NIWVPLRDAK LIIRLDAAVA LGKCLTIIQD RDPALGKQWF QRLFQGCTHG LSL NTNDSV HATLLVFREL LSLKAPYLRD KYDDIYKSTM KYKEYKFDVI RREVYAILPL LAAFDPAIFT KKYLDRIMVH YLRY LKNID MNAANNSDKP FILVSIGDIA FEVGSSISPY MTLILDNIRE GLRTKFKVRK QFEKDLFYCI GKLACALGPA FAKHL NKDL LNLMLNCPMS DHMQETLMIL NEKIPSLEST VNSRILNLLS ISLSGEKFIQ SNQYDFNNQF SIEKARKSRN QSFMKK TGE SNDDITDAQI LIQCFKMLQL IHHQYSLTEF VRLITISYIE HEDSSVRKLA ALTSCDLFIK DDICKQTSVH ALHSVSE VL SKLLMIAITD PVAEIRLEIL QHLGSNFDPQ LAQPDNLRLL FMALNDEIFG IQLEAIKIIG RLSSVNPAYV VPSLRKTL L ELLTQLKFSN MPKKKEESAT LLCTLINSSD EVAKPYIDPI LDVILPKCQD ASSAVASTAL KVLGELSVVG GKEMTRYLK ELMPLIINTF QDQSNSFKRD AALTTLGQLA ASSGYVVGPL LDYPELLGIL INILKTENNP HIRRGTVRLI GILGALDPYK HREIEVTSN SKSSVEQNAP SIDIALLMQG VSPSNDEYYP TVVIHNLMKI LNDPSLSIHH TAAIQAIMHI FQNLGLRCVS F LDQIIPGI ILVMRSCPPS QLDFYFQQLG SLISIVKQHI RPHVEKIYGV IREFFPIIKL QITIISVIES ISKALEGEFK RF VPETLTF FLDILENDQS NKRIVPIRIL KSLVTFGPNL EDYSHLIMPI VVRMTEYSAG SLKKISIITL GRLAKNINLS EMS SRIVQA LVRILNNGDR ELTKATMNTL SLLLLQLGTD FVVFVPVINK ALLRNRIQHS VYDQLVNKLL NNECLPTNII FDKE NEVPE RKNYEDEMQV TKLPVNQNIL KNAWYCSQQK TKEDWQEWIR RLSIQLLKES PSACLRSCSS LVSVYYPLAR ELFNA SFSS CWVELQTSYQ EDLIQALCKA LSSSENPPEI YQMLLNLVEF MEHDDKPLPI PIHTLGKYAQ KCHAFAKALH YKEVEF LEE PKNSTIEALI SINNQLHQTD SAIGILKHAQ QHNELQLKET WYEKLQRWED ALAAYNEKEA AGEDSVEVMM GKLRSLY AL GEWEELSKLA SEKWGTAKPE VKKAMAPLAA GAAWGLEQWD EIAQYTSVMK SQSPDKEFYD AILCLHRNNF KKAEVHIF N ARDLLVTELS ALVNESYNRA YNVVVRAQII AELEEIIKYK KLPQNSDKRL TMRETWNTRL LGCQKNIDVW QRILRVRSL VIKPKEDAQV RIKFANLCRK SGRMALAKKV LNTLLEETDD PDHPNTAKAS PPVVYAQLKY LWATGLQDEA LKQLINFTSR MAHDLGLDP NNMIAQSVPQ QSKRVPRHVE DYTKLLARCF LKQGEWRVCL QPKWRLSNPD SILGSYLLAT HFDNTWYKAW H NWALANFE VISMLTSVSK KKQEGSDASS VTDINEFDNG MIGVNTFDAK EVHYSSNLIH RHVIPAIKGF FHSISLSESS SL QDALRLL TLWFTFGGIP EATQAMHEGF NLIQIGTWLE VLPQLISRIH QPNQIVSRSL LSLLSDLGKA HPQALVYPLM VAI KSESLS RQKAALSIIE KMRIHSPVLV DQAELVSHEL IRMAVLWHEQ WYEGLDDASR QFFGEHNTEK MFAALEPLYE MLKR GPETL REISFQNSFG RDLNDAYEWL MNYKKSKDVS NLNQAWDIYY NVFRKIGKQL PQLQTLELQH VSPKLLSAHD LELAV PGTR ASGGKPIVKI SKFEPVFSVI SSKQRPRKFC IKGSDGKDYK YVLKGHEDIR QDSLVMQLFG LVNTLLQNDA ECFRRH LDI QQYPAIPLSP KSGLLGWVPN SDTFHVLIRE HREAKKIPLN IEHWVMLQMA PDYDNLTLLQ KVEVFTYALN NTEGQDL YK VLWLKSRSSE TWLERRTTYT RSLAVMSMTG YILGLGDRHP SNLMLDRITG KVIHIDFGDC FEAAILREKF PEKVPFRL T RMLTYAMEVS GIEGSFRITC ENVMKVLRDN KGSLMAILEA FAFDPLINWG FDLPTKKIEE ETGIQLPVMN ANELLSNGA ITEEEVQRVE NEHKNAIRNA RAMLVLKRIT DKLTGNDIRR FNDLDVPEQV DKLIQQATSV ENLCQHYIGW CPFW UniProtKB: Serine/threonine-protein kinase TOR2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||||||||
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 2-16 / 実像数: 4901 / 平均露光時間: 8.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 37000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial homology models of Tor2 and Lst8 were generated using Phyre2, while a model of Kog1 was generated using ITasser. Homology models of Tor2, Lst8 and Kog1 were first manually placed in the final 3D map followed by rigid-body fitting in Chimera. The rigid-body fitted models were subsequently subjected to a round of flexible fitting using Imodfit followed by automatic molecular dynamics flexible fitting using NAMDINATOR. The flexibly fitted structure was then refined using the Phenix software package. |
|---|---|
| 精密化 | プロトコル: OTHER |
| 得られたモデル |  PDB-7pqh: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)