+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11004 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Mycobacterium smegmatis HelD protein in complex with RNA polymerase core - State II, primary channel engaged and active site interfering | |||||||||
 マップデータ マップデータ | Structure of Mycobacterium smegmatis HelD protein in complex with RNA polymerase core (State 2) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transcription cycle helicase-like protein RNA polymerase / trasncription / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA helicase complex / recombinational repair / 3'-5' DNA helicase activity / DNA-directed RNA polymerase complex / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / protein dimerization activity / hydrolase activity / response to antibiotic ...DNA helicase complex / recombinational repair / 3'-5' DNA helicase activity / DNA-directed RNA polymerase complex / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / protein dimerization activity / hydrolase activity / response to antibiotic / DNA-templated transcription / magnesium ion binding / DNA binding / zinc ion binding / ATP binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||
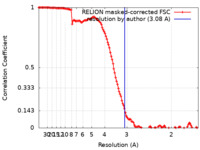
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||
 データ登録者 データ登録者 | Kouba T / Koval T | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Mycobacterial HelD is a nucleic acids-clearing factor for RNA polymerase. 著者: Tomáš Kouba / Tomáš Koval' / Petra Sudzinová / Jiří Pospíšil / Barbora Brezovská / Jarmila Hnilicová / Hana Šanderová / Martina Janoušková / Michaela Šiková / Petr Halada / ...著者: Tomáš Kouba / Tomáš Koval' / Petra Sudzinová / Jiří Pospíšil / Barbora Brezovská / Jarmila Hnilicová / Hana Šanderová / Martina Janoušková / Michaela Šiková / Petr Halada / Michal Sýkora / Ivan Barvík / Jiří Nováček / Mária Trundová / Jarmila Dušková / Tereza Skálová / URee Chon / Katsuhiko S Murakami / Jan Dohnálek / Libor Krásný /    要旨: RNA synthesis is central to life, and RNA polymerase (RNAP) depends on accessory factors for recovery from stalled states and adaptation to environmental changes. Here, we investigated the mechanism ...RNA synthesis is central to life, and RNA polymerase (RNAP) depends on accessory factors for recovery from stalled states and adaptation to environmental changes. Here, we investigated the mechanism by which a helicase-like factor HelD recycles RNAP. We report a cryo-EM structure of a complex between the Mycobacterium smegmatis RNAP and HelD. The crescent-shaped HelD simultaneously penetrates deep into two RNAP channels that are responsible for nucleic acids binding and substrate delivery to the active site, thereby locking RNAP in an inactive state. We show that HelD prevents non-specific interactions between RNAP and DNA and dissociates stalled transcription elongation complexes. The liberated RNAP can either stay dormant, sequestered by HelD, or upon HelD release, restart transcription. Our results provide insights into the architecture and regulation of the highly medically-relevant mycobacterial transcription machinery and define HelD as a clearing factor that releases RNAP from nonfunctional complexes with nucleic acids. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11004.map.gz emd_11004.map.gz | 84.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11004-v30.xml emd-11004-v30.xml emd-11004.xml emd-11004.xml | 27.1 KB 27.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11004_fsc.xml emd_11004_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11004.png emd_11004.png | 111 KB | ||
| Filedesc metadata |  emd-11004.cif.gz emd-11004.cif.gz | 8.3 KB | ||
| その他 |  emd_11004_additional_1.map.gz emd_11004_additional_1.map.gz emd_11004_half_map_1.map.gz emd_11004_half_map_1.map.gz emd_11004_half_map_2.map.gz emd_11004_half_map_2.map.gz | 166.7 MB 140.9 MB 140.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11004 http://ftp.pdbj.org/pub/emdb/structures/EMD-11004 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11004 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11004 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11004_validation.pdf.gz emd_11004_validation.pdf.gz | 881.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11004_full_validation.pdf.gz emd_11004_full_validation.pdf.gz | 880.9 KB | 表示 | |
| XML形式データ |  emd_11004_validation.xml.gz emd_11004_validation.xml.gz | 19.8 KB | 表示 | |
| CIF形式データ |  emd_11004_validation.cif.gz emd_11004_validation.cif.gz | 25.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11004 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11004 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11004 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11004 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11004.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11004.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Mycobacterium smegmatis HelD protein in complex with RNA polymerase core (State 2) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8311 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
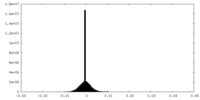
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Structure of Mycobacterium smegmatis HelD protein in complex...
| ファイル | emd_11004_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Mycobacterium smegmatis HelD protein in complex with RNA polymerase core (State 2) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
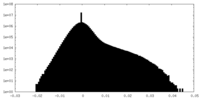
| 密度ヒストグラム |
-ハーフマップ: Structure of Mycobacterium smegmatis HelD protein in complex...
| ファイル | emd_11004_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Mycobacterium smegmatis HelD protein in complex with RNA polymerase core halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Structure of Mycobacterium smegmatis HelD protein in complex...
| ファイル | emd_11004_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Mycobacterium smegmatis HelD protein in complex with RNA polymerase core (State 2) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterium smegmatis HelD protein in complex with RNA polymera...
| 全体 | 名称: Mycobacterium smegmatis HelD protein in complex with RNA polymerase core (State 2) |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacterium smegmatis HelD protein in complex with RNA polymera...
| 超分子 | 名称: Mycobacterium smegmatis HelD protein in complex with RNA polymerase core (State 2) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 444 KDa |
-分子 #1: DNA-directed RNA polymerase subunit alpha
| 分子 | 名称: DNA-directed RNA polymerase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 37.959441 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLISQRPTLS EETVAENRSR FVIEPLEPGF GYTLGNSLRR TLLSSIPGAA VTSIRIDGVL HEFTTVPGVK EDVTDIILNL KGLVVSSDD DEPVTMYLRK QGPGVVTAGD IVPPAGVTVH NPDMHIATLN DKGKLEVELV VERGRGYVPA VQNKASGAEI G RIPVDSIY ...文字列: MLISQRPTLS EETVAENRSR FVIEPLEPGF GYTLGNSLRR TLLSSIPGAA VTSIRIDGVL HEFTTVPGVK EDVTDIILNL KGLVVSSDD DEPVTMYLRK QGPGVVTAGD IVPPAGVTVH NPDMHIATLN DKGKLEVELV VERGRGYVPA VQNKASGAEI G RIPVDSIY SPVLKVTYKV EATRVEQRTD FDKLIIDVET KNSISPRDAL ASAGGTLVEL FGLARELNAD SEHIEIGPSP AE ADHIASF ALPIDDLDLT VRSYNCLKRE GVHTVGELVA RTESDLLDIR NFGQKSIDEV KIKLHQLGLS LKDSPATFDP SEV AGYDAA TGTWTSDAGY DLDDNQDYAE TEQL UniProtKB: DNA-directed RNA polymerase subunit alpha |
-分子 #2: DNA-directed RNA polymerase subunit beta
| 分子 | 名称: DNA-directed RNA polymerase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 128.680141 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLEGCILAVS SQSKSNAITN NSVPGAPNRV SFAKLREPLE VPGLLDVQTD SFEWLVGSDR WRQAAIDRGE ENPVGGLEEV LAELSPIED FSGSMSLSFS DPRFDEVKAS VDECKDKDMT YAAPLFVTAE FINNNTGEIK SQTVFMGDFP MMTEKGTFII N GTERVVVS ...文字列: MLEGCILAVS SQSKSNAITN NSVPGAPNRV SFAKLREPLE VPGLLDVQTD SFEWLVGSDR WRQAAIDRGE ENPVGGLEEV LAELSPIED FSGSMSLSFS DPRFDEVKAS VDECKDKDMT YAAPLFVTAE FINNNTGEIK SQTVFMGDFP MMTEKGTFII N GTERVVVS QLVRSPGVYF DETIDKSTEK TLHSVKVIPG RGAWLEFDVD KRDTVGVRID RKRRQPVTVL LKALGWTNEQ IV ERFGFSE IMMGTLEKDT TSGTDEALLD IYRKLRPGEP PTKESAQTLL ENLFFKEKRY DLARVGRYKV NKKLGLNAGK PIT SSTLTE EDVVATIEYL VRLHEGQTSM TVPGGVEVPV EVDDIDHFGN RRLRTVGELI QNQIRVGLSR MERVVRERMT TQDV EAITP QTLINIRPVV AAIKEFFGTS QLSQFMDQNN PLSGLTHKRR LSALGPGGLS RERAGLEVRD VHPSHYGRMC PIETP EGPN IGLIGSLSVY ARVNPFGFIE TPYRKVENGV VTDQIDYLTA DEEDRHVVAQ ANSPTDENGR FTEDRVMVRK KGGEVE FVS ADQVDYMDVS PRQMVSVATA MIPFLEHDDA NRALMGANMQ RQAVPLVRSE APLVGTGMEL RAAIDAGDVV VADKTGV IE EVSADYITVM ADDGTRQSYR LRKFARSNHG TCANQRPIVD AGQRVEAGQV IADGPCTQNG EMALGKNLLV AIMPWEGH N YEDAIILSNR LVEEDVLTSI HIEEHEIDAR DTKLGAEEIT RDIPNVSDEV LADLDERGIV RIGAEVRDGD ILVGKVTPK GETELTPEER LLRAIFGEKA REVRDTSLKV PHGESGKVIG IRVFSREDDD ELPAGVNELV RVYVAQKRKI SDGDKLAGRH GNKGVIGKI LPVEDMPFLP DGTPVDIILN THGVPRRMNI GQILETHLGW VAKAGWNIDV AAGVPDWASK LPEELYSAPA D STVATPVF DGAQEGELAG LLGSTLPNRD GEVMVDADGK STLFDGRSGE PFPYPVTVGY MYILKLHHLV DDKIHARSTG PY SMITQQP LGGKAQFGGQ RFGEMECWAM QAYGAAYTLQ ELLTIKSDDT VGRVKVYEAI VKGENIPEPG IPESFKVLLK ELQ SLCLNV EVLSSDGAAI EMRDGDDEDL ERAAANLGIN LSRNESASVE DLA UniProtKB: DNA-directed RNA polymerase subunit beta |
-分子 #3: DNA-directed RNA polymerase subunit beta'
| 分子 | 名称: DNA-directed RNA polymerase subunit beta' / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 146.712891 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLDVNFFDEL RIGLATADDI RNWSYGEVKK PETINYRTLK PEKDGLFCEK IFGPTRDWEC YCGKYKRVRF KGIICERCGV EVTRAKVRR ERMGHIELAA PVTHIWYFKG VPSRLGYLLD LAPKDLEKII YFAAYVITSV DDEMRHNELS TLEAEMAVEK K AVEDQRDA ...文字列: MLDVNFFDEL RIGLATADDI RNWSYGEVKK PETINYRTLK PEKDGLFCEK IFGPTRDWEC YCGKYKRVRF KGIICERCGV EVTRAKVRR ERMGHIELAA PVTHIWYFKG VPSRLGYLLD LAPKDLEKII YFAAYVITSV DDEMRHNELS TLEAEMAVEK K AVEDQRDA DLEARAQKLE ADLAELEAEG AKSDVRRKVR DSGEREMRQL RDRAQRELDR LDEIWNTFTK LAPKQLIVDE VL YRELQDR YGEYFTGAMG AESIKKLIEN FDIDAEAESL REVIRSGKGQ KKLRALKRLK VVAAFQQSGN SPMGMVLDAV PVI PPELRP MVQLDGGRFA TSDLNDLYRR VINRNNRLKR LIDLGAPEII VNNEKRMLQE SVDALFDNGR RGRPVTGPGN RPLK SLSDL LKGKQGRFRQ NLLGKRVDYS GRSVIVVGPQ LKLHQCGLPK LMALELFKPF VMKRLVDLNH AQNIKSAKRM VERQR PQVW DVLEEVIAEH PVLLNRAPTL HRLGIQAFEP QLVEGKAIQL HPLVCEAFNA DFDGDQMAVH LPLSAEAQAE ARILML SSN NILSPASGKP LAMPRLDMVT GLYYLTTLVE GATGEYQAAT KDAPEQGVYS SPAEAIMAMD RGALSVRAKI KVRLTEL RP PTDLEAQLFE NGWKPGDAWT AETTLGRVMF NELLPKSYPF VNEQMHKKVQ ARIINDLAER FPMIVVAQTV DKLKDAGF Y WATRSGVTVS MADVLVPPQK QEILERHEAE ADAIERKYQR GALNHTERNE SLVKIWQDAT EEVGKALEEF YPADNPIIT IVKSGATGNL TQTRTLAGMK GLVTNPKGEF IPRPIKSSFR EGLTVLEYFI NTHGARKGLA DTALRTADSG YLTRRLVDVS QDVIVREHD CETERGINVT LAERGPDGTL IRDAHVETSA FARTLATDAV DANGNVIIER GHDLGDPAID ALLAAGITTV K VRSVLTCT SATGVCAMCY GRSMATGKLV DIGEAVGIVA AQSIGEPGTQ LTMRTFHQGG VTGGADIVGG LPRVQELFEA RV PRNKAPI ADVAGRVRLE ESDKFFKITI VPDDGGEEVV YDKLSKRQRL RVITHEDGTE GVLSDGDHVE VGDQLMEGAA DPH EVLRVQ GPREVQIHLV KEVQEVYRAQ GVSIHDKHIE VIVRQMLRRV TIIDSGSTEF LPGSLTERAE FEAENRRVVA EGGE PAAGR PVLMGITKAS LATDSWLSAA SFQETTRVLT DAAINCRSDK LNGLKENVII GKLIPAGTGI SRYRNIQVQP TEEAR AAAY TIPSYEDQYY SPDFGQATGA AVPLDDYGYS DYR UniProtKB: DNA-directed RNA polymerase subunit beta' |
-分子 #4: DNA-directed RNA polymerase subunit omega
| 分子 | 名称: DNA-directed RNA polymerase subunit omega / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 11.544763 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSTPHADAQL NAADDLGIDS SAASAYDTPL GITNPPIDEL LSRASSKYAL VIYAAKRARQ INDYYNQLGD GILEYVGPLV EPGLQEKPL SIALREIHGD LLEHTEGE UniProtKB: DNA-directed RNA polymerase subunit omega |
-分子 #5: RNA polymerase-associated transcription factor HelD
| 分子 | 名称: RNA polymerase-associated transcription factor HelD / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 81.298078 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRDYEDEL QSERDYVAGL YARLDAERAQ SQRRYAAALR EHGGTAVERD AEVRALAKDI ARLNVADNGL CFGRLDTLDD ARLYIGRLG IFDRDNDFEP LLLDWRAPMA RPFYVATAAN PENMRRRRQF HTLGRKVVDF TDEILGRPTG AEHDATNDAA L LAAVNAPR ...文字列: MSGRDYEDEL QSERDYVAGL YARLDAERAQ SQRRYAAALR EHGGTAVERD AEVRALAKDI ARLNVADNGL CFGRLDTLDD ARLYIGRLG IFDRDNDFEP LLLDWRAPMA RPFYVATAAN PENMRRRRQF HTLGRKVVDF TDEILGRPTG AEHDATNDAA L LAAVNAPR GEGMRDIVAT IQAEQDQVIR LDHTGVLVIE GGPGTGKTVV ALHRVAYLLY TYRKQMERHG VLVVGPTPAF LD HIGRVLP SLGESDAVFM TPGDFVPGLH VTAEDTPEAA EVKGSLKILD VLKAAVADRQ ELPSEPIPID LSDVTMRIDA ETA KWARDE ARKTGLPHNE ARAEFVDVVT YVVTERAVAR IGRGWLTRDD KHAWEKMRAD VVGELEDHEQ FNAALDALWP ILTP EDVLA QLYTSHERLR AAGAPECLWR ADGEAWTVSD VPLLDELVDL LGRNKAADEA AERERREEEA YAAGVLDLMV DREDL MDDE DHLLAQDLID AEELADRFKE QDNRELSERA AADREWTYGH VVVDEAQELS EMDWRLLMRR CPRRSFTIVG DLAQRR SPA GARSWGAMLD SYVPGRWVYK SLSVNYRTPA EIMAVAAAVL AEFAPDATPP DSVRACGVAP WARQVTDDDI ASAIAEF VS EEAGREGTSV VIGPPDVPGT VPPSETKGLE FDAVLVVEPE RILADGPRGA AELYVALTRA TQRLGVLYRD ALPQALAG L AEGDAAATVE QRTSA UniProtKB: Superfamily protein I DNA or RNA helicase |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | プロトコル: AB INITIO MODEL | ||||||
| 得られたモデル |  PDB-6yys: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)