+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8154 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
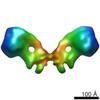
| タイトル | Masked monomer of ATP synthase dimer from Yarrowia lipolytica, subclass 2 | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) | |||||||||
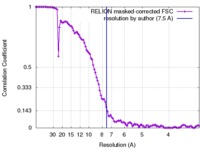
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.5 Å クライオ電子顕微鏡法 / 解像度: 7.5 Å | |||||||||
 データ登録者 データ登録者 | Hahn A / Parey K / Bublitz M / Mills DJ / Zickermann V / Vonck J / Kuehlbrandt W / Meier T | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2016 ジャーナル: Mol Cell / 年: 2016タイトル: Structure of a Complete ATP Synthase Dimer Reveals the Molecular Basis of Inner Mitochondrial Membrane Morphology. 著者: Alexander Hahn / Kristian Parey / Maike Bublitz / Deryck J Mills / Volker Zickermann / Janet Vonck / Werner Kühlbrandt / Thomas Meier /  要旨: We determined the structure of a complete, dimeric F1Fo-ATP synthase from yeast Yarrowia lipolytica mitochondria by a combination of cryo-EM and X-ray crystallography. The final structure resolves 58 ...We determined the structure of a complete, dimeric F1Fo-ATP synthase from yeast Yarrowia lipolytica mitochondria by a combination of cryo-EM and X-ray crystallography. The final structure resolves 58 of the 60 dimer subunits. Horizontal helices of subunit a in Fo wrap around the c-ring rotor, and a total of six vertical helices assigned to subunits a, b, f, i, and 8 span the membrane. Subunit 8 (A6L in human) is an evolutionary derivative of the bacterial b subunit. On the lumenal membrane surface, subunit f establishes direct contact between the two monomers. Comparison with a cryo-EM map of the F1Fo monomer identifies subunits e and g at the lateral dimer interface. They do not form dimer contacts but enable dimer formation by inducing a strong membrane curvature of ∼100°. Our structure explains the structural basis of cristae formation in mitochondria, a landmark signature of eukaryotic cell morphology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8154.map.gz emd_8154.map.gz | 94 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8154-v30.xml emd-8154-v30.xml emd-8154.xml emd-8154.xml | 12 KB 12 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8154_fsc.xml emd_8154_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8154.png emd_8154.png | 36.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8154 http://ftp.pdbj.org/pub/emdb/structures/EMD-8154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8154 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8154.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8154.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.63 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fo ATP synthase dimer
| 全体 | 名称: Fo ATP synthase dimer |
|---|---|
| 要素 |
|
-超分子 #1: Fo ATP synthase dimer
| 超分子 | 名称: Fo ATP synthase dimer / タイプ: complex / ID: 1 / 親要素: 0 詳細: The Fo section of the F1Fo dimer was masked for refinement |
|---|---|
| 由来(天然) | 生物種:   Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 1.2 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 30 mM MOPS-NaOH pH 7.5, 2 mM MgCl2, 0.5 mM EDTA, 50 mM NaCl, 0.05% (w/v) digitonin |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: blotting for 7 to 9 s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 30675 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 4.2 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 20000 Bright-field microscopy / Cs: 4.2 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 20000 |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 2-21 / 実像数: 2500 / 平均露光時間: 6.0 sec. / 平均電子線量: 18.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー