+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8196 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of NNQQNY from yeast prion Sup35 with zinc acetate determined by MicroED | |||||||||||||||||||||
 マップデータ マップデータ | NNQQNY from yeast prion Sup35 with zinc acetate | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード |  amyloid (アミロイド) / amyloid (アミロイド) /  yeast prion / PROTEIN FIBRIL yeast prion / PROTEIN FIBRIL | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Eukaryotic Translation Termination / translation release factor complex / translation release factor activity / nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / translational termination /  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / cytoplasmic stress granule / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / cytoplasmic stress granule /  翻訳 (生物学) ...Eukaryotic Translation Termination / translation release factor complex / translation release factor activity / nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / translational termination / 翻訳 (生物学) ...Eukaryotic Translation Termination / translation release factor complex / translation release factor activity / nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / translational termination /  加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / cytoplasmic stress granule / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / cytoplasmic stress granule /  翻訳 (生物学) / 翻訳 (生物学) /  mRNA binding / mRNA binding /  GTPase activity / GTP binding / identical protein binding / GTPase activity / GTP binding / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||||||||||||||
| 手法 |  電子線結晶学 / 電子線結晶学 /  クライオ電子顕微鏡法 クライオ電子顕微鏡法 | |||||||||||||||||||||
 データ登録者 データ登録者 | Rodriguez JA / Sawaya MR | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2016 ジャーナル: Proc Natl Acad Sci U S A / 年: 2016タイトル: Ab initio structure determination from prion nanocrystals at atomic resolution by MicroED. 著者: Michael R Sawaya / Jose Rodriguez / Duilio Cascio / Michael J Collazo / Dan Shi / Francis E Reyes / Johan Hattne / Tamir Gonen / David S Eisenberg /  要旨: Electrons, because of their strong interaction with matter, produce high-resolution diffraction patterns from tiny 3D crystals only a few hundred nanometers thick in a frozen-hydrated state. This ...Electrons, because of their strong interaction with matter, produce high-resolution diffraction patterns from tiny 3D crystals only a few hundred nanometers thick in a frozen-hydrated state. This discovery offers the prospect of facile structure determination of complex biological macromolecules, which cannot be coaxed to form crystals large enough for conventional crystallography or cannot easily be produced in sufficient quantities. Two potential obstacles stand in the way. The first is a phenomenon known as dynamical scattering, in which multiple scattering events scramble the recorded electron diffraction intensities so that they are no longer informative of the crystallized molecule. The second obstacle is the lack of a proven means of de novo phase determination, as is required if the molecule crystallized is insufficiently similar to one that has been previously determined. We show with four structures of the amyloid core of the Sup35 prion protein that, if the diffraction resolution is high enough, sufficiently accurate phases can be obtained by direct methods with the cryo-EM method microelectron diffraction (MicroED), just as in X-ray diffraction. The success of these four experiments dispels the concern that dynamical scattering is an obstacle to ab initio phasing by MicroED and suggests that structures of novel macromolecules can also be determined by direct methods. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8196.map.gz emd_8196.map.gz | 395.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8196-v30.xml emd-8196-v30.xml emd-8196.xml emd-8196.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8196.png emd_8196.png | 126.1 KB | ||
| Filedesc metadata |  emd-8196.cif.gz emd-8196.cif.gz | 6 KB | ||
| Filedesc structureFactors |  emd_8196_sf.cif.gz emd_8196_sf.cif.gz | 58 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8196 http://ftp.pdbj.org/pub/emdb/structures/EMD-8196 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8196 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8196 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5k2eMC  8197C  8198C 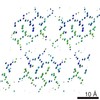 8199C  5k2fC  5k2gC  5k2hC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8196.map.gz / 形式: CCP4 / 大きさ: 1.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8196.map.gz / 形式: CCP4 / 大きさ: 1.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NNQQNY from yeast prion Sup35 with zinc acetate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.33563 Å / Y: 0.30938 Å / Z: 0.33153 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Prion fibril composed of a 6-residue segment of Sup35 and zinc
| 全体 | 名称: Prion fibril composed of a 6-residue segment of Sup35 and zinc |
|---|---|
| 要素 |
|
-超分子 #1: Prion fibril composed of a 6-residue segment of Sup35 and zinc
| 超分子 | 名称: Prion fibril composed of a 6-residue segment of Sup35 and zinc タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 3.25 kDa/nm |
-分子 #1: Eukaryotic peptide chain release factor GTP-binding subunit
| 分子 | 名称: Eukaryotic peptide chain release factor GTP-binding subunit タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 779.755 Da |
| 配列 | 文字列: NNQQNY UniProtKB: Eukaryotic peptide chain release factor GTP-binding subunit |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: ACETIC ACID
| 分子 | 名称: ACETIC ACID / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ACY |
|---|---|
| 分子量 | 理論値: 60.052 Da |
| Chemical component information |  ChemComp-ACY: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 6 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線結晶学 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 30 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 30 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV / 詳細: Plunged into liquid ethane (FEI VITROBOT MARK IV). | ||||||||||||
| 詳細 | crystal | ||||||||||||
| 結晶化 | 脂質混合液: none / 装置: 24-well plate 雰囲気: in air, in sealed chamber, in equilibrium with reservoir solution 温度: 298.0 K / 時間: 1.0 DAY 詳細: Crystals were grown by hanging-drop vapor diffusion at ~20 degrees C. The reservoir solution contained 100 mM HEPES, pH 7.0, and 1 M sodium acetate (pH not adjusted). Drops were prepared by ...詳細: Crystals were grown by hanging-drop vapor diffusion at ~20 degrees C. The reservoir solution contained 100 mM HEPES, pH 7.0, and 1 M sodium acetate (pH not adjusted). Drops were prepared by pipetting 5 microliters of 30 mg/mL aqueous peptide solution, 4 microliters of reservoir solution, and 1 microliter of 0.1 M zinc sulfate onto a glass coverslip. Crystals grew within a day. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION 回折 / カメラ長: 1350 mm 回折 / カメラ長: 1350 mm |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 100.0 K / 最高: 100.0 K |
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 2 / 回折像の数: 879 / 平均露光時間: 2.0 sec. / 平均電子線量: 0.01 e/Å2 詳細: The detector was operated in rolling shutter mode with 2x2 pixel binning. |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| Crystallography statistics | Number intensities measured: 16753 / Number structure factors: 2399 / Fourier space coverage: 82.7 / R sym: 15.1 / R merge: 15.1 / Overall phase error: 0.1 / Overall phase residual: 0.1 / Phase error rejection criteria: 0 / High resolution: 1.0 Å 詳細: Phase statistics are not applicable. No imaging was used. The phases were obtained by a crystallographic direct methods program, SHELXD. 殻:
| ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 最終 再構成 | 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES / ソフトウェア - 名称: SHELXD (ver. 2013/2) / ソフトウェア - 詳細: direct methods 詳細: The density map was obtained using measured diffraction intensities and phases acquired from crystallographic direct methods program SHELXD. | ||||||||||||||||||||||||||||||||||||||||||
| Merging software list | ソフトウェア - 名称: SCALEPACK (ver. 1.98.7) |
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER / 温度因子: 4.4 / 当てはまり具合の基準: maximum likelihood |
|---|---|
| 得られたモデル |  PDB-5k2e: |
 ムービー
ムービー コントローラー
コントローラー