+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 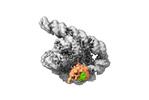 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
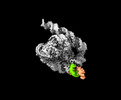
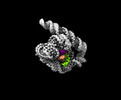
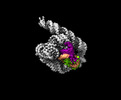
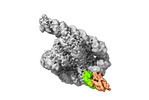
| タイトル | The intermediate pre-Tet-S1 state of G264A mutated Tetrahymena group I intron with 6nt 3'/5'-exon and 2-aminopurine nucleoside | ||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 生物種 |   Tetrahymena thermophila (真核生物) Tetrahymena thermophila (真核生物) | ||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.52 Å クライオ電子顕微鏡法 / 解像度: 3.52 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Luo B / Zhang C / Ling X / Mukherjee S / Jia G / Xie J / Jia X / Liu L / Baulin EF / Luo Y ...Luo B / Zhang C / Ling X / Mukherjee S / Jia G / Xie J / Jia X / Liu L / Baulin EF / Luo Y / Jiang L / Dong H / Wei X / Bujnicki JM / Su Z | ||||||||||||||||||||||||
| 資金援助 |  中国, 中国,  ポーランド, European Union, 7件 ポーランド, European Union, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Catal / 年: 2023 ジャーナル: Nat Catal / 年: 2023タイトル: Cryo-EM reveals dynamics of Tetrahymena group I intron self-splicing 著者: Luo B / Zhang C / Ling X / Mukherjee S / Jia G / Xie J / Jia X / Liu L / Baulin EF / Luo Y / Jiang L / Dong H / Wei X / Bujnicki JM / Su Z | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34671.map.gz emd_34671.map.gz | 28.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34671-v30.xml emd-34671-v30.xml emd-34671.xml emd-34671.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34671.png emd_34671.png | 40.4 KB | ||
| その他 |  emd_34671_half_map_1.map.gz emd_34671_half_map_1.map.gz emd_34671_half_map_2.map.gz emd_34671_half_map_2.map.gz | 33 MB 33 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34671 http://ftp.pdbj.org/pub/emdb/structures/EMD-34671 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34671 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34671 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34671.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34671.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_34671_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
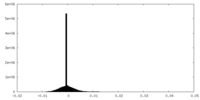
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34671_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
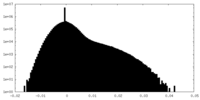
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Co-transcriptional folded G264A mutated Tetrahymena group I intro...
| 全体 | 名称: Co-transcriptional folded G264A mutated Tetrahymena group I intron with 6-nt 3'/5'-exon and 2-aminopurine nucleoside |
|---|---|
| 要素 |
|
-超分子 #1: Co-transcriptional folded G264A mutated Tetrahymena group I intro...
| 超分子 | 名称: Co-transcriptional folded G264A mutated Tetrahymena group I intron with 6-nt 3'/5'-exon and 2-aminopurine nucleoside タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Tetrahymena thermophila (真核生物) Tetrahymena thermophila (真核生物) |
-分子 #1: The intermediate pre-Tet-S1 state molecule of co-transcriptional ...
| 分子 | 名称: The intermediate pre-Tet-S1 state molecule of co-transcriptional folded G264A mutant Tetrahymena group I intron with 6nt 3'/5'-exon and 2-aminopurine nucleoside タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Tetrahymena thermophila (真核生物) Tetrahymena thermophila (真核生物) |
| 分子量 | 理論値: 135.155719 KDa |
| 配列 | 文字列: CUCUCUAAAU AGCAAUAUUU ACCUUUGGAG GGAAAAGUUA UCAGGCAUGC ACCUGGUAGC UAGUCUUUAA ACCAAUAGAU UGCAUCGGU UUAAAAGGCA AGACCGUCAA AUUGCGGGAA AGGGGUCAAC AGCCGUUCAG UACCAAGUCU CAGGGGAAAC U UUGAGAUG ...文字列: CUCUCUAAAU AGCAAUAUUU ACCUUUGGAG GGAAAAGUUA UCAGGCAUGC ACCUGGUAGC UAGUCUUUAA ACCAAUAGAU UGCAUCGGU UUAAAAGGCA AGACCGUCAA AUUGCGGGAA AGGGGUCAAC AGCCGUUCAG UACCAAGUCU CAGGGGAAAC U UUGAGAUG GCCUUGCAAA GGGUAUGGUA AUAAGCUGAC GGACAUGGUC CUAACCACGC AGCCAAGUCC UAAGUCAACA GA UCUUCUG UUGAUAUGGA UGCAGUUCAC AAACUAAAUG UCGGUCGGGG AAGAUGUAUU CUUCUCAUAA GAUAUAGUCG GAC CUCUCC UUAAUGGGAG CUAGCGGAUG AAGUGAUGCA ACACUGGAGC CGCUGGGAAC UAAUUUGUAU GCGAAAGUAU AUUG AUUAG UUUUGGAGUA CUCG |
-分子 #2: SPERMIDINE
| 分子 | 名称: SPERMIDINE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: SPD |
|---|---|
| 分子量 | 理論値: 145.246 Da |
| Chemical component information |  ChemComp-SPD: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 27 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.36 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.7 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.7 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 165000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 79.3 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-30 / 平均電子線量: 55.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 4012136 |
|---|---|
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 3次元分類 | クラス数: 6 / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 再構成 | 使用したクラス数: 1 / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.52 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 111196 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8hd7: |
 ムービー
ムービー コントローラー
コントローラー



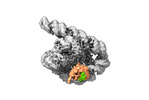










 Z
Z Y
Y X
X