+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 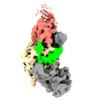 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of DNMT1 (aa:351-1616) in complex with ubiquitinated H3 | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  DNA methyltransferase (DNAメチルトランスフェラーゼ) / DNA methyltransferase (DNAメチルトランスフェラーゼ) /  TRANSFERASE (転移酵素) TRANSFERASE (転移酵素) | |||||||||||||||
| 機能・相同性 |  : :  機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
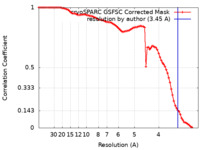
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.45 Å クライオ電子顕微鏡法 / 解像度: 3.45 Å | |||||||||||||||
 データ登録者 データ登録者 | Onoda H / Kikuchi A / Kori S / Yoshimi S / Yamagata A / Arita K | |||||||||||||||
| 資金援助 |  日本, 4件 日本, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for activation of DNMT1. 著者: Amika Kikuchi / Hiroki Onoda / Kosuke Yamaguchi / Satomi Kori / Shun Matsuzawa / Yoshie Chiba / Shota Tanimoto / Sae Yoshimi / Hiroki Sato / Atsushi Yamagata / Mikako Shirouzu / Naruhiko ...著者: Amika Kikuchi / Hiroki Onoda / Kosuke Yamaguchi / Satomi Kori / Shun Matsuzawa / Yoshie Chiba / Shota Tanimoto / Sae Yoshimi / Hiroki Sato / Atsushi Yamagata / Mikako Shirouzu / Naruhiko Adachi / Jafar Sharif / Haruhiko Koseki / Atsuya Nishiyama / Makoto Nakanishi / Pierre-Antoine Defossez / Kyohei Arita /   要旨: DNMT1 is an essential enzyme that maintains genomic DNA methylation, and its function is regulated by mechanisms that are not yet fully understood. Here, we report the cryo-EM structure of human ...DNMT1 is an essential enzyme that maintains genomic DNA methylation, and its function is regulated by mechanisms that are not yet fully understood. Here, we report the cryo-EM structure of human DNMT1 bound to its two natural activators: hemimethylated DNA and ubiquitinated histone H3. We find that a hitherto unstudied linker, between the RFTS and CXXC domains, plays a key role for activation. It contains a conserved α-helix which engages a crucial "Toggle" pocket, displacing a previously described inhibitory linker, and allowing the DNA Recognition Helix to spring into the active conformation. This is accompanied by large-scale reorganization of the inhibitory RFTS and CXXC domains, allowing the enzyme to gain full activity. Our results therefore provide a mechanistic basis for the activation of DNMT1, with consequences for basic research and drug design. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33299.map.gz emd_33299.map.gz | 32.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33299-v30.xml emd-33299-v30.xml emd-33299.xml emd-33299.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33299_fsc.xml emd_33299_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33299.png emd_33299.png | 69.6 KB | ||
| マスクデータ |  emd_33299_msk_1.map emd_33299_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-33299.cif.gz emd-33299.cif.gz | 6.6 KB | ||
| その他 |  emd_33299_half_map_1.map.gz emd_33299_half_map_1.map.gz emd_33299_half_map_2.map.gz emd_33299_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33299 http://ftp.pdbj.org/pub/emdb/structures/EMD-33299 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33299 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33299 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33299.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33299.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.477 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33299_msk_1.map emd_33299_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
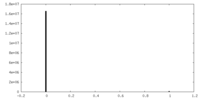
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33299_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
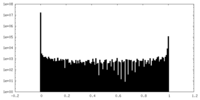
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33299_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
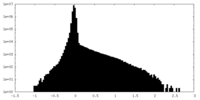
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human DNMT1 351_1616
| 全体 | 名称: human DNMT1 351_1616 |
|---|---|
| 要素 |
|
-超分子 #1: human DNMT1 351_1616
| 超分子 | 名称: human DNMT1 351_1616 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.4 MDa |
-分子 #1: DNA (cytosine-5)-methyltransferase 1
| 分子 | 名称: DNA (cytosine-5)-methyltransferase 1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号:  DNAメチルトランスフェラーゼ DNAメチルトランスフェラーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 株: Isoform 1 Homo sapiens (ヒト) / 株: Isoform 1 |
| 組換発現 | 生物種:   Baculovirus expression vector pFastBac1-HM (バキュロウイルス科) Baculovirus expression vector pFastBac1-HM (バキュロウイルス科) |
| 配列 | 文字列: PKCIQCGQYL DDPDLKYGQH PPDAVDEPQ MLTNEKLSIF D ANESGFES YEALPQHKLT CF SVYCKHG HLCPIDTGLI EKN IELFFS GSAKPIYDDD PSLE GGVNG KNLGPINEWW ITGFD GGEK ALIGFSTSFA EYILMD PSP EYAPIFGLMQ EKIYISK IV ...文字列: PKCIQCGQYL DDPDLKYGQH PPDAVDEPQ MLTNEKLSIF D ANESGFES YEALPQHKLT CF SVYCKHG HLCPIDTGLI EKN IELFFS GSAKPIYDDD PSLE GGVNG KNLGPINEWW ITGFD GGEK ALIGFSTSFA EYILMD PSP EYAPIFGLMQ EKIYISK IV VEFLQSNSDS TYEDLINK I ETTVPPSGLN LNRFTEDSL LRHAQFVVEQ VESYDEAGDS DEQPIFLTP CMRDLIKLAG V TLGQRRAQ ARRQTIRHST RE KDRGPTK ATTTKLVYQI FDT FFAEQI EKDDREDKEN AFKR RRCGV CEVCQQPECG KCKAC KDMV KFGGSGRSKQ ACQERR CPN MAMKEADDDE EVDDNIP EM PSPKKMHQGK KKKQNKNR I SWVGEAVKTD GKKSYYKKV CIDAETLEVG DCVSVIPDDS SKPLYLARV TALWEDSSNG Q MFHAHWFC AGTDTVLGAT SD PLELFLV DECEDMQLSY IHS KVKVIY KAPSENWAME GGMD PESLL EGDDGKTYFY QLWYD QDYA RFESPPKTQP TEDNKF KFC VSCARLAEMR QKEIPRV LE QLEDLDSRVL YYSATKNG I LYRVGDGVYL PPEAFTFNI KLSSPVKRPR KEPVDEDLYP EHYRKYSDY IKGSNLDAPE P YRIGRIKE IFCPKKSNGR PN ETDIKIR VNKFYRPENT HKS TPASYH ADINLLYWSD EEAV VDFKA VQGRCTVEYG EDLPE CVQV YSMGGPNRFY FLEAYN AKS KSFEDPPNHA RSPGNKG KG KGKGKGKPKS QACEPSEP E IEIKLPKLRT LDVFSGCGG LSEGFHQAGI SDTLWAIEMW DPAAQAFRL NNPGSTVFTE D CNILLKLV MAGETTNSRG QR LPQKGDV EMLCGGPPCQ GFS GMNRFN SRTYSKFKNS LVVS FLSYC DYYRPRFFLL ENVRN FVSF KRSMVLKLTL RCLVRM GYQ CTFGVLQAGQ YGVAQTR RR AIILAAAPGE KLPLFPEP L HVFAPRACQL SVVVDDKKF VSNITRLSSG PFRTITVRDT MSDLPEVRN GASALEISYN G EPQSWFQR QLRGAQYQPI LR DHICKDM SALVAARMRH IPL APGSDW RDLPNIEVRL SDGT MARKL RYTHHDRKNG RSSSG ALRG VCSCVEAGKA CDPAAR QFN TLIPWCLPHT GNRHNHW AG LYGRLEWDGF FSTTVTNP E PMGKQGRVLH PEQHRVVSV RECARSQGFP DTYRLFGNIL DKHRQVGNA VPPPLAKAIG L EIKLCMLA KARESASAKI KE EEAAKD UniProtKB:  UNIPROTKB: NP_001370.1 UNIPROTKB: NP_001370.1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 4 seconds before plunging. | |||||||||
| 詳細 | This sample was monodisperse by Size-exclusion chromatography |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 1.4000000000000001 µm 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 1 / 実像数: 2071 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: B / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X