+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3185 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the in vitro assembled bacteriophage phi6 polymerase complex | |||||||||
 マップデータ マップデータ | Icosahedral reconstruction of in vitro assembled bacteriophage phi6 polymerase complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacteriophage phi6 /  polymerase complex (ポリメラーゼ) polymerase complex (ポリメラーゼ) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=2 icosahedral viral capsid /  RNA uridylyltransferase activity / viral procapsid / viral genome packaging / viral inner capsid / ribonucleoside triphosphate phosphatase activity / RNA uridylyltransferase activity / viral procapsid / viral genome packaging / viral inner capsid / ribonucleoside triphosphate phosphatase activity /  virion component / virion component /  カプシド / nucleoside-triphosphate phosphatase / viral nucleocapsid ...T=2 icosahedral viral capsid / カプシド / nucleoside-triphosphate phosphatase / viral nucleocapsid ...T=2 icosahedral viral capsid /  RNA uridylyltransferase activity / viral procapsid / viral genome packaging / viral inner capsid / ribonucleoside triphosphate phosphatase activity / RNA uridylyltransferase activity / viral procapsid / viral genome packaging / viral inner capsid / ribonucleoside triphosphate phosphatase activity /  virion component / virion component /  カプシド / nucleoside-triphosphate phosphatase / viral nucleocapsid / カプシド / nucleoside-triphosphate phosphatase / viral nucleocapsid /  RNA依存性RNAポリメラーゼ / viral RNA genome replication / RNA依存性RNAポリメラーゼ / viral RNA genome replication /  RNA-dependent RNA polymerase activity / RNA-dependent RNA polymerase activity /  nucleotide binding / DNA-templated transcription / nucleotide binding / DNA-templated transcription /  RNA binding / RNA binding /  ATP binding / identical protein binding / ATP binding / identical protein binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) | |||||||||
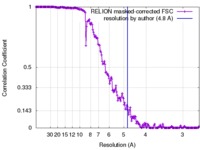
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.8 Å クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Ilca S / Kotecha A / Sun X / Poranen MP / Stuart DI / Huiskonen JT | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2015 ジャーナル: Nat Commun / 年: 2015タイトル: Localized reconstruction of subunits from electron cryomicroscopy images of macromolecular complexes. 著者: Serban L Ilca / Abhay Kotecha / Xiaoyu Sun / Minna M Poranen / David I Stuart / Juha T Huiskonen /   要旨: Electron cryomicroscopy can yield near-atomic resolution structures of highly ordered macromolecular complexes. Often however some subunits bind in a flexible manner, have different symmetry from the ...Electron cryomicroscopy can yield near-atomic resolution structures of highly ordered macromolecular complexes. Often however some subunits bind in a flexible manner, have different symmetry from the rest of the complex, or are present in sub-stoichiometric amounts, limiting the attainable resolution. Here we report a general method for the localized three-dimensional reconstruction of such subunits. After determining the particle orientations, local areas corresponding to the subunits can be extracted and treated as single particles. We demonstrate the method using three examples including a flexible assembly and complexes harbouring subunits with either partial occupancy or mismatched symmetry. Most notably, the method allows accurate fitting of the monomeric RNA-dependent RNA polymerase bound at the threefold axis of symmetry inside a viral capsid, revealing for the first time its exact orientation and interactions with the capsid proteins. Localized reconstruction is expected to provide novel biological insights in a range of challenging biological systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3185.map.gz emd_3185.map.gz | 385.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3185-v30.xml emd-3185-v30.xml emd-3185.xml emd-3185.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3185_fsc.xml emd_3185_fsc.xml | 16.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  EMD-3185.tiff EMD-3185.tiff emd_3185.tiff emd_3185.tiff | 1.1 MB 1.1 MB | ||
| マスクデータ |  emd_3185_msk_1.map emd_3185_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3185 http://ftp.pdbj.org/pub/emdb/structures/EMD-3185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3185 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3185.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3185.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Icosahedral reconstruction of in vitro assembled bacteriophage phi6 polymerase complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: Mask used for FSC
| 注釈 | Mask used for FSC | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_3185_msk_1.map emd_3185_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage phi6 polymerase complex assembled in vitro from pur...
| 全体 | 名称: Bacteriophage phi6 polymerase complex assembled in vitro from purified proteins P1, P2, and P4 |
|---|---|
| 要素 |
|
-超分子 #1000: Bacteriophage phi6 polymerase complex assembled in vitro from pur...
| 超分子 | 名称: Bacteriophage phi6 polymerase complex assembled in vitro from purified proteins P1, P2, and P4 タイプ: sample / ID: 1000 / 集合状態: Icosahedral assembly with 120 copies of P1 / Number unique components: 3 |
|---|
-分子 #1: P1 protein from bacteriophage phi6
| 分子 | 名称: P1 protein from bacteriophage phi6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 120 集合状態: 60 asymmetric dimers from an icosahedral shell 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 分子量 | 理論値: 85 KDa |
| 組換発現 | 生物種:   Pseudomonas syringae (シュードモナス・シリンガエ) Pseudomonas syringae (シュードモナス・シリンガエ)組換株: pathovar phaseolicola / 組換プラスミド: pLM358 |
| 配列 | UniProtKB: Major inner protein P1 |
-分子 #2: P2 protein from bacteriophage phi6
| 分子 | 名称: P2 protein from bacteriophage phi6 / タイプ: protein_or_peptide / ID: 2 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas phage phi6 (ファージ) / 別称: bacteriophage phi6 Pseudomonas phage phi6 (ファージ) / 別称: bacteriophage phi6 |
| 分子量 | 理論値: 75 KDa |
| 組換発現 | 生物種:   Pseudomonas syringae (シュードモナス・シリンガエ) Pseudomonas syringae (シュードモナス・シリンガエ)組換株: pathovar phaseolicola / 組換プラスミド: pLM358 |
| 配列 | UniProtKB:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
-分子 #3: P4 protein from bacteriophage phi6
| 分子 | 名称: P4 protein from bacteriophage phi6 / タイプ: protein_or_peptide / ID: 3 / 集合状態: hexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas phage phi6 (ファージ) / 別称: bacteriophage phi6 Pseudomonas phage phi6 (ファージ) / 別称: bacteriophage phi6 |
| 分子量 | 理論値: 35 KDa |
| 組換発現 | 生物種:   Pseudomonas syringae (シュードモナス・シリンガエ) Pseudomonas syringae (シュードモナス・シリンガエ)組換株: pathovar phaseolicola / 組換プラスミド: pLM358 |
| 配列 | UniProtKB: Packaging enzyme P4 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.4 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 50 mM Tris |
| グリッド | 詳細: glow discharged Cflat grid (CF-2/1-2C-T) |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK IV / 手法: Blot 4 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 160000 Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 160000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF QUANTUM LS エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| 温度 | 最低: 81 K / 最高: 120 K / 平均: 81 K |
| 詳細 | dose rate 6-8 e-/pix/s |
| 日付 | 2014年6月12日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - サンプリング間隔: 5 µm / 実像数: 834 / 平均電子線量: 16 e/Å2 詳細: Every image is the average of 22 frames recorded by the direct electron detector ビット/ピクセル: 16 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X