+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2846 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
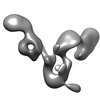
| タイトル | Three-dimensional structure of the autophagic phosphatidylinositol 3-kinase complex. | |||||||||
 マップデータ マップデータ | Three-dimensional structure of the autophagic phosphatidylinositol 3-kinase complex, PI3KC3-C1. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  kinase (キナーゼ) / kinase (キナーゼ) /  lipid kinase (キナーゼ) / lipid kinase (キナーゼ) /  protein kinase (プロテインキナーゼ) / protein kinase (プロテインキナーゼ) /  autophagy (オートファジー) / autophagy (オートファジー) /  phosphatidylinositol 3-kinase (PI3キナーゼ) / phosphatidylinositol 3-kinase (PI3キナーゼ) /  PI3K (PI3キナーゼ) / PI3K (PI3キナーゼ) /  phosphatidylinositol 3-phosphate / PI(3)P phosphatidylinositol 3-phosphate / PI(3)P | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 extrinsic component of omegasome membrane / phosphatidylinositol 3-kinase inhibitor activity / regulation of triglyceride metabolic process / extrinsic component of omegasome membrane / phosphatidylinositol 3-kinase inhibitor activity / regulation of triglyceride metabolic process /  extrinsic component of phagophore assembly site membrane / nucleus-vacuole junction / cellular response to aluminum ion / Toll Like Receptor 9 (TLR9) Cascade / extrinsic component of phagophore assembly site membrane / nucleus-vacuole junction / cellular response to aluminum ion / Toll Like Receptor 9 (TLR9) Cascade /  protein lipidation / Synthesis of PIPs at the late endosome membrane / Synthesis of PIPs at the early endosome membrane ... protein lipidation / Synthesis of PIPs at the late endosome membrane / Synthesis of PIPs at the early endosome membrane ... extrinsic component of omegasome membrane / phosphatidylinositol 3-kinase inhibitor activity / regulation of triglyceride metabolic process / extrinsic component of omegasome membrane / phosphatidylinositol 3-kinase inhibitor activity / regulation of triglyceride metabolic process /  extrinsic component of phagophore assembly site membrane / nucleus-vacuole junction / cellular response to aluminum ion / Toll Like Receptor 9 (TLR9) Cascade / extrinsic component of phagophore assembly site membrane / nucleus-vacuole junction / cellular response to aluminum ion / Toll Like Receptor 9 (TLR9) Cascade /  protein lipidation / Synthesis of PIPs at the late endosome membrane / Synthesis of PIPs at the early endosome membrane / phosphatidylinositol 3-kinase complex, class III / cellular response to oxygen-glucose deprivation / phosphatidylinositol 3-kinase complex, class III, type II / phosphatidylinositol 3-kinase complex, class III, type I / mitochondria-associated endoplasmic reticulum membrane contact site / positive regulation by host of viral genome replication / response to mitochondrial depolarisation / positive regulation of attachment of mitotic spindle microtubules to kinetochore / cytoplasmic side of mitochondrial outer membrane / negative regulation of lysosome organization / Synthesis of PIPs at the Golgi membrane / positive regulation of autophagosome assembly / engulfment of apoptotic cell / negative regulation of autophagosome assembly / receptor catabolic process / regulation of protein complex stability / phosphatidylinositol kinase activity / protein localization to phagophore assembly site / phagophore assembly site membrane / protein targeting to vacuole / protein targeting to lysosome / suppression by virus of host autophagy / early endosome to late endosome transport / cellular response to nitrogen starvation / late endosome to vacuole transport / SMAD protein signal transduction / phosphatidylinositol-mediated signaling / phagophore assembly site / Translation of Replicase and Assembly of the Replication Transcription Complex / response to iron(II) ion / negative regulation of programmed cell death / protein lipidation / Synthesis of PIPs at the late endosome membrane / Synthesis of PIPs at the early endosome membrane / phosphatidylinositol 3-kinase complex, class III / cellular response to oxygen-glucose deprivation / phosphatidylinositol 3-kinase complex, class III, type II / phosphatidylinositol 3-kinase complex, class III, type I / mitochondria-associated endoplasmic reticulum membrane contact site / positive regulation by host of viral genome replication / response to mitochondrial depolarisation / positive regulation of attachment of mitotic spindle microtubules to kinetochore / cytoplasmic side of mitochondrial outer membrane / negative regulation of lysosome organization / Synthesis of PIPs at the Golgi membrane / positive regulation of autophagosome assembly / engulfment of apoptotic cell / negative regulation of autophagosome assembly / receptor catabolic process / regulation of protein complex stability / phosphatidylinositol kinase activity / protein localization to phagophore assembly site / phagophore assembly site membrane / protein targeting to vacuole / protein targeting to lysosome / suppression by virus of host autophagy / early endosome to late endosome transport / cellular response to nitrogen starvation / late endosome to vacuole transport / SMAD protein signal transduction / phosphatidylinositol-mediated signaling / phagophore assembly site / Translation of Replicase and Assembly of the Replication Transcription Complex / response to iron(II) ion / negative regulation of programmed cell death /  オートファゴソーム / オートファゴソーム /  PI3キナーゼ / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity / PI3キナーゼ / phosphatidylinositol-3-phosphate biosynthetic process / 1-phosphatidylinositol-3-kinase activity /  オートファジー / post-transcriptional regulation of gene expression / mitotic metaphase chromosome alignment / lysosome organization / autophagosome membrane docking / endosome to lysosome transport / cytoplasmic pattern recognition receptor signaling pathway / positive regulation of cardiac muscle hypertrophy / p38MAPK cascade / オートファジー / post-transcriptional regulation of gene expression / mitotic metaphase chromosome alignment / lysosome organization / autophagosome membrane docking / endosome to lysosome transport / cytoplasmic pattern recognition receptor signaling pathway / positive regulation of cardiac muscle hypertrophy / p38MAPK cascade /  axoneme / autophagosome maturation / autophagosome membrane / phosphatidylinositol phosphate biosynthetic process / axoneme / autophagosome maturation / autophagosome membrane / phosphatidylinositol phosphate biosynthetic process /  マイトファジー / マイトファジー /  autophagosome assembly / RHO GTPases Activate NADPH Oxidases / PI3K Cascade / neuron development / autophagosome assembly / RHO GTPases Activate NADPH Oxidases / PI3K Cascade / neuron development /  オートファゴソーム / negative regulation of reactive oxygen species metabolic process / response to vitamin E / オートファゴソーム / negative regulation of reactive oxygen species metabolic process / response to vitamin E /  regulation of macroautophagy / cellular response to glucose starvation / cellular defense response / regulation of macroautophagy / cellular response to glucose starvation / cellular defense response /  phosphatidylinositol 3-kinase binding / phagocytic vesicle / amyloid-beta metabolic process / positive regulation of autophagy / protein-membrane adaptor activity / positive regulation of intrinsic apoptotic signaling pathway / JNK cascade / cellular response to epidermal growth factor stimulus / cellular response to copper ion / cellular response to amino acid starvation / cellular response to starvation / negative regulation of protein phosphorylation / phosphatidylinositol 3-kinase/protein kinase B signal transduction / phosphatidylinositol 3-kinase binding / phagocytic vesicle / amyloid-beta metabolic process / positive regulation of autophagy / protein-membrane adaptor activity / positive regulation of intrinsic apoptotic signaling pathway / JNK cascade / cellular response to epidermal growth factor stimulus / cellular response to copper ion / cellular response to amino acid starvation / cellular response to starvation / negative regulation of protein phosphorylation / phosphatidylinositol 3-kinase/protein kinase B signal transduction /  regulation of cytokinesis / regulation of cytokinesis /  regulation of autophagy / Antigen Presentation: Folding, assembly and peptide loading of class I MHC / regulation of autophagy / Antigen Presentation: Folding, assembly and peptide loading of class I MHC /  オートファジー / response to lead ion / オートファジー / response to lead ion /  regulation of protein phosphorylation / regulation of protein phosphorylation /  ゴルジ体 / ISG15 antiviral mechanism / cellular response to hydrogen peroxide / ゴルジ体 / ISG15 antiviral mechanism / cellular response to hydrogen peroxide /  オートファジー / オートファジー /  エンドサイトーシス / エンドサイトーシス /  ペルオキシソーム / phagocytic vesicle membrane / microtubule cytoskeleton / protein-macromolecule adaptor activity / late endosome / ペルオキシソーム / phagocytic vesicle membrane / microtubule cytoskeleton / protein-macromolecule adaptor activity / late endosome /  GTPase binding / Translation of Replicase and Assembly of the Replication Transcription Complex GTPase binding / Translation of Replicase and Assembly of the Replication Transcription Complex類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 27.5 Å ネガティブ染色法 / 解像度: 27.5 Å | |||||||||
 データ登録者 データ登録者 | Baskaran S / Carlson L-A / Stjepanovic G / Young LN / Kim DJ / Grob P / Stanley RE / Nogales E / Hurley JH | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2014 ジャーナル: Elife / 年: 2014タイトル: Architecture and dynamics of the autophagic phosphatidylinositol 3-kinase complex. 著者: Sulochanadevi Baskaran / Lars-Anders Carlson / Goran Stjepanovic / Lindsey N Young / Do Jin Kim / Patricia Grob / Robin E Stanley / Eva Nogales / James H Hurley /  要旨: The class III phosphatidylinositol 3-kinase complex I (PI3KC3-C1) that functions in early autophagy consists of the lipid kinase VPS34, the scaffolding protein VPS15, the tumor suppressor BECN1, and ...The class III phosphatidylinositol 3-kinase complex I (PI3KC3-C1) that functions in early autophagy consists of the lipid kinase VPS34, the scaffolding protein VPS15, the tumor suppressor BECN1, and the autophagy-specific subunit ATG14. The structure of the ATG14-containing PI3KC3-C1 was determined by single-particle EM, revealing a V-shaped architecture. All of the ordered domains of VPS34, VPS15, and BECN1 were mapped by MBP tagging. The dynamics of the complex were defined using hydrogen-deuterium exchange, revealing a novel 20-residue ordered region C-terminal to the VPS34 C2 domain. VPS15 organizes the complex and serves as a bridge between VPS34 and the ATG14:BECN1 subcomplex. Dynamic transitions occur in which the lipid kinase domain is ejected from the complex and VPS15 pivots at the base of the V. The N-terminus of BECN1, the target for signaling inputs, resides near the pivot point. These observations provide a framework for understanding the allosteric regulation of lipid kinase activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2846.map.gz emd_2846.map.gz | 3.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2846-v30.xml emd-2846-v30.xml emd-2846.xml emd-2846.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2846.tif emd_2846.tif | 86.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2846 http://ftp.pdbj.org/pub/emdb/structures/EMD-2846 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2846 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2846 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2846.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2846.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Three-dimensional structure of the autophagic phosphatidylinositol 3-kinase complex, PI3KC3-C1. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.01 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Autophagic phosphatidylinositol 3-kinase complex, PI3KC3-C1.
| 全体 | 名称: Autophagic phosphatidylinositol 3-kinase complex, PI3KC3-C1. |
|---|---|
| 要素 |
|
-超分子 #1000: Autophagic phosphatidylinositol 3-kinase complex, PI3KC3-C1.
| 超分子 | 名称: Autophagic phosphatidylinositol 3-kinase complex, PI3KC3-C1. タイプ: sample / ID: 1000 / 集合状態: heterotetramer / Number unique components: 4 |
|---|---|
| 分子量 | 理論値: 361.8 KDa |
-分子 #1: phosphatidylinositol 3-kinase, catalytic subunit type 3
| 分子 | 名称: phosphatidylinositol 3-kinase, catalytic subunit type 3 タイプ: protein_or_peptide / ID: 1 / Name.synonym: VPS34, PI3KC3 / コピー数: 1 / 集合状態: one component of heterotetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane |
| 分子量 | 理論値: 102 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG |
| 配列 | UniProtKB: Phosphatidylinositol 3-kinase catalytic subunit type 3 GO: 1-phosphatidylinositol-3-kinase activity, phosphatidylinositol 3-kinase complex, class III, type I,  オートファジー, オートファジー,  autophagosome assembly autophagosome assembly |
-分子 #2: phosphoinositide-3-kinase, regulatory subunit 4
| 分子 | 名称: phosphoinositide-3-kinase, regulatory subunit 4 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: VPS15, p150, PIK3R4 / コピー数: 1 / 集合状態: one component of heterotetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane |
| 分子量 | 理論値: 153 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG |
| 配列 | UniProtKB: Phosphoinositide 3-kinase regulatory subunit 4 GO: phosphatidylinositol 3-kinase complex, class III, type I,  オートファジー, オートファジー,  autophagosome assembly autophagosome assembly |
-分子 #3: beclin1
| 分子 | 名称: beclin1 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: BECN1 / コピー数: 1 / 集合状態: one component of heterotetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane |
| 分子量 | 理論値: 52 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG |
| 配列 | UniProtKB:  Beclin-1 Beclin-1GO: phosphatidylinositol 3-kinase complex, class III, type I,  オートファジー, オートファジー,  autophagosome assembly autophagosome assembly |
-分子 #4: autophagy related 14
| 分子 | 名称: autophagy related 14 / タイプ: protein_or_peptide / ID: 4 / Name.synonym: ATG14, ATG14L, BARKOR / コピー数: 1 / 集合状態: one component of heterotetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane Homo sapiens (ヒト) / 別称: Human / Organelle: autophagosome / 細胞中の位置: autophagosome membrane |
| 分子量 | 理論値: 55 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: pCAG |
| 配列 | UniProtKB: Beclin 1-associated autophagy-related key regulator GO: phosphatidylinositol 3-kinase complex, class III, type I,  オートファジー, オートファジー,  autophagosome assembly autophagosome assembly |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM Tris-HCl, 200 mM NaCl, 2 mM MgCl2, 1 mM TCEP, 3% trehalose |
| 染色 | タイプ: NEGATIVE 詳細: Protein was incubated on grids for 30s, followed by sequential 10 s incubations on four 50 ul drops of 1% uranyl formate. The stained grids were blotted to near dryness with a filter paper and air-dried. |
| グリッド | 詳細: Continuous carbon, plasma cleaned for 10 s in a Gatan Solarus at 10% O2. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 100333 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 80000 Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle max: 45 |
| 温度 | 最低: 292 K / 最高: 294 K / 平均: 293 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 80,000 times magnification. |
| 日付 | 2014年5月28日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 1614 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 32 |
| Tilt angle min | 0 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: tilt-dependent phase flip |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 27.5 Å / 解像度の算出法: OTHER ソフトウェア - 名称: Spider, EMAN, EMAN2, IMAGIC, RELION 詳細: Final map calculated in RELION using a single 3D class. Data acquired at 0, 30, and 45 degrees were used to refine an initial RCT model low-pass filtered to 80A. Gold standard FSC calculated using RELION. 使用した粒子像数: 38745 |
| 詳細 | The final three-dimensional structure was calculated in RELION using a single 3D class. Starting model for this calculation was a random conical tilt model low-pass filtered to 80A. |
 ムービー
ムービー コントローラー
コントローラー