+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure INO80core Hexasome complex Arp5 Ies6 refinement state2 | |||||||||||||||
 マップデータ マップデータ | ctINO80 hexasome complex Arp5 Ies6 refinement sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | ATP-dependent chromatin remodeler /  DNA BINDING PROTEIN (DNA結合タンパク質) DNA BINDING PROTEIN (DNA結合タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / attachment of spindle microtubules to kinetochore / Ino80 complex / attachment of mitotic spindle microtubules to kinetochore /  紡錘体 / 紡錘体 /  動原体 / 動原体 /  クロマチンリモデリング クロマチンリモデリング類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) | |||||||||||||||
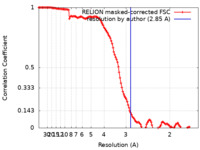
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.85 Å クライオ電子顕微鏡法 / 解像度: 2.85 Å | |||||||||||||||
 データ登録者 データ登録者 | Zhang M / Jungblut A / Hoffmann T / Eustermann S | |||||||||||||||
| 資金援助 |  ドイツ, European Union, 4件 ドイツ, European Union, 4件
| |||||||||||||||
 引用 引用 | ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2010 タイトル: Features and development of Coot. 著者: P Emsley / B Lohkamp / W G Scott / K Cowtan /  要旨: Coot is a molecular-graphics application for model building and validation of biological macromolecules. The program displays electron-density maps and atomic models and allows model manipulations ...Coot is a molecular-graphics application for model building and validation of biological macromolecules. The program displays electron-density maps and atomic models and allows model manipulations such as idealization, real-space refinement, manual rotation/translation, rigid-body fitting, ligand search, solvation, mutations, rotamers and Ramachandran idealization. Furthermore, tools are provided for model validation as well as interfaces to external programs for refinement, validation and graphics. The software is designed to be easy to learn for novice users, which is achieved by ensuring that tools for common tasks are 'discoverable' through familiar user-interface elements (menus and toolbars) or by intuitive behaviour (mouse controls). Recent developments have focused on providing tools for expert users, with customisable key bindings, extensions and an extensive scripting interface. The software is under rapid development, but has already achieved very widespread use within the crystallographic community. The current state of the software is presented, with a description of the facilities available and of some of the underlying methods employed. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17028.map.gz emd_17028.map.gz | 8.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17028-v30.xml emd-17028-v30.xml emd-17028.xml emd-17028.xml | 28.3 KB 28.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17028_fsc.xml emd_17028_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17028.png emd_17028.png | 58.5 KB | ||
| その他 |  emd_17028_additional_1.map.gz emd_17028_additional_1.map.gz emd_17028_half_map_1.map.gz emd_17028_half_map_1.map.gz emd_17028_half_map_2.map.gz emd_17028_half_map_2.map.gz | 140.4 MB 140.9 MB 140.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17028 http://ftp.pdbj.org/pub/emdb/structures/EMD-17028 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17028 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17028 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ootMC  8oo7C  8oo9C  8ooaC  8oocC  8oofC  8ookC  8oopC  8oorC  8oosC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17028.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17028.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ctINO80 hexasome complex Arp5 Ies6 refinement sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: ctINO80 hexasome complex Arp5 Ies6 refinement unsharpened map
| ファイル | emd_17028_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ctINO80 hexasome complex Arp5 Ies6 refinement unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
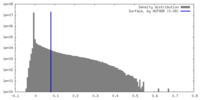
| 密度ヒストグラム |
-ハーフマップ: ctINO80 hexasome complex Arp5 Ies6 refinement half1 map
| ファイル | emd_17028_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ctINO80 hexasome complex Arp5 Ies6 refinement half1 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
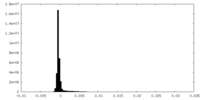
| 密度ヒストグラム |
-ハーフマップ: ctINO80 hexasome complex Arp5 Ies6 refinement half2 map
| ファイル | emd_17028_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ctINO80 hexasome complex Arp5 Ies6 refinement half2 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
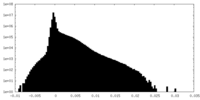
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : INO80 core module in complex with hexasome
| 全体 | 名称: INO80 core module in complex with hexasome |
|---|---|
| 要素 |
|
-超分子 #1: INO80 core module in complex with hexasome
| 超分子 | 名称: INO80 core module in complex with hexasome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: 11-subunit ct INO80 contains two modules (core and Arp8 module) Each module was picked and analyzed separately The core module + hexasome has an overall weight of 0.861MDa The 11-subunit ct ...詳細: 11-subunit ct INO80 contains two modules (core and Arp8 module) Each module was picked and analyzed separately The core module + hexasome has an overall weight of 0.861MDa The 11-subunit ct INO80 + hexasome has an overall weight of 1.1MDa Ino80, Ies2, Ies6, Ies4,Arp6, Rvb1, Rvb2, Arp8, Arp4, Actin, Taf14 Hexasome DNA, 2xH3, 2xH4, H2A, H2B |
|---|---|
| 分子量 | 理論値: 861 KDa |
-超分子 #2: Arp6-Ies6 of INO80 complex
| 超分子 | 名称: Arp6-Ies6 of INO80 complex / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
-分子 #1: Chromatin-remodeling complex subunit IES6
| 分子 | 名称: Chromatin-remodeling complex subunit IES6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
| 分子量 | 理論値: 23.127523 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSNPDAQSAQ AAHQALVEQL DLHSIHKTFR NPNWRPNQRR NKTIKAILGE SQRKEASSTS AVATPRADDN GGGSGADTPA NNDNNDGLS TSGTSTPANG NGSGAGTPAS NGQPNLAQAS RSLQKLVLEK SLASAQAPDK KAANGFASSA PTATYTNIES A PSLAPMKH ...文字列: MSNPDAQSAQ AAHQALVEQL DLHSIHKTFR NPNWRPNQRR NKTIKAILGE SQRKEASSTS AVATPRADDN GGGSGADTPA NNDNNDGLS TSGTSTPANG NGSGAGTPAS NGQPNLAQAS RSLQKLVLEK SLASAQAPDK KAANGFASSA PTATYTNIES A PSLAPMKH YCDVTGLPAP YLDPKTRLRY HNKEIFAMIR NLPQGMGEQF LEARGAHTVL K UniProtKB: Vps72/YL1 C-terminal domain-containing protein |
-分子 #2: Actin-related protein 5
| 分子 | 名称: Actin-related protein 5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
| 分子量 | 理論値: 87.773086 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAPSAVAEPP PIPQRDEPWK RLPPPTVYPV KEARFEKYIP PQLDGRERAL AQPPGQVAIV IDNGSHSVRA GWNFEDKPRL AIPPIMSKY RDRKMGKTFS FAGSDCYADT TARSHIRNAF EAGTGIVSNW DVMEHVLDYV FVKLGMNECD GAIDMPIVMT E AVANLPYS ...文字列: MAPSAVAEPP PIPQRDEPWK RLPPPTVYPV KEARFEKYIP PQLDGRERAL AQPPGQVAIV IDNGSHSVRA GWNFEDKPRL AIPPIMSKY RDRKMGKTFS FAGSDCYADT TARSHIRNAF EAGTGIVSNW DVMEHVLDYV FVKLGMNECD GAIDMPIVMT E AVANLPYS RKSMSEIIFE CYGAPSLVYG IDSLFSFRHN QGQTGLVVSS SYSATHVIPV YNRKALLSQA IRLNWGGWHM AE YMLKLLK LKYYTGFPGK LNSSQTEHMV RDFCYVSLDY DRELAGYLDW TGLEDRERIV QYPYTEEVVV QKTEEELARI AER KKESGR RLQEQAAKMR LERLMKKEQE LEYYKDIQRR MQGESKKEIK RLLDEAELKD EAALERVIRD LERSIKRARQ KDLG EPEEE EVPDFSLLDV PDDQLDEAGL RQKRQQRLLK SNWEARQRAK AEKEAEKARL AEEARLDEER RKNDLEGWLE EKRQL RLAK LNQLKERERL KADLGNRKSL ASQIRMKNIA NLASDNPTGS GSRKRRRGGA GADQDDDFGA DDADWGVYRS VAIGAN KGD DSDDEEGEED LEAAIRSLEN DLLRYDKTFS YDMTLDAQRD WSKSLLHAFR YGPRPFDPSS QAETHRVHLN VERIRVP EV LFQPAAIAGV DQAGLVEIAG DILCQRLPSL PGIQDAPDAF LRDVFLTGGN TLFQNFDERL RQGLMALLPV GAPLRVRR A QDAILDAWRG AAGWACTEEA KAAWITREEY LEKGGEYIKE HDLGNAFA UniProtKB: Uncharacterized protein |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.88 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 30mM HEPES, pH7.5 50mM NaCl 0.25mM CaCl2 0.25mM DTT 2mM ADP 3.3mM MgCl2 10mM NaF 2mM AlCl3 0.05% octyl-beta-glucoside |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 15 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV 詳細: wait time of 5s, blot force at 3, and a blot time of 2s with Whatman blotting paper (Cytiva, CAT No. 10311807). |
| 詳細 | 11-subunit ctINO80 reconstituted with hexasome |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 15384 / 平均電子線量: 50.36 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)