+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
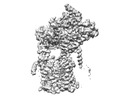
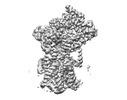
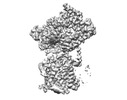
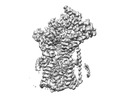
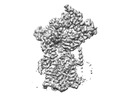
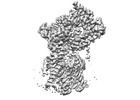
| タイトル | Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  translocon (トランスロコン) / translocon (トランスロコン) /  inhibitor (酵素阻害剤) / inhibitor (酵素阻害剤) /  protein translocation / protein translocation /  PROTEIN TRANSPORT / PROTEIN TRANSPORT-INHIBITOR complex PROTEIN TRANSPORT / PROTEIN TRANSPORT-INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endoplasmic reticulum Sec complex / endoplasmic reticulum quality control compartment / pronephric nephron development / cotranslational protein targeting to membrane / Ssh1 translocon complex / Sec61 translocon complex / protein targeting to ER / protein insertion into ER membrane /  post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation ...endoplasmic reticulum Sec complex / endoplasmic reticulum quality control compartment / pronephric nephron development / cotranslational protein targeting to membrane / Ssh1 translocon complex / Sec61 translocon complex / protein targeting to ER / protein insertion into ER membrane / post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation ...endoplasmic reticulum Sec complex / endoplasmic reticulum quality control compartment / pronephric nephron development / cotranslational protein targeting to membrane / Ssh1 translocon complex / Sec61 translocon complex / protein targeting to ER / protein insertion into ER membrane /  post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation / post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation /  signal sequence binding / SRP-dependent cotranslational protein targeting to membrane / signal sequence binding / SRP-dependent cotranslational protein targeting to membrane /  post-translational protein targeting to membrane, translocation / endoplasmic reticulum organization / retrograde protein transport, ER to cytosol / post-translational protein targeting to membrane, translocation / endoplasmic reticulum organization / retrograde protein transport, ER to cytosol /  epidermal growth factor binding / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane / protein transmembrane transporter activity / : / epidermal growth factor binding / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane / protein transmembrane transporter activity / : /  calcium channel activity / calcium channel activity /  ribosome binding / ER-Phagosome pathway / endoplasmic reticulum membrane / ribosome binding / ER-Phagosome pathway / endoplasmic reticulum membrane /  小胞体 / 小胞体 /  RNA binding / RNA binding /  生体膜 / 生体膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.22 Å クライオ電子顕微鏡法 / 解像度: 3.22 Å | |||||||||
 データ登録者 データ登録者 | Park E / Itskanov S | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: A common mechanism of Sec61 translocon inhibition by small molecules. 著者: Samuel Itskanov / Laurie Wang / Tina Junne / Rumi Sherriff / Li Xiao / Nicolas Blanchard / Wei Q Shi / Craig Forsyth / Dominic Hoepfner / Martin Spiess / Eunyong Park /    要旨: The Sec61 complex forms a protein-conducting channel in the endoplasmic reticulum membrane that is required for secretion of soluble proteins and production of many membrane proteins. Several natural ...The Sec61 complex forms a protein-conducting channel in the endoplasmic reticulum membrane that is required for secretion of soluble proteins and production of many membrane proteins. Several natural and synthetic small molecules specifically inhibit Sec61, generating cellular effects that are useful for therapeutic purposes, but their inhibitory mechanisms remain unclear. Here we present near-atomic-resolution structures of human Sec61 inhibited by a comprehensive panel of structurally distinct small molecules-cotransin, decatransin, apratoxin, ipomoeassin, mycolactone, cyclotriazadisulfonamide and eeyarestatin. All inhibitors bind to a common lipid-exposed pocket formed by the partially open lateral gate and plug domain of Sec61. Mutations conferring resistance to the inhibitors are clustered at this binding pocket. The structures indicate that Sec61 inhibitors stabilize the plug domain in a closed state, thereby preventing the protein-translocation pore from opening. Our study provides the atomic details of Sec61-inhibitor interactions and the structural framework for further pharmacological studies and drug design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27589.map.gz emd_27589.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27589-v30.xml emd-27589-v30.xml emd-27589.xml emd-27589.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27589_fsc.xml emd_27589_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27589.png emd_27589.png | 93.1 KB | ||
| その他 |  emd_27589_half_map_1.map.gz emd_27589_half_map_1.map.gz emd_27589_half_map_2.map.gz emd_27589_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27589 http://ftp.pdbj.org/pub/emdb/structures/EMD-27589 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27589 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27589 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8do3MC  8dnvC  8dnwC  8dnxC  8dnyC  8dnzC  8do0C  8do1C  8do2C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27589.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27589.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Cryo-EM structure of the human Sec61 complex inhibited...
| ファイル | emd_27589_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM structure of the human Sec61 complex inhibited...
| ファイル | emd_27589_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A human-yeast chimeric Sec complex treated with eeyarestatin I
| 全体 | 名称: A human-yeast chimeric Sec complex treated with eeyarestatin I |
|---|---|
| 要素 |
|
-超分子 #1: A human-yeast chimeric Sec complex treated with eeyarestatin I
| 超分子 | 名称: A human-yeast chimeric Sec complex treated with eeyarestatin I タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / Organelle: endoplasmic reticulum Homo sapiens (ヒト) / Organelle: endoplasmic reticulum |
-分子 #1: Protein transport protein Sec61 subunit gamma
| 分子 | 名称: Protein transport protein Sec61 subunit gamma / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 7.752325 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MDQVMQFVEP SRQFVKDSIR LVKRCTKPDR KEFQKIAMAT AIGFAIMGFI GFFVKLIHIP INNIIVGG UniProtKB:  Protein transport protein Sec61 subunit gamma Protein transport protein Sec61 subunit gamma |
-分子 #2: Protein transport protein Sec61 subunit beta
| 分子 | 名称: Protein transport protein Sec61 subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 9.987456 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MPGPTPSGTN VGSSGRSPSK AVAARAAGST VRQRKNASCG TRSAGRTTSA GTGGMWRFYT EDSPGLKVGP VPVLVMSLLF IASVFMLHI WGKYTRS UniProtKB:  Protein transport protein Sec61 subunit beta Protein transport protein Sec61 subunit beta |
-分子 #3: Protein transport protein Sec61 subunit alpha isoform 1
| 分子 | 名称: Protein transport protein Sec61 subunit alpha isoform 1 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 52.202438 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAIKFLEVIK PFCVILPEIQ KPERKIQFKE KVLWTAITLF IFLVCCQIPL FGIMSSDSAD PFYWMRVILA SNRGTLMELG ISPIVTSGL IMQLLAGAKI IEVGDTPKDR ALFNGAQKLF GMIITIGQSI VYVMTGMYGD PSEMGAGICL LITIQLFVAG L IVLLLDEL ...文字列: MAIKFLEVIK PFCVILPEIQ KPERKIQFKE KVLWTAITLF IFLVCCQIPL FGIMSSDSAD PFYWMRVILA SNRGTLMELG ISPIVTSGL IMQLLAGAKI IEVGDTPKDR ALFNGAQKLF GMIITIGQSI VYVMTGMYGD PSEMGAGICL LITIQLFVAG L IVLLLDEL LQKGYGLGSG ISLFIATNIC ETIVWKAFSP TTVNTGRGME FEGAIIALFH LLATRTDKVR ALREAFYRQN LP NLMNLIA TIFVFAVVIY FQGFRYELPI RSTKVRGQIG IYPIKLFYTS NIPIILQSAL VSNLYVISQM LSARFSGNLL VSL LGTWSD TSSGGPARAY PVGGLCYYLS PPESFGSVLE DPVHAVVYIV FMLGSCAFFS KTWIEVSGSS PRDIAKQFKD QGMV INGKR ETSIYRELKK IIPTAAAFGG LCIGALSVLA DFLGAIGSGT GILLAVTIIY QYFEIFVKEQ SEVGSMGALL F UniProtKB:  Protein transport protein Sec61 subunit alpha isoform 1 Protein transport protein Sec61 subunit alpha isoform 1 |
-分子 #4: N'-(4-chlorophenyl)-N-[(4R)-3-(4-chlorophenyl)-5,5-dimethyl-1-(2-...
| 分子 | 名称: N'-(4-chlorophenyl)-N-[(4R)-3-(4-chlorophenyl)-5,5-dimethyl-1-(2-{(2E)-2-[(2E)-3-(5-nitrofuran-2-yl)prop-2-en-1-ylidene]hydrazinyl}-2-oxoethyl)-2-oxoimidazolidin-4-yl]-N-hydroxyurea タイプ: ligand / ID: 4 / コピー数: 1 / 式: SWR |
|---|---|
| 分子量 | 理論値: 631.444 Da |
| Chemical component information | 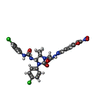 ChemComp-SWR: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 4 seconds before plunging. | ||||||||||||||||||
| 詳細 | Reconsitituted into a peptidisc. Monodisperse peak from a Superose 6 column. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8do3: |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X


















