+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6yxd | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Room temperature structure of human adiponectin receptor 2 (ADIPOR2) at 2.9 A resolution determined by Serial Crystallography (SSX) using CrystalDirect | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  Adiponectin receptor / ADIPOR / serial synchrotron crystallography / SSX / CrystalDirect / LCP crystallization / in meso / Adiponectin receptor / ADIPOR / serial synchrotron crystallography / SSX / CrystalDirect / LCP crystallization / in meso /  Membrane proteins (膜タンパク質) Membrane proteins (膜タンパク質) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 adiponectin binding / adipokinetic hormone receptor activity / adiponectin-activated signaling pathway / AMPK inhibits chREBP transcriptional activation activity / vascular wound healing / adiponectin binding / adipokinetic hormone receptor activity / adiponectin-activated signaling pathway / AMPK inhibits chREBP transcriptional activation activity / vascular wound healing /  Β酸化 / hormone-mediated signaling pathway / Β酸化 / hormone-mediated signaling pathway /  glucose homeostasis / glucose homeostasis /  signaling receptor activity / positive regulation of cold-induced thermogenesis ... signaling receptor activity / positive regulation of cold-induced thermogenesis ... adiponectin binding / adipokinetic hormone receptor activity / adiponectin-activated signaling pathway / AMPK inhibits chREBP transcriptional activation activity / vascular wound healing / adiponectin binding / adipokinetic hormone receptor activity / adiponectin-activated signaling pathway / AMPK inhibits chREBP transcriptional activation activity / vascular wound healing /  Β酸化 / hormone-mediated signaling pathway / Β酸化 / hormone-mediated signaling pathway /  glucose homeostasis / glucose homeostasis /  signaling receptor activity / positive regulation of cold-induced thermogenesis / identical protein binding / signaling receptor activity / positive regulation of cold-induced thermogenesis / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.9 Å 分子置換 / 解像度: 2.9 Å | ||||||||||||
 データ登録者 データ登録者 | Healey, R.D. / Basu, S. / Humm, A.S. / Leyrat, C. / Dupeux, F. / Pica, A. / Granier, S. / Marquez, J.A. | ||||||||||||
| 資金援助 | European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Rep Methods / 年: 2021 ジャーナル: Cell Rep Methods / 年: 2021タイトル: An automated platform for structural analysis of membrane proteins through serial crystallography. 著者: Healey, R.D. / Basu, S. / Humm, A.S. / Leyrat, C. / Cong, X. / Golebiowski, J. / Dupeux, F. / Pica, A. / Granier, S. / Marquez, J.A. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6yxd.cif.gz 6yxd.cif.gz | 225.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6yxd.ent.gz pdb6yxd.ent.gz | 179.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6yxd.json.gz 6yxd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yx/6yxd https://data.pdbj.org/pub/pdb/validation_reports/yx/6yxd ftp://data.pdbj.org/pub/pdb/validation_reports/yx/6yxd ftp://data.pdbj.org/pub/pdb/validation_reports/yx/6yxd | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  / Progestin and adipoQ receptor family member 2 / Progestin and adipoQ receptor family member II / Progestin and adipoQ receptor family member 2 / Progestin and adipoQ receptor family member II分子量: 33097.793 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: ADIPOR2, PAQR2 Homo sapiens (ヒト) / 遺伝子: ADIPOR2, PAQR2発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ)参照: UniProt: Q86V24 |
|---|
-抗体 , 2種, 2分子 HL
| #2: 抗体 |  抗体 抗体分子量: 13160.635 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
|---|---|
| #3: 抗体 |  抗体 抗体分子量: 11647.850 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)発現宿主:   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
-非ポリマー , 5種, 63分子 
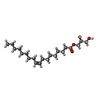







| #4: 化合物 | ChemComp-ZN / | ||||||
|---|---|---|---|---|---|---|---|
| #5: 化合物 | ChemComp-OLB / ( #6: 化合物 | ChemComp-OLA / |  オレイン酸 オレイン酸#7: 化合物 | ChemComp-GOL / |  グリセリン グリセリン#8: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.77 Å3/Da / 溶媒含有率: 67.39 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 詳細: CrystalDirect-2 plates (MiTeGen) in 30 or 50 nL bolus overlaid with 600 nl precipitant solution. 42.3% PEG 400, 110 mM potassium citrate, 100 mM HEPES pH 7.0 for ADIPOR2. |
-データ収集
| 回折 | 平均測定温度: 293 K / Serial crystal experiment: Y |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / ビームライン: P14 (MX2) / 波長: 0.988 Å / ビームライン: P14 (MX2) / 波長: 0.988 Å |
| 検出器 | タイプ: DECTRIS EIGER2 X 4M / 検出器: PIXEL / 日付: 2019年7月1日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.988 Å / 相対比: 1 : 0.988 Å / 相対比: 1 |
| 反射 | 解像度: 2.9→50 Å / Num. obs: 15292 / % possible obs: 100 % / 冗長度: 130.57 % / CC1/2: 0.98 / Net I/σ(I): 8.6 |
| 反射 シェル | 解像度: 2.9→2.98 Å / Num. unique obs: 1971 / CC1/2: 0.737 |
| Serial crystallography sample delivery | 解説: CrystalDirect-2 plate / 手法: fixed target |
| Serial crystallography data reduction | Crystal hits: 28949 / Frames indexed: 25297 / Frames total: 382552 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5LWY 解像度: 2.9→50 Å / Cor.coef. Fo:Fc: 0.889 / Cor.coef. Fo:Fc free: 0.825 / 交差検証法: THROUGHOUT / SU Rfree Blow DPI: 0.419
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 48.56 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.39 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.9→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.9→3 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj









