+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6s1u | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of dimeric M-PMV protease C7A/D26N/C106A mutant in complex with inhibitor | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / HYDROLASE (加水分解酵素) /  Mason-Pfizer Monkey Virus / M-PMV / Mason-Pfizer Monkey Virus / M-PMV /  retrovirus (レトロウイルス科) / retrovirus (レトロウイルス科) /  retropepsin / retropepsin /  aspartic protease (アスパラギン酸プロテアーゼ) / aspartic protease (アスパラギン酸プロテアーゼ) /  dimerization / dimerization /  inhibitor (酵素阻害剤) / flap structure inhibitor (酵素阻害剤) / flap structure | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 dUTP diphosphatase / dUTP diphosphatase /  dUTP diphosphatase activity / dUTP diphosphatase activity /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ /  リボヌクレアーゼH / RNA stem-loop binding / DNA integration / リボヌクレアーゼH / RNA stem-loop binding / DNA integration /  逆転写酵素 / viral genome integration into host DNA / establishment of integrated proviral latency / 逆転写酵素 / viral genome integration into host DNA / establishment of integrated proviral latency /  RNA-directed DNA polymerase activity ... RNA-directed DNA polymerase activity ... dUTP diphosphatase / dUTP diphosphatase /  dUTP diphosphatase activity / dUTP diphosphatase activity /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ /  リボヌクレアーゼH / RNA stem-loop binding / DNA integration / リボヌクレアーゼH / RNA stem-loop binding / DNA integration /  逆転写酵素 / viral genome integration into host DNA / establishment of integrated proviral latency / 逆転写酵素 / viral genome integration into host DNA / establishment of integrated proviral latency /  RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity /  転移酵素; リンを含む基を移すもの; 核酸を移すもの / viral nucleocapsid / DNA recombination / structural constituent of virion / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / viral nucleocapsid / DNA recombination / structural constituent of virion /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  DNAポリメラーゼ / aspartic-type endopeptidase activity / DNAポリメラーゼ / aspartic-type endopeptidase activity /  DNA-directed DNA polymerase activity / symbiont entry into host cell / DNA-directed DNA polymerase activity / symbiont entry into host cell /  タンパク質分解 / タンパク質分解 /  DNA binding / zinc ion binding DNA binding / zinc ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Mason-Pfizer monkey virus (ウイルス) Mason-Pfizer monkey virus (ウイルス)synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Wosicki, S. / Gilski, M. / Jaskolski, M. / Zabranska, H. / Pichova, I. | ||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019タイトル: Comparison of a retroviral protease in monomeric and dimeric states. 著者: Wosicki, S. / Gilski, M. / Zabranska, H. / Pichova, I. / Jaskolski, M. #1:  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011タイトル: Crystal structure of a monomeric retroviral protease solved by protein folding game players. 著者: Khatib, F. / DiMaio, F. / Cooper, S. / Kazmierczyk, M. / Gilski, M. / Krzywda, S. / Zabranska, H. / Pichova, I. / Thompson, J. / Popovic, Z. / Jaskolski, M. / Baker, D. #2: ジャーナル: Acta Crystallogr.,Sect.D / 年: 2011 タイトル: High-resolution structure of a retroviral protease folded as a monomer. 著者: Gilski, M. / Kazmierczyk, M. / Krzywda, S. / Zabranska, H. / Cooper, S. / Popovic, Z. / Khatib, F. / DiMaio, F. / Thompson, J. / Baker, D. / Pichova, I. / Jaskolski, M. #3: ジャーナル: Nature / 年: 1989 タイトル: Crystal structure of a retroviral protease proves relationship to aspartic protease family. 著者: Miller, M. / Jaskolski, M. / Rao, J.K. / Leis, J. / Wlodawer, A. #4:  ジャーナル: Science / 年: 1989 ジャーナル: Science / 年: 1989タイトル: Conserved folding in retroviral proteases: crystal structure of a synthetic HIV-1 protease. 著者: Wlodawer, A. / Miller, M. / Jaskolski, M. / Sathyanarayana, B.K. / Baldwin, E. / Weber, I.T. / Selk, L.M. / Clawson, L. / Schneider, J. / Kent, S.B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6s1u.cif.gz 6s1u.cif.gz | 105.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6s1u.ent.gz pdb6s1u.ent.gz | 81 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6s1u.json.gz 6s1u.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/s1/6s1u https://data.pdbj.org/pub/pdb/validation_reports/s1/6s1u ftp://data.pdbj.org/pub/pdb/validation_reports/s1/6s1u ftp://data.pdbj.org/pub/pdb/validation_reports/s1/6s1u | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  6s1vC  6s1wC  3sqfS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 実験データセット #1 | データ参照:  10.18150/repod.0005795 / データの種類: diffraction image data / Metadata reference: 10.18150/repod.0005795 10.18150/repod.0005795 / データの種類: diffraction image data / Metadata reference: 10.18150/repod.0005795 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 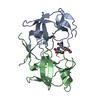
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 0 / Ens-ID: 1 / Beg auth comp-ID: TRP / Beg label comp-ID: TRP / End auth comp-ID: PRO / End label comp-ID: PRO / Refine code: 0 / Auth seq-ID: 1 - 108 / Label seq-ID: 1 - 108
|
- 要素
要素
| #1: タンパク質 | 分子量: 12899.834 Da / 分子数: 2 / 変異: C7A, D26N, C106A; ENGINEERED MUTATION / 由来タイプ: 組換発現 詳細: Gaps in the sequence indicate residues that were not modeled because of poor electron density. 由来: (組換発現)   Mason-Pfizer monkey virus (ウイルス) Mason-Pfizer monkey virus (ウイルス)遺伝子: gag-pro-pol / プラスミド: pBPS13ATG 発現宿主:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌)参照: UniProt: P07572, dUTP diphosphatase, 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ, 逆転写酵素, ...参照: UniProt: P07572,  dUTP diphosphatase, dUTP diphosphatase,  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ, 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ,  逆転写酵素, 逆転写酵素,  DNAポリメラーゼ, DNAポリメラーゼ,  リボヌクレアーゼH, リボヌクレアーゼH,  転移酵素; リンを含む基を移すもの; 核酸を移すもの, 転移酵素; リンを含む基を移すもの; 核酸を移すもの,  加水分解酵素; エステル加水分解酵素 加水分解酵素; エステル加水分解酵素#2: タンパク質・ペプチド | | 分子量: 886.065 Da / 分子数: 1 / 由来タイプ: 合成 詳細: In the standard definition used by Refmac, the Cgamma atom of the PSA residue is labeled as CA. In the PDB Validation Report this label is interpreted as Calpha causing geometrical alerts. ...詳細: In the standard definition used by Refmac, the Cgamma atom of the PSA residue is labeled as CA. In the PDB Validation Report this label is interpreted as Calpha causing geometrical alerts. These alerts are false and should be ignored. 由来: (合成) synthetic construct (人工物) #3: 水 | ChemComp-HOH / |  水 水研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.365 Å3/Da / 溶媒含有率: 47.99 % / 解説: plate |
|---|---|
結晶化 | 温度: 292 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.5 詳細: Protein solution: 6.8 mg/mL protein with 1.2-fold molar excess (relative to dimeric protein) of Pro-0A1-Val-PSA-Ala-Met-Thr (inhibitor), 10 mM Tris buffer pH 7.4; Reservoir solution: 0.1 M ...詳細: Protein solution: 6.8 mg/mL protein with 1.2-fold molar excess (relative to dimeric protein) of Pro-0A1-Val-PSA-Ala-Met-Thr (inhibitor), 10 mM Tris buffer pH 7.4; Reservoir solution: 0.1 M imidazole buffer, 1.2 M sodium acetate; |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  BESSY BESSY  / ビームライン: 14.2 / 波長: 0.91841 Å / ビームライン: 14.2 / 波長: 0.91841 Å |
| 検出器 | タイプ: RAYONIX MX-225 / 検出器: CCD / 日付: 2012年4月22日 |
| 放射 | モノクロメーター: Double Crystal Monochromator, Si-111 crystal プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.91841 Å / 相対比: 1 : 0.91841 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→48.3 Å / Num. obs: 20007 / % possible obs: 99.1 % / 冗長度: 4.02 % / Biso Wilson estimate: 29.7 Å2 / CC1/2: 0.997 / Rmerge(I) obs: 0.108 / Rrim(I) all: 0.124 / Net I/σ(I): 11.49 |
| 反射 シェル | 解像度: 1.9→2.02 Å / 冗長度: 3.67 % / Rmerge(I) obs: 0.98 / Mean I/σ(I) obs: 1.53 / Num. unique obs: 3082 / CC1/2: 0.969 / Rrim(I) all: 1.146 / % possible all: 95.9 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 3sqf, chain A 解像度: 1.9→48.3 Å / Cor.coef. Fo:Fc: 0.96 / Cor.coef. Fo:Fc free: 0.935 / SU B: 8.787 / SU ML: 0.12 / 交差検証法: FREE R-VALUE / ESU R: 0.151 / ESU R Free: 0.144 / 詳細: HYDROGEN ATOMS WERE ADDED AT RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 38.38 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 1.9→48.3 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj


