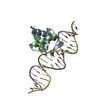
登録情報 データベース : PDB / ID : 6m3dタイトル X-ray crystal structure of tandemly connected engrailed homeodomains (EHD) with R53A mutations and DNA complex DNA (5'-D(*GP*GP*AP*TP*TP*AP*GP*GP*AP*TP*TP*A)-3')DNA (5'-D(*TP*AP*AP*TP*CP*CP*TP*AP*AP*TP*CP*C)-3')Segmentation polarity homeobox protein engrailed,Segmentation polarity homeobox protein engrailed キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Drosophila melanogaster (キイロショウジョウバエ)synthetic construct (人工物) 手法 / / / 解像度 : 1.6 Å データ登録者 Sunami, T. / Hirano, Y. / Tamada, T. / Kono, H. 資金援助 組織 認可番号 国 Japan Society for the Promotion of Science (JSPS) 26840054 Japan Society for the Promotion of Science (JSPS) 26330339 Japan Society for the Promotion of Science (JSPS) JP18H05534
ジャーナル : Acta Crystallogr D Struct Biol / 年 : 2020タイトル : Structural basis for designing an array of engrailed homeodomains.著者 : Sunami, T. / Hirano, Y. / Tamada, T. / Kono, H. 履歴 登録 2020年3月3日 登録サイト / 処理サイト 改定 1.0 2020年9月16日 Provider / タイプ 改定 1.1 2023年11月29日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HOMEODOMAIN FOLD (ホメオボックス) /
HOMEODOMAIN FOLD (ホメオボックス) /  PROTEIN-DNA COMPLEX (デオキシリボ核酸) /
PROTEIN-DNA COMPLEX (デオキシリボ核酸) /  GENOME EDITING (ゲノム編集) / DNA BINDING PROTEIN-DNA complex
GENOME EDITING (ゲノム編集) / DNA BINDING PROTEIN-DNA complex 機能・相同性情報
機能・相同性情報 軸索誘導 / RNA polymerase II transcription regulatory region sequence-specific DNA binding / neuron differentiation / DNA-binding transcription repressor activity, RNA polymerase II-specific /
軸索誘導 / RNA polymerase II transcription regulatory region sequence-specific DNA binding / neuron differentiation / DNA-binding transcription repressor activity, RNA polymerase II-specific /  遺伝子発現の調節 / negative regulation of neuron apoptotic process / sequence-specific DNA binding / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of gene expression / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /
遺伝子発現の調節 / negative regulation of neuron apoptotic process / sequence-specific DNA binding / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of gene expression / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  細胞核
細胞核
 Drosophila melanogaster (キイロショウジョウバエ)
Drosophila melanogaster (キイロショウジョウバエ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 日本, 3件
日本, 3件  引用
引用 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2020
ジャーナル: Acta Crystallogr D Struct Biol / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6m3d.cif.gz
6m3d.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6m3d.ent.gz
pdb6m3d.ent.gz PDB形式
PDB形式 6m3d.json.gz
6m3d.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/m3/6m3d
https://data.pdbj.org/pub/pdb/validation_reports/m3/6m3d ftp://data.pdbj.org/pub/pdb/validation_reports/m3/6m3d
ftp://data.pdbj.org/pub/pdb/validation_reports/m3/6m3d
 リンク
リンク 集合体
集合体
 要素
要素
 Drosophila melanogaster (キイロショウジョウバエ)
Drosophila melanogaster (キイロショウジョウバエ)
 Escherichia coli BL21 (大腸菌) / 参照: UniProt: P02836
Escherichia coli BL21 (大腸菌) / 参照: UniProt: P02836 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Photon Factory
Photon Factory  / ビームライン: BL-17A / 波長: 0.98 Å
/ ビームライン: BL-17A / 波長: 0.98 Å : 0.98 Å / 相対比: 1
: 0.98 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj









































