+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5dpr | ||||||
|---|---|---|---|---|---|---|---|
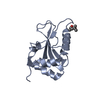
| タイトル | Crystal structure of PLEKHM1 LIR-fused human LC3A_2-121 | ||||||
 要素 要素 | Pleckstrin homology domain-containing family M member 1,Microtubule-associated proteins 1A/1B light chain 3A | ||||||
 キーワード キーワード |  PROTEIN BINDING (タンパク質) / PROTEIN BINDING (タンパク質) /  Autophagy (オートファジー) / Autophagy (オートファジー) /  PLEKHM1 / PLEKHM1 /  Atg8 / LC3 / Atg8 / LC3 /  GABARAP / GABARAP /  chimeric protein (融合タンパク質) chimeric protein (融合タンパク質) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to oxygen-glucose deprivation / late endosome to lysosome transport / autophagosome-lysosome fusion / autolysosome membrane / autophagy of mitochondrion / lysosome localization / cellular response to nitrogen starvation / SMAD protein signal transduction / response to iron(II) ion / positive regulation of ruffle assembly ...cellular response to oxygen-glucose deprivation / late endosome to lysosome transport / autophagosome-lysosome fusion / autolysosome membrane / autophagy of mitochondrion / lysosome localization / cellular response to nitrogen starvation / SMAD protein signal transduction / response to iron(II) ion / positive regulation of ruffle assembly /  オートファゴソーム / オートファゴソーム /  オートファジー / Receptor Mediated Mitophagy / p38MAPK cascade / autophagosome membrane / organelle membrane / autophagosome maturation / オートファジー / Receptor Mediated Mitophagy / p38MAPK cascade / autophagosome membrane / organelle membrane / autophagosome maturation /  autophagosome assembly / autophagosome assembly /  オートファゴソーム / positive regulation of bone resorption / JNK cascade / cellular response to copper ion / PINK1-PRKN Mediated Mitophagy / cellular response to amino acid starvation / cellular response to starvation / オートファゴソーム / positive regulation of bone resorption / JNK cascade / cellular response to copper ion / PINK1-PRKN Mediated Mitophagy / cellular response to amino acid starvation / cellular response to starvation /  オートファジー / response to lead ion / オートファジー / response to lead ion /  phospholipid binding / cellular response to hydrogen peroxide / phospholipid binding / cellular response to hydrogen peroxide /  protein transport / late endosome / late endosome membrane / protein transport / late endosome / late endosome membrane /  microtubule binding / microtubule binding /  微小管 / 微小管 /  リソソーム / intracellular membrane-bounded organelle / glutamatergic synapse / リソソーム / intracellular membrane-bounded organelle / glutamatergic synapse /  ubiquitin protein ligase binding / ubiquitin protein ligase binding /  核小体 / 核小体 /  metal ion binding / metal ion binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | ||||||
 データ登録者 データ登録者 | Ravichandran, A.C. / Suzuki, H. / Dobson, R.C.J. | ||||||
 引用 引用 |  ジャーナル: EMBO Rep. / 年: 2017 ジャーナル: EMBO Rep. / 年: 2017タイトル: Structural and functional analysis of the GABARAP interaction motif (GIM). 著者: Rogov, V.V. / Stolz, A. / Ravichandran, A.C. / Rios-Szwed, D.O. / Suzuki, H. / Kniss, A. / Lohr, F. / Wakatsuki, S. / Dotsch, V. / Dikic, I. / Dobson, R.C. / McEwan, D.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5dpr.cif.gz 5dpr.cif.gz | 305.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5dpr.ent.gz pdb5dpr.ent.gz | 254.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5dpr.json.gz 5dpr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dp/5dpr https://data.pdbj.org/pub/pdb/validation_reports/dp/5dpr ftp://data.pdbj.org/pub/pdb/validation_reports/dp/5dpr ftp://data.pdbj.org/pub/pdb/validation_reports/dp/5dpr | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 15932.104 Da / 分子数: 4 断片: UNP Q9Y4G2 residues 627-638, UNP Q9H492 residues 2-121,UNP Q9Y4G2 residues 627-638, UNP Q9H492 residues 2-121 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: PLEKHM1, KIAA0356, MAP1LC3A / プラスミド: pET30 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PLEKHM1, KIAA0356, MAP1LC3A / プラスミド: pET30 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q9Y4G2, UniProt: Q9H492 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q9Y4G2, UniProt: Q9H492#2: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.59 Å3/Da / 溶媒含有率: 52.46 % |
|---|---|
結晶化 | 温度: 278 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5.5 詳細: 0.2 M ammonium acetate, 0.1 M Bis Tris, pH 5.5, 25% w/v Polyethlene glycol 3,350 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Australian Synchrotron Australian Synchrotron  / ビームライン: MX2 / 波長: 0.9537 Å / ビームライン: MX2 / 波長: 0.9537 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2015年4月9日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9537 Å / 相対比: 1 : 0.9537 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→45.57 Å / Num. obs: 22767 / % possible obs: 100 % / 冗長度: 3.8 % / Net I/σ(I): 7.1 |
| 反射 シェル | 解像度: 2.5→2.5 Å / 冗長度: 3.8 % / Rmerge(I) obs: 0.614 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.5→45.567 Å / SU ML: 0.37 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 28.59 / 立体化学のターゲット値: ML 分子置換 / 解像度: 2.5→45.567 Å / SU ML: 0.37 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 28.59 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→45.567 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj







