登録情報 データベース : PDB / ID : 3v6oタイトル Leptin Receptor-antibody complex (Monoclonal antibody 9F8 fab fragment ...) x 2 Leptin receptor キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)Mus musculus (ハツカネズミ)手法 / / / 解像度 : 1.95 Å データ登録者 Carpenter, B. / Hemsworth, G.R. / Ross, R.J. / Artymiuk, P.J. ジャーナル : Structure / 年 : 2012タイトル : Structure of the human obesity receptor leptin-binding domain reveals the mechanism of leptin antagonism by a monoclonal antibody.著者 : Carpenter, B. / Hemsworth, G.R. / Wu, Z. / Maamra, M. / Strasburger, C.J. / Ross, R.J. / Artymiuk, P.J. 履歴 登録 2011年12月20日 登録サイト / 処理サイト 改定 1.0 2012年3月14日 Provider / タイプ 改定 1.1 2012年4月25日 Group 改定 1.2 2020年7月29日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Structure summary カテゴリ chem_comp / entity ... chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / pdbx_struct_conn_angle / struct_conn / struct_ref_seq / struct_site / struct_site_gen Item _chem_comp.name / _chem_comp.type ... _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end 解説 / Provider / タイプ 改定 1.3 2023年12月27日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Structure summary カテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 / struct_conn Item _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ... _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード IMMUNE SYSTEM (免疫系) / Receptor-antibody complex /
IMMUNE SYSTEM (免疫系) / Receptor-antibody complex /  Cytokine receptor / Antibody Fab fragment / Immunoglobulin fold / Leptin Receptor-antibody complex
Cytokine receptor / Antibody Fab fragment / Immunoglobulin fold / Leptin Receptor-antibody complex 機能・相同性情報
機能・相同性情報 leptin receptor activity / regulation of transport /
leptin receptor activity / regulation of transport /  骨化 / leptin-mediated signaling pathway /
骨化 / leptin-mediated signaling pathway /  regulation of bone remodeling / response to leptin /
regulation of bone remodeling / response to leptin /  regulation of feeding behavior /
regulation of feeding behavior /  有性生殖 / multicellular organism development / energy reserve metabolic process ...
有性生殖 / multicellular organism development / energy reserve metabolic process ... leptin receptor activity / regulation of transport /
leptin receptor activity / regulation of transport /  骨化 / leptin-mediated signaling pathway /
骨化 / leptin-mediated signaling pathway /  regulation of bone remodeling / response to leptin /
regulation of bone remodeling / response to leptin /  regulation of feeding behavior /
regulation of feeding behavior /  有性生殖 / multicellular organism development / energy reserve metabolic process / Signaling by Leptin /
有性生殖 / multicellular organism development / energy reserve metabolic process / Signaling by Leptin /  cytokine receptor activity / glycogen metabolic process /
cytokine receptor activity / glycogen metabolic process /  cytokine binding / transport across blood-brain barrier / T cell differentiation / glial cell proliferation / negative regulation of gluconeogenesis /
cytokine binding / transport across blood-brain barrier / T cell differentiation / glial cell proliferation / negative regulation of gluconeogenesis /  食作用 /
食作用 /  energy homeostasis / cholesterol metabolic process / negative regulation of autophagy /
energy homeostasis / cholesterol metabolic process / negative regulation of autophagy /  糖新生 / cytokine-mediated signaling pathway / transmembrane signaling receptor activity /
糖新生 / cytokine-mediated signaling pathway / transmembrane signaling receptor activity /  glucose homeostasis / positive regulation of cold-induced thermogenesis / basolateral plasma membrane /
glucose homeostasis / positive regulation of cold-induced thermogenesis / basolateral plasma membrane /  血管新生 / cell surface receptor signaling pathway /
血管新生 / cell surface receptor signaling pathway /  receptor complex / positive regulation of protein phosphorylation / external side of plasma membrane / extracellular region / identical protein binding
receptor complex / positive regulation of protein phosphorylation / external side of plasma membrane / extracellular region / identical protein binding
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.95 Å
分子置換 / 解像度: 1.95 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Structure / 年: 2012
ジャーナル: Structure / 年: 2012 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3v6o.cif.gz
3v6o.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3v6o.ent.gz
pdb3v6o.ent.gz PDB形式
PDB形式 3v6o.json.gz
3v6o.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/v6/3v6o
https://data.pdbj.org/pub/pdb/validation_reports/v6/3v6o ftp://data.pdbj.org/pub/pdb/validation_reports/v6/3v6o
ftp://data.pdbj.org/pub/pdb/validation_reports/v6/3v6o リンク
リンク 集合体
集合体


 要素
要素
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ)
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ)

 / LEP-R / HuB219 / OB receptor / OB-R
/ LEP-R / HuB219 / OB receptor / OB-R
 Homo sapiens (ヒト) / 遺伝子: DB, LEPR, OBR / プラスミド: pET21a(+) / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: DB, LEPR, OBR / プラスミド: pET21a(+) / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)-RIPL / 参照: UniProt: P48357
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)-RIPL / 参照: UniProt: P48357 N-アセチルグルコサミン
N-アセチルグルコサミン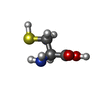








 システイン
システイン エチレングリコール
エチレングリコール 酢酸塩
酢酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I02 / 波長: 0.9796 Å
/ ビームライン: I02 / 波長: 0.9796 Å : 0.9796 Å / 相対比: 1
: 0.9796 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 1.95→49 Å / Cor.coef. Fo:Fc: 0.964 / Cor.coef. Fo:Fc free: 0.947 / Occupancy max: 1 / Occupancy min: 1 / SU B: 6.891 / SU ML: 0.088 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.12 / ESU R Free: 0.122 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
分子置換 / 解像度: 1.95→49 Å / Cor.coef. Fo:Fc: 0.964 / Cor.coef. Fo:Fc free: 0.947 / Occupancy max: 1 / Occupancy min: 1 / SU B: 6.891 / SU ML: 0.088 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.12 / ESU R Free: 0.122 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj








