+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3dar | ||||||
|---|---|---|---|---|---|---|---|
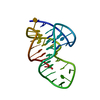
| タイトル | Crystal structure of D2 domain from human FGFR2 | ||||||
 要素 要素 | Fibroblast growth factor receptor 2 | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / IMMUNOGLOBULIN FOLD / ATP-binding / TRANSFERASE (転移酵素) / IMMUNOGLOBULIN FOLD / ATP-binding /  Craniosynostosis (頭蓋骨縫合早期癒合症) / Disease mutation / Craniosynostosis (頭蓋骨縫合早期癒合症) / Disease mutation /  Ectodermal dysplasia / Ectodermal dysplasia /  Glycoprotein (糖タンパク質) / Heparin-binding / Glycoprotein (糖タンパク質) / Heparin-binding /  Immunoglobulin domain (免疫グロブリンフォールド) / Immunoglobulin domain (免疫グロブリンフォールド) /  Kinase (キナーゼ) / Lacrimo-auriculo-dento-digital syndrome / Kinase (キナーゼ) / Lacrimo-auriculo-dento-digital syndrome /  Membrane (生体膜) / Nucleotide-binding / Membrane (生体膜) / Nucleotide-binding /  Phosphoprotein / Receptor / Phosphoprotein / Receptor /  Secreted (分泌) / Secreted (分泌) /  Transmembrane (膜貫通型タンパク質) / Tyrosine-protein kinase Transmembrane (膜貫通型タンパク質) / Tyrosine-protein kinase | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Signaling by FGFR2 amplification mutants / Signaling by FGFR2 fusions / fibroblast growth factor receptor signaling pathway involved in negative regulation of apoptotic process in bone marrow cell / fibroblast growth factor receptor signaling pathway involved in hemopoiesis / fibroblast growth factor receptor signaling pathway involved in positive regulation of cell proliferation in bone marrow / fibroblast growth factor receptor signaling pathway involved in mammary gland specification / lateral sprouting from an epithelium / mammary gland bud formation / branch elongation involved in salivary gland morphogenesis / mesenchymal cell differentiation involved in lung development ...Signaling by FGFR2 amplification mutants / Signaling by FGFR2 fusions / fibroblast growth factor receptor signaling pathway involved in negative regulation of apoptotic process in bone marrow cell / fibroblast growth factor receptor signaling pathway involved in hemopoiesis / fibroblast growth factor receptor signaling pathway involved in positive regulation of cell proliferation in bone marrow / fibroblast growth factor receptor signaling pathway involved in mammary gland specification / lateral sprouting from an epithelium / mammary gland bud formation / branch elongation involved in salivary gland morphogenesis / mesenchymal cell differentiation involved in lung development / lacrimal gland development / otic vesicle formation / prostate gland morphogenesis / regulation of smooth muscle cell differentiation / orbitofrontal cortex development / regulation of morphogenesis of a branching structure / squamous basal epithelial stem cell differentiation involved in prostate gland acinus development / branching morphogenesis of a nerve / embryonic organ morphogenesis / endochondral bone growth / morphogenesis of embryonic epithelium / epidermis morphogenesis / bud elongation involved in lung branching / positive regulation of epithelial cell proliferation involved in lung morphogenesis / pyramidal neuron development / membranous septum morphogenesis / reproductive structure development / limb bud formation / lung lobe morphogenesis / gland morphogenesis / fibroblast growth factor receptor signaling pathway involved in orbitofrontal cortex development / ventricular zone neuroblast division / embryonic digestive tract morphogenesis / epithelial cell proliferation involved in salivary gland morphogenesis / mesenchymal cell differentiation / branching involved in prostate gland morphogenesis / mesenchymal cell proliferation involved in lung development / branching involved in labyrinthine layer morphogenesis / FGFR2b ligand binding and activation / FGFR2c ligand binding and activation / Activated point mutants of FGFR2 / regulation of osteoblast proliferation / Phospholipase C-mediated cascade; FGFR2 /  fibroblast growth factor receptor activity / branching involved in salivary gland morphogenesis / embryonic pattern specification / outflow tract septum morphogenesis / positive regulation of phospholipase activity / lung-associated mesenchyme development / digestive tract development / mesodermal cell differentiation / regulation of smoothened signaling pathway / embryonic cranial skeleton morphogenesis / bone morphogenesis / fibroblast growth factor receptor activity / branching involved in salivary gland morphogenesis / embryonic pattern specification / outflow tract septum morphogenesis / positive regulation of phospholipase activity / lung-associated mesenchyme development / digestive tract development / mesodermal cell differentiation / regulation of smoothened signaling pathway / embryonic cranial skeleton morphogenesis / bone morphogenesis /  skeletal system morphogenesis / skeletal system morphogenesis /  歯の発生 / ureteric bud development / positive regulation of mesenchymal cell proliferation / regulation of osteoblast differentiation / ventricular cardiac muscle tissue morphogenesis / Signaling by FGFR2 IIIa TM / inner ear morphogenesis / organ growth / midbrain development / hair follicle morphogenesis / lung alveolus development / prostate epithelial cord arborization involved in prostate glandular acinus morphogenesis / 歯の発生 / ureteric bud development / positive regulation of mesenchymal cell proliferation / regulation of osteoblast differentiation / ventricular cardiac muscle tissue morphogenesis / Signaling by FGFR2 IIIa TM / inner ear morphogenesis / organ growth / midbrain development / hair follicle morphogenesis / lung alveolus development / prostate epithelial cord arborization involved in prostate glandular acinus morphogenesis /  fibroblast growth factor binding / PI-3K cascade:FGFR2 / fibroblast growth factor binding / PI-3K cascade:FGFR2 /  bone mineralization / prostate epithelial cord elongation / positive regulation of cell division / bone mineralization / prostate epithelial cord elongation / positive regulation of cell division /  excitatory synapse / positive regulation of Wnt signaling pathway / PI3K Cascade / negative regulation of keratinocyte proliferation / excitatory synapse / positive regulation of Wnt signaling pathway / PI3K Cascade / negative regulation of keratinocyte proliferation /  上皮間葉転換 / fibroblast growth factor receptor signaling pathway / embryonic organ development / 上皮間葉転換 / fibroblast growth factor receptor signaling pathway / embryonic organ development /  細胞分化 / positive regulation of cell cycle / SHC-mediated cascade:FGFR2 / cellular response to retinoic acid / positive regulation of cardiac muscle cell proliferation / FRS-mediated FGFR2 signaling / positive regulation of vascular associated smooth muscle cell proliferation / cellular response to transforming growth factor beta stimulus / Signaling by FGFR2 in disease / epithelial cell differentiation / 細胞分化 / positive regulation of cell cycle / SHC-mediated cascade:FGFR2 / cellular response to retinoic acid / positive regulation of cardiac muscle cell proliferation / FRS-mediated FGFR2 signaling / positive regulation of vascular associated smooth muscle cell proliferation / cellular response to transforming growth factor beta stimulus / Signaling by FGFR2 in disease / epithelial cell differentiation /  軸索誘導 / regulation of ERK1 and ERK2 cascade / post-embryonic development / positive regulation of epithelial cell proliferation / Negative regulation of FGFR2 signaling / animal organ morphogenesis / lung development / 軸索誘導 / regulation of ERK1 and ERK2 cascade / post-embryonic development / positive regulation of epithelial cell proliferation / Negative regulation of FGFR2 signaling / animal organ morphogenesis / lung development /  骨 / 骨 /  受容体型チロシンキナーゼ / peptidyl-tyrosine phosphorylation / 受容体型チロシンキナーゼ / peptidyl-tyrosine phosphorylation /  cell surface receptor protein tyrosine kinase signaling pathway cell surface receptor protein tyrosine kinase signaling pathway類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Brown, A. / Blundell, T.L. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Crystal structure of the FGFR2 D2 domain 著者: Brown, A. / Blundell, T.L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
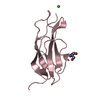
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3dar.cif.gz 3dar.cif.gz | 56.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3dar.ent.gz pdb3dar.ent.gz | 39.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3dar.json.gz 3dar.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/da/3dar https://data.pdbj.org/pub/pdb/validation_reports/da/3dar ftp://data.pdbj.org/pub/pdb/validation_reports/da/3dar ftp://data.pdbj.org/pub/pdb/validation_reports/da/3dar | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1e0oS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Ens-ID: 1 / Beg auth comp-ID: LYS / Beg label comp-ID: LYS / End auth comp-ID: VAL / End label comp-ID: VAL / Auth seq-ID: 151 - 249 / Label seq-ID: 7 - 105
NCS oper:
|
- 要素
要素
| #1: タンパク質 |  / FGFR-2 / Keratinocyte growth factor receptor 2 / CD332 antigen / FGFR-2 / Keratinocyte growth factor receptor 2 / CD332 antigen分子量: 12183.853 Da / 分子数: 2 / 断片: D2 domain, Ig-like C2-type 2, UNP residues 146-249 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: FGFR2, BEK, KGFR, KSAM / プラスミド: pBAT4 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: FGFR2, BEK, KGFR, KSAM / プラスミド: pBAT4 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): Rosetta2 Escherichia coli (大腸菌) / 株 (発現宿主): Rosetta2参照: UniProt: P21802,  受容体型チロシンキナーゼ 受容体型チロシンキナーゼ#2: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.89 Å3/Da / 溶媒含有率: 57.4 % |
|---|---|
結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 詳細: 0.2 M sodium acetate, 0.1 M Tris-HCl, 30% PEG 4000, VAPOR DIFFUSION, HANGING DROP, temperature 291.0K, pH 8.5 |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID29 / 波長: 0.9763 Å / ビームライン: ID29 / 波長: 0.9763 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2006年5月6日 / 詳細: Mirrors | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: SI(311) MONOCHROMATOR / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.9763 Å / 相対比: 1 : 0.9763 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.2→85.9 Å / Num. all: 14756 / Num. obs: 14667 / % possible obs: 98.8 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 2 / 冗長度: 7.3 % / Biso Wilson estimate: 44.3 Å2 / Rmerge(I) obs: 0.08 / Rsym value: 0.08 / Net I/σ(I): 6.7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
位相決定 | 手法:  分子置換 分子置換 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR | Model details: Phaser MODE: MR_AUTO
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PBD Entry 1E0O, chain B, residues 149-249 解像度: 2.2→33.917 Å / FOM work R set: 0.787 / Isotropic thermal model: isotropic / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: mlhl
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | Bsol: 58.254 Å2 / ksol: 0.352 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 78.28 Å2 / Biso mean: 38.91 Å2 / Biso min: 20.58 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→33.917 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: chain B & A / Rms dev position: 0.035 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





