+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2y4z | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of the amino-terminal capsid restriction escape mutation N- MLV L10W | ||||||
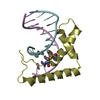
 要素 要素 | CAPSID PROTEIN P30 カプシド カプシド | ||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / RESTRICTION VIRAL PROTEIN (ウイルスタンパク質) / RESTRICTION | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding via host ESCRT complex / host multivesicular body / viral nucleocapsid / structural constituent of virion / host cell plasma membrane /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  生体膜 生体膜類似検索 - 分子機能 | ||||||
| 生物種 |   MURINE LEUKEMIA VIRUS (ネズミ白血病ウイルス) MURINE LEUKEMIA VIRUS (ネズミ白血病ウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.001 Å 分子置換 / 解像度: 2.001 Å | ||||||
 データ登録者 データ登録者 | Goldstone, D.C. / Holden-Dye, K. / Ohkura, S. / Stoye, J.P. / Taylor, I.A. | ||||||
 引用 引用 |  ジャーナル: Plos Pathog. / 年: 2011 ジャーナル: Plos Pathog. / 年: 2011タイトル: Novel Escape Mutants Suggest an Extensive Trim5Alpha Binding Site Spanning the Entire Outer Surface of the Murine Leukemia Virus Capsid Protein. 著者: Ohkura, S. / Goldstone, D.C. / Yap, M.W. / Holden-Dye, K. / Taylor, I.A. / Stoye, J.P. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: PROVIDED BY DEPOSITOR | ||||||
| Remark 700 | SHEET DETERMINATION METHOD: PROVIDED BY DEPOSITOR |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2y4z.cif.gz 2y4z.cif.gz | 70.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2y4z.ent.gz pdb2y4z.ent.gz | 52.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2y4z.json.gz 2y4z.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/y4/2y4z https://data.pdbj.org/pub/pdb/validation_reports/y4/2y4z ftp://data.pdbj.org/pub/pdb/validation_reports/y4/2y4z ftp://data.pdbj.org/pub/pdb/validation_reports/y4/2y4z | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1u7kS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| 単位格子 |
| |||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 |  カプシド / CA カプシド / CA分子量: 16105.835 Da / 分子数: 1 / 断片: N-TERMINAL DOMAIN, RESIDUES 215-346 / 変異: YES / 由来タイプ: 組換発現 由来: (組換発現)   MURINE LEUKEMIA VIRUS (ネズミ白血病ウイルス) MURINE LEUKEMIA VIRUS (ネズミ白血病ウイルス)発現宿主:   ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P03336 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: P03336 | ||
|---|---|---|---|
| #2: 化合物 | ChemComp-GOL /  グリセリン グリセリン | ||
| #3: 水 | ChemComp-HOH /  水 水 | ||
| 構成要素の詳細 | ENGINEERED| 配列の詳細 | THE MUTATION MENTIONED IN REMARK 400 AND THE SEQADV RECORD CORRESPOND | |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.06 Å3/Da / 溶媒含有率: 40 % / 解説: NONE |
|---|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / 波長: 1.5418 回転陽極 / 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU RAXIS IV / 検出器: IMAGE PLATE |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2→25 Å / Num. obs: 9015 / % possible obs: 99.6 % / Observed criterion σ(I): 2 / 冗長度: 3.4 % / Biso Wilson estimate: 29.07 Å2 / Rmerge(I) obs: 0.04 / Net I/σ(I): 25.1 |
| 反射 シェル | 解像度: 2→2.07 Å / 冗長度: 2.9 % / Rmerge(I) obs: 0.18 / Mean I/σ(I) obs: 6.3 / % possible all: 97 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1U7K (CHAIN A) 解像度: 2.001→24.681 Å / SU ML: 0.25 / σ(F): 1.34 / 位相誤差: 22.93 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 39.319 Å2 / ksol: 0.364 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 36.3 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.001→24.681 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


