+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2prl | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | The structures of apo- and inhibitor bound human dihydroorotate dehydrogenase reveal conformational flexibility within the inhibitor binding site | ||||||
 要素 要素 | Dihydroorotate dehydrogenase, mitochondrial | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / protein inhibitor complex OXIDOREDUCTASE (酸化還元酵素) / protein inhibitor complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pyrimidine ribonucleotide biosynthetic process /  dihydroorotate dehydrogenase (quinone) activity / dihydroorotate dehydrogenase (quinone) activity /  ジヒドロオロト酸デヒドロゲナーゼ (キノン) / ジヒドロオロト酸デヒドロゲナーゼ (キノン) /  dihydroorotate dehydrogenase activity / dihydroorotate dehydrogenase activity /  dihydroorotase activity / dihydroorotase activity /  Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process / Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process /  ミトコンドリア内膜 ...pyrimidine ribonucleotide biosynthetic process / ミトコンドリア内膜 ...pyrimidine ribonucleotide biosynthetic process /  dihydroorotate dehydrogenase (quinone) activity / dihydroorotate dehydrogenase (quinone) activity /  ジヒドロオロト酸デヒドロゲナーゼ (キノン) / ジヒドロオロト酸デヒドロゲナーゼ (キノン) /  dihydroorotate dehydrogenase activity / dihydroorotate dehydrogenase activity /  dihydroorotase activity / dihydroorotase activity /  Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process / Pyrimidine biosynthesis / UDP biosynthetic process / 'de novo' UMP biosynthetic process / 'de novo' pyrimidine nucleobase biosynthetic process /  ミトコンドリア内膜 / ミトコンドリア内膜 /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å | ||||||
 データ登録者 データ登録者 | Walse, B. / Dufe, V.T. / Al-Karadaghi, S. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2008 ジャーナル: Biochemistry / 年: 2008タイトル: The structures of human dihydroorotate dehydrogenase with and without inhibitor reveal conformational flexibility in the inhibitor and substrate binding sites 著者: Walse, B. / Dufe, V.T. / Svensson, B. / Fritzson, I. / Dahlberg, L. / Khairoullina, A. / Wellmar, U. / Al-Karadaghi, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2prl.cif.gz 2prl.cif.gz | 95.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2prl.ent.gz pdb2prl.ent.gz | 70.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2prl.json.gz 2prl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pr/2prl https://data.pdbj.org/pub/pdb/validation_reports/pr/2prl ftp://data.pdbj.org/pub/pdb/validation_reports/pr/2prl ftp://data.pdbj.org/pub/pdb/validation_reports/pr/2prl | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | biological unit is a monomer |
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  / E.C.1.3.3.1 / (Dihydroorotate oxidase / DHOdehase / E.C.1.3.3.1 / (Dihydroorotate oxidase / DHOdehase分子量: 39856.535 Da / 分子数: 1 / 変異: N-terminus truncated / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 細胞内の位置: mitochondria Homo sapiens (ヒト) / 細胞内の位置: mitochondria ミトコンドリア / 遺伝子: DHODH / プラスミド: pET-19b / 発現宿主: ミトコンドリア / 遺伝子: DHODH / プラスミド: pET-19b / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q02127, EC: 1.3.99.11 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q02127, EC: 1.3.99.11 |
|---|
-非ポリマー , 7種, 238分子 


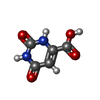
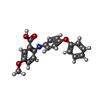
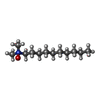







| #2: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩 | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| #3: 化合物 |  酢酸塩 酢酸塩#4: 化合物 | ChemComp-FMN / |  フラビンモノヌクレオチド フラビンモノヌクレオチド#5: 化合物 | ChemComp-ORO / |  オロト酸 オロト酸#6: 化合物 | ChemComp-R2C / | #7: 化合物 | ChemComp-DDQ / | #8: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.61 Å3/Da / 溶媒含有率: 65.94 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.8 詳細: DROPS WERE FORMED BY MIXING EQUAL AMOUNTS OF 18-24 MG/ML PROTEIN IN 100 MM HEPES PH 7.0, 400 MM NACL, 30% GLYCEROL, 1 MM EDTA AND 10 MM N,N- DIMETHYLUNDECYLAMIN-N-OXIDE (C11DAO) WITH A ...詳細: DROPS WERE FORMED BY MIXING EQUAL AMOUNTS OF 18-24 MG/ML PROTEIN IN 100 MM HEPES PH 7.0, 400 MM NACL, 30% GLYCEROL, 1 MM EDTA AND 10 MM N,N- DIMETHYLUNDECYLAMIN-N-OXIDE (C11DAO) WITH A PRECIPITANT SOLUTION OF 0.1 M ACETATE PH 4.8 40 MM C11DAO, 20.8 MM N,-DIMETHYLDECYLAMINE-N-OXIDE (DDAO), 2 MM DIHYDROOROTATE (DHO) THE HANGING DROPS WERE INCUBATED AGAINST 0.5 ML RESERVOIR OF 0.1 M ACETATE PH 4.8, 1.6-2.2 M AMMONIUM SULFATE AND 30% GLYCEROL., VAPOR DIFFUSION, HANGING DROP, temperature 293K |
-データ収集
| 回折 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  MAX II MAX II  / ビームライン: I711 / 波長: 1.092 Å / ビームライン: I711 / 波長: 1.092 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: MAR scanner 345 mm plate / 検出器: IMAGE PLATE / 日付: 2004年4月27日 / 詳細: mirrors | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 1.092 Å / 相対比: 1 : 1.092 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.1→18.83 Å / Num. obs: 33570 / % possible obs: 98 % / Observed criterion σ(I): -3 / Biso Wilson estimate: 22.487 Å2 / Rmerge(I) obs: 0.114 / Net I/σ(I): 12.17 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1,2
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 1d3g 解像度: 2.1→18.83 Å / Cor.coef. Fo:Fc: 0.943 / Cor.coef. Fo:Fc free: 0.923 / SU B: 5.642 / SU ML: 0.08 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.145 / ESU R Free: 0.13 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 10.597 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→18.83 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.1→2.154 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj






