+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2i0i | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | X-ray crystal structure of Sap97 PDZ3 bound to the C-terminal peptide of HPV18 E6 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PEPTIDE BINDING PROTEIN / Sap97 PDZ3 / HPV18 E6 /  tumor suppressor (がん抑制遺伝子) / cervical carcinoma tumor suppressor (がん抑制遺伝子) / cervical carcinoma | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tissue morphogenesis / regulation of protein localization to synapse / regulation of potassium ion import /  L27 domain binding / regulation of potassium ion export across plasma membrane / MPP7-DLG1-LIN7 complex / membrane raft organization / hard palate development / establishment of centrosome localization / negative regulation of p38MAPK cascade ...tissue morphogenesis / regulation of protein localization to synapse / regulation of potassium ion import / L27 domain binding / regulation of potassium ion export across plasma membrane / MPP7-DLG1-LIN7 complex / membrane raft organization / hard palate development / establishment of centrosome localization / negative regulation of p38MAPK cascade ...tissue morphogenesis / regulation of protein localization to synapse / regulation of potassium ion import /  L27 domain binding / regulation of potassium ion export across plasma membrane / MPP7-DLG1-LIN7 complex / membrane raft organization / hard palate development / establishment of centrosome localization / negative regulation of p38MAPK cascade / cortical microtubule organization / embryonic skeletal system morphogenesis / astral microtubule organization / structural constituent of postsynaptic density / lateral loop / reproductive structure development / myelin sheath abaxonal region / immunological synapse formation / L27 domain binding / regulation of potassium ion export across plasma membrane / MPP7-DLG1-LIN7 complex / membrane raft organization / hard palate development / establishment of centrosome localization / negative regulation of p38MAPK cascade / cortical microtubule organization / embryonic skeletal system morphogenesis / astral microtubule organization / structural constituent of postsynaptic density / lateral loop / reproductive structure development / myelin sheath abaxonal region / immunological synapse formation /  蠕動 / cell projection membrane / smooth muscle tissue development / paranode region of axon / bicellular tight junction assembly / Trafficking of AMPA receptors / positive regulation of potassium ion transport / 蠕動 / cell projection membrane / smooth muscle tissue development / paranode region of axon / bicellular tight junction assembly / Trafficking of AMPA receptors / positive regulation of potassium ion transport /  ランヴィエの絞輪 / Activation of Ca-permeable Kainate Receptor / regulation of ventricular cardiac muscle cell action potential / protein-containing complex localization / establishment or maintenance of epithelial cell apical/basal polarity / amyloid precursor protein metabolic process / Synaptic adhesion-like molecules / RAF/MAP kinase cascade / neurotransmitter receptor localization to postsynaptic specialization membrane / lens development in camera-type eye / endothelial cell proliferation / ureteric bud development / ランヴィエの絞輪 / Activation of Ca-permeable Kainate Receptor / regulation of ventricular cardiac muscle cell action potential / protein-containing complex localization / establishment or maintenance of epithelial cell apical/basal polarity / amyloid precursor protein metabolic process / Synaptic adhesion-like molecules / RAF/MAP kinase cascade / neurotransmitter receptor localization to postsynaptic specialization membrane / lens development in camera-type eye / endothelial cell proliferation / ureteric bud development /  regulation of myelination / cortical actin cytoskeleton organization / branching involved in ureteric bud morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / positive regulation of actin filament polymerization / receptor clustering / regulation of myelination / cortical actin cytoskeleton organization / branching involved in ureteric bud morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / positive regulation of actin filament polymerization / receptor clustering /  微絨毛 / 微絨毛 /  kinesin binding / kinesin binding /  免疫シナプス / 免疫シナプス /  基底膜 / lateral plasma membrane / potassium channel regulator activity / Unblocking of NMDA receptors, glutamate binding and activation / bicellular tight junction / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / 基底膜 / lateral plasma membrane / potassium channel regulator activity / Unblocking of NMDA receptors, glutamate binding and activation / bicellular tight junction / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  phosphatase binding / phosphatase binding /  postsynaptic density, intracellular component / T cell proliferation / negative regulation of T cell proliferation / cellular response to brain-derived neurotrophic factor stimulus / postsynaptic density, intracellular component / T cell proliferation / negative regulation of T cell proliferation / cellular response to brain-derived neurotrophic factor stimulus /  横行小管 / 横行小管 /  ionotropic glutamate receptor binding / ionotropic glutamate receptor binding /  T細胞 / actin filament polymerization / basal plasma membrane / phosphatidylinositol 3-kinase/protein kinase B signal transduction / T細胞 / actin filament polymerization / basal plasma membrane / phosphatidylinositol 3-kinase/protein kinase B signal transduction /  synaptic membrane / synaptic membrane /  regulation of membrane potential / protein localization to plasma membrane / regulation of membrane potential / protein localization to plasma membrane /  PDZ domain binding / actin filament organization / positive regulation of protein localization to plasma membrane / postsynaptic density membrane / PDZ domain binding / actin filament organization / positive regulation of protein localization to plasma membrane / postsynaptic density membrane /  neuromuscular junction / neuromuscular junction /  protein localization / negative regulation of ERK1 and ERK2 cascade / cytoplasmic side of plasma membrane / cerebral cortex development / synaptic vesicle membrane / protein localization / negative regulation of ERK1 and ERK2 cascade / cytoplasmic side of plasma membrane / cerebral cortex development / synaptic vesicle membrane /  細胞接着 / 細胞接着 /  regulation of protein localization / negative regulation of epithelial cell proliferation / cell-cell junction / regulation of protein localization / negative regulation of epithelial cell proliferation / cell-cell junction /  細胞結合 / 細胞結合 /  presynaptic membrane / regulation of cell shape / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / chemical synaptic transmission / basolateral plasma membrane / presynaptic membrane / regulation of cell shape / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / chemical synaptic transmission / basolateral plasma membrane /  postsynaptic membrane / symbiont-mediated perturbation of host ubiquitin-like protein modification / postsynaptic membrane / symbiont-mediated perturbation of host ubiquitin-like protein modification /  微小管 / cell population proliferation / transmembrane transporter binding / host cell cytoplasm / 微小管 / cell population proliferation / transmembrane transporter binding / host cell cytoplasm /  postsynaptic density / molecular adaptor activity / postsynaptic density / molecular adaptor activity /  細胞接着 / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / neuron projection / 細胞接着 / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / neuron projection /  脂質ラフト / apical plasma membrane / virus-mediated perturbation of host defense response 脂質ラフト / apical plasma membrane / virus-mediated perturbation of host defense response類似検索 - 分子機能 | ||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Chen, X.S. / Zhang, Y. / Dasgupta, J. / Banks, L. / Thomas, M. | ||||||
 引用 引用 |  ジャーナル: J.Virol. / 年: 2007 ジャーナル: J.Virol. / 年: 2007タイトル: Structures of a Human Papillomavirus (HPV) E6 Polypeptide Bound to MAGUK Proteins: Mechanisms of Targeting Tumor Suppressors by a High-Risk HPV Oncoprotein. 著者: Zhang, Y. / Dasgupta, J. / Ma, R.Z. / Banks, L. / Thomas, M. / Chen, X.S. #1: ジャーナル: Oncogene / 年: 2001 タイトル: HPV E6 and MAGUK protein interactions: determination of the molecular basis for specific protein recognition and degradation. 著者: Thomas, M. / Glaunsinger, B. / Pim, D. / Javier, R. / Banks, L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2i0i.cif.gz 2i0i.cif.gz | 59.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2i0i.ent.gz pdb2i0i.ent.gz | 45 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2i0i.json.gz 2i0i.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i0/2i0i https://data.pdbj.org/pub/pdb/validation_reports/i0/2i0i ftp://data.pdbj.org/pub/pdb/validation_reports/i0/2i0i ftp://data.pdbj.org/pub/pdb/validation_reports/i0/2i0i | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 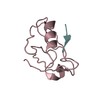
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 8776.911 Da / 分子数: 3 / 断片: PDZ3 / 由来タイプ: 組換発現 / 由来: (組換発現)   Rattus norvegicus (ドブネズミ) / 発現宿主: Rattus norvegicus (ドブネズミ) / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: Q62696 Escherichia coli (大腸菌) / 参照: UniProt: Q62696#2: タンパク質・ペプチド | 分子量: 947.074 Da / 分子数: 3 / 由来タイプ: 合成 詳細: The sequence of this peptide can be found in human papillomavirus type 18 (virus). 参照: UniProt: P06463 #3: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.39 Å3/Da / 溶媒含有率: 48.43 % |
|---|---|
結晶化 | 温度: 291 K / 手法: 蒸気拡散法 / pH: 6.5 詳細: PEG4000, 0.1M MES, pH 6.5, VAPOR DIFFUSION, temperature 291K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU / 波長: 1.54 Å 回転陽極 / タイプ: RIGAKU / 波長: 1.54 Å |
| 検出器 | タイプ: RIGAKU RAXIS IV / 検出器: IMAGE PLATE / 日付: 2006年5月7日 |
| 放射 | モノクロメーター: copper K alpha / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.54 Å / 相対比: 1 : 1.54 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→30 Å / Num. all: 6843 / Num. obs: 6381 / % possible obs: 93.2 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Biso Wilson estimate: 52.9 Å2 |
- 解析
解析
| ソフトウェア | 名称: CNS / バージョン: 1.1 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 2.8→19.97 Å / Rfactor Rfree error: 0.015 / Data cutoff high absF: 1594112.31 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber 分子置換 / 解像度: 2.8→19.97 Å / Rfactor Rfree error: 0.015 / Data cutoff high absF: 1594112.31 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 39.2021 Å2 / ksol: 0.301438 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 50.4 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→19.97 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.97 Å / Rfactor Rfree error: 0.064 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj






