+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2cce | ||||||
|---|---|---|---|---|---|---|---|
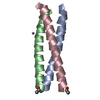
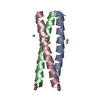
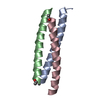
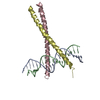
| タイトル | Parallel Configuration of pLI E20S | ||||||
 要素 要素 | General control protein GCN4 | ||||||
 キーワード キーワード | FOUR HELIX BUNDLE / ANTIPARALLEL / PARALLEL / PLI / DNA-BINDING /  NUCLEAR PROTEIN / NUCLEAR PROTEIN /  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  TRANSCRIPTION REGULATION / ACTIVATOR / AMINO-ACID BIOSYNTHESIS TRANSCRIPTION REGULATION / ACTIVATOR / AMINO-ACID BIOSYNTHESIS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein localization to nuclear periphery / Activation of the AP-1 family of transcription factors / response to amino acid starvation /  mediator complex binding / negative regulation of ribosomal protein gene transcription by RNA polymerase II / positive regulation of cellular response to amino acid starvation / nitrogen catabolite activation of transcription from RNA polymerase II promoter / TFIID-class transcription factor complex binding / amino acid biosynthetic process / positive regulation of transcription initiation by RNA polymerase II ...protein localization to nuclear periphery / Activation of the AP-1 family of transcription factors / response to amino acid starvation / mediator complex binding / negative regulation of ribosomal protein gene transcription by RNA polymerase II / positive regulation of cellular response to amino acid starvation / nitrogen catabolite activation of transcription from RNA polymerase II promoter / TFIID-class transcription factor complex binding / amino acid biosynthetic process / positive regulation of transcription initiation by RNA polymerase II ...protein localization to nuclear periphery / Activation of the AP-1 family of transcription factors / response to amino acid starvation /  mediator complex binding / negative regulation of ribosomal protein gene transcription by RNA polymerase II / positive regulation of cellular response to amino acid starvation / nitrogen catabolite activation of transcription from RNA polymerase II promoter / TFIID-class transcription factor complex binding / amino acid biosynthetic process / positive regulation of transcription initiation by RNA polymerase II / positive regulation of RNA polymerase II transcription preinitiation complex assembly / cellular response to amino acid starvation / RNA polymerase II transcription regulator complex / : / DNA-binding transcription activator activity, RNA polymerase II-specific / mediator complex binding / negative regulation of ribosomal protein gene transcription by RNA polymerase II / positive regulation of cellular response to amino acid starvation / nitrogen catabolite activation of transcription from RNA polymerase II promoter / TFIID-class transcription factor complex binding / amino acid biosynthetic process / positive regulation of transcription initiation by RNA polymerase II / positive regulation of RNA polymerase II transcription preinitiation complex assembly / cellular response to amino acid starvation / RNA polymerase II transcription regulator complex / : / DNA-binding transcription activator activity, RNA polymerase II-specific /  transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding / sequence-specific DNA binding / DNA-binding transcription factor activity, RNA polymerase II-specific / intracellular signal transduction / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding / sequence-specific DNA binding / DNA-binding transcription factor activity, RNA polymerase II-specific / intracellular signal transduction / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity /  chromatin binding / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / identical protein binding / chromatin binding / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / identical protein binding /  細胞核 細胞核類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Yadav, M.K. / Leman, L.J. / Price, D.J. / Brooks 3rd, C.L. / Stout, C.D. / Ghadiri, M.R. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2006 ジャーナル: Biochemistry / 年: 2006タイトル: Coiled coils at the edge of configurational heterogeneity. Structural analyses of parallel and antiparallel homotetrameric coiled coils reveal configurational sensitivity to a single ...タイトル: Coiled coils at the edge of configurational heterogeneity. Structural analyses of parallel and antiparallel homotetrameric coiled coils reveal configurational sensitivity to a single solvent-exposed amino acid substitution. 著者: Yadav, M.K. / Leman, L.J. / Price, D.J. / Brooks 3rd, C.L. / Stout, C.D. / Ghadiri, M.R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2cce.cif.gz 2cce.cif.gz | 24 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2cce.ent.gz pdb2cce.ent.gz | 16.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2cce.json.gz 2cce.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cc/2cce https://data.pdbj.org/pub/pdb/validation_reports/cc/2cce ftp://data.pdbj.org/pub/pdb/validation_reports/cc/2cce ftp://data.pdbj.org/pub/pdb/validation_reports/cc/2cce | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4030.802 Da / 分子数: 2 / 断片: RESIDUES 249-281 / 変異: YES / 由来タイプ: 合成 / 由来: (合成)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P03069 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P03069#2: 水 | ChemComp-HOH / |  水 水構成要素の詳細 | ENGINEERED | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.7 Å3/Da / 溶媒含有率: 54 % |
|---|---|
結晶化 | pH: 0 / 詳細: 2M NACL, 10% PEG 6K, pH 0.00 |
-データ収集
| 回折 | 平均測定温度: 180 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL11-1 / 波長: 0.979 / ビームライン: BL11-1 / 波長: 0.979 |
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 2005年7月12日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.979 Å / 相対比: 1 : 0.979 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→50 Å / Num. obs: 7118 / % possible obs: 100 % / Observed criterion σ(I): 2 / 冗長度: 10.9 % / Rmerge(I) obs: 0.06 / Net I/σ(I): 20.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 1.9→56.52 Å / Cor.coef. Fo:Fc: 0.904 / Cor.coef. Fo:Fc free: 0.884 / SU B: 3.802 / SU ML: 0.113 / 交差検証法: THROUGHOUT / ESU R: 0.174 / ESU R Free: 0.166 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 分子置換 / 解像度: 1.9→56.52 Å / Cor.coef. Fo:Fc: 0.904 / Cor.coef. Fo:Fc free: 0.884 / SU B: 3.802 / SU ML: 0.113 / 交差検証法: THROUGHOUT / ESU R: 0.174 / ESU R Free: 0.166 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. HIGH R-FREE BUT CONSISTENT WITH RELATED SEQUENCES IN SAME LATTICE. ELECTRON DENSITY MAPS ARE EXCELLENT.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 44.9 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→56.52 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


































































 PDBj
PDBj

