| 登録情報 | データベース: PDB / ID: 1ujw
|
|---|
| タイトル | Structure of the complex between BtuB and Colicin E3 Receptor binding domain |
|---|
 要素 要素 | - Colicin E3
- Vitamin B12 receptor
|
|---|
 キーワード キーワード | TRANSPORT PROTEIN/HYDROLASE /  beta-barrel (Βバレル) / beta-barrel (Βバレル) /  coiled-coil (コイルドコイル) / TRANSPORT PROTEIN-HYDROLASE COMPLEX coiled-coil (コイルドコイル) / TRANSPORT PROTEIN-HYDROLASE COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of ion transmembrane transporter activity / ABC-type vitamin B12 transporter activity /  extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity / extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity /  porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane ...negative regulation of ion transmembrane transporter activity / ABC-type vitamin B12 transporter activity / porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane ...negative regulation of ion transmembrane transporter activity / ABC-type vitamin B12 transporter activity /  extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity / extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity /  porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane / porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane /  ribosome binding / ribosome binding /  付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - /  endonuclease activity / killing of cells of another organism / transmembrane transporter binding / defense response to bacterium / protein domain specific binding / endonuclease activity / killing of cells of another organism / transmembrane transporter binding / defense response to bacterium / protein domain specific binding /  calcium ion binding / calcium ion binding /  RNA binding / RNA binding /  生体膜類似検索 - 分子機能 生体膜類似検索 - 分子機能 Helix Hairpins / Colicin E3-like ribonuclease domain / Colicin E3-like ribonuclease domain superfamily /  細胞毒性 / TonB-dependent vitamin B12 transporter BtuB / 細胞毒性 / TonB-dependent vitamin B12 transporter BtuB /  Colicin, receptor domain / Coiled-coil receptor-binding R-domain of colicin E2 / TonB-dependent receptor (TBDR) proteins signature 1. / TonB-dependent receptor, beta-barrel domain / Cloacin colicin family ...Helix Hairpins / Colicin E3-like ribonuclease domain / Colicin E3-like ribonuclease domain superfamily / Colicin, receptor domain / Coiled-coil receptor-binding R-domain of colicin E2 / TonB-dependent receptor (TBDR) proteins signature 1. / TonB-dependent receptor, beta-barrel domain / Cloacin colicin family ...Helix Hairpins / Colicin E3-like ribonuclease domain / Colicin E3-like ribonuclease domain superfamily /  細胞毒性 / TonB-dependent vitamin B12 transporter BtuB / 細胞毒性 / TonB-dependent vitamin B12 transporter BtuB /  Colicin, receptor domain / Coiled-coil receptor-binding R-domain of colicin E2 / TonB-dependent receptor (TBDR) proteins signature 1. / TonB-dependent receptor, beta-barrel domain / Cloacin colicin family / Colicin-like bacteriocin tRNase domain / TonB-dependent receptor, plug domain / Pyosin/cloacin translocation domain / Pyosin/cloacin translocation domain superfamily / Colicin, receptor domain / Coiled-coil receptor-binding R-domain of colicin E2 / TonB-dependent receptor (TBDR) proteins signature 1. / TonB-dependent receptor, beta-barrel domain / Cloacin colicin family / Colicin-like bacteriocin tRNase domain / TonB-dependent receptor, plug domain / Pyosin/cloacin translocation domain / Pyosin/cloacin translocation domain superfamily /  Maltoporin; Chain A / TonB box, conserved site / Ferric Hydroxamate Uptake Protein; Chain A, domain 1 / TonB-dependent receptor, conserved site / TonB-dependent receptor (TBDR) proteins signature 2. / TonB-dependent receptor (TBDR) proteins profile. / Vitamin B12 transporter BtuB-like / TonB-dependent receptor-like, beta-barrel / TonB dependent receptor-like, beta-barrel / TonB-dependent receptor, plug domain superfamily / TonB-dependent receptor, plug domain / TonB-dependent receptor-like, beta-barrel domain superfamily / Maltoporin; Chain A / TonB box, conserved site / Ferric Hydroxamate Uptake Protein; Chain A, domain 1 / TonB-dependent receptor, conserved site / TonB-dependent receptor (TBDR) proteins signature 2. / TonB-dependent receptor (TBDR) proteins profile. / Vitamin B12 transporter BtuB-like / TonB-dependent receptor-like, beta-barrel / TonB dependent receptor-like, beta-barrel / TonB-dependent receptor, plug domain superfamily / TonB-dependent receptor, plug domain / TonB-dependent receptor-like, beta-barrel domain superfamily /  TonB-dependent Receptor Plug Domain / Beta Complex / Helix Hairpins / TonB-dependent Receptor Plug Domain / Beta Complex / Helix Hairpins /  Βバレル / Orthogonal Bundle / Mainly Beta / Mainly Alpha類似検索 - ドメイン・相同性 Βバレル / Orthogonal Bundle / Mainly Beta / Mainly Alpha類似検索 - ドメイン・相同性  アセト酢酸 / Chem-GP1 / 3-OXO-PENTADECANOIC ACID / Colicin E3 / Vitamin B12 transporter BtuB類似検索 - 構成要素 アセト酢酸 / Chem-GP1 / 3-OXO-PENTADECANOIC ACID / Colicin E3 / Vitamin B12 transporter BtuB類似検索 - 構成要素 |
|---|
| 生物種 |    Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.75 Å 分子置換 / 解像度: 2.75 Å |
|---|
 データ登録者 データ登録者 | Kurisu, G. / Zakharov, S.D. / Zhalnina, M.V. / Bano, S. / Eroukova, V.Y. / Rokitskaya, T.I. / Antonenko, Y.N. / Wiener, M.C. / Cramer, W.A. |
|---|
 引用 引用 |  ジャーナル: NAT.STRUCT.BIOL. / 年: 2003 ジャーナル: NAT.STRUCT.BIOL. / 年: 2003
タイトル: The structure of BtuB with bound colicin E3 R-domain implies a translocon
著者: Kurisu, G. / Zakharov, S.D. / Zhalnina, M.V. / Bano, S. / Eroukova, V.Y. / Rokitskaya, T.I. / Antonenko, Y.N. / Wiener, M.C. / Cramer, W.A. |
|---|
| 履歴 | | 登録 | 2003年8月12日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2003年11月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Advisory / Version format compliance |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_conn / struct_site / struct_site_gen
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_label_atom_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2023年10月25日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR DETERMINED |
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード beta-barrel (Βバレル) /
beta-barrel (Βバレル) /  coiled-coil (コイルドコイル) / TRANSPORT PROTEIN-HYDROLASE COMPLEX
coiled-coil (コイルドコイル) / TRANSPORT PROTEIN-HYDROLASE COMPLEX 機能・相同性情報
機能・相同性情報 extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity /
extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity /  porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane ...negative regulation of ion transmembrane transporter activity / ABC-type vitamin B12 transporter activity /
porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane ...negative regulation of ion transmembrane transporter activity / ABC-type vitamin B12 transporter activity /  extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity /
extrachromosomal circular DNA / cobalamin transport / siderophore uptake transmembrane transporter activity /  porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane /
porin activity / transmembrane transporter complex / pore complex / monoatomic ion transmembrane transport / cell outer membrane /  ribosome binding /
ribosome binding /  付加脱離酵素(リアーゼ); P-Oリアーゼ類; - /
付加脱離酵素(リアーゼ); P-Oリアーゼ類; - /  endonuclease activity / killing of cells of another organism / transmembrane transporter binding / defense response to bacterium / protein domain specific binding /
endonuclease activity / killing of cells of another organism / transmembrane transporter binding / defense response to bacterium / protein domain specific binding /  calcium ion binding /
calcium ion binding /  RNA binding /
RNA binding /  生体膜
生体膜
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.75 Å
分子置換 / 解像度: 2.75 Å  データ登録者
データ登録者 引用
引用 ジャーナル: NAT.STRUCT.BIOL. / 年: 2003
ジャーナル: NAT.STRUCT.BIOL. / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1ujw.cif.gz
1ujw.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1ujw.ent.gz
pdb1ujw.ent.gz PDB形式
PDB形式 1ujw.json.gz
1ujw.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/uj/1ujw
https://data.pdbj.org/pub/pdb/validation_reports/uj/1ujw ftp://data.pdbj.org/pub/pdb/validation_reports/uj/1ujw
ftp://data.pdbj.org/pub/pdb/validation_reports/uj/1ujw リンク
リンク 集合体
集合体
 要素
要素
 Escherichia coli (大腸菌) / プラスミド: pJC3 / 発現宿主:
Escherichia coli (大腸菌) / プラスミド: pJC3 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P06129
Escherichia coli (大腸菌) / 参照: UniProt: P06129
 Escherichia coli (大腸菌) / プラスミド: pET-216(+) / 発現宿主:
Escherichia coli (大腸菌) / プラスミド: pET-216(+) / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P00646
Escherichia coli (大腸菌) / 参照: UniProt: P00646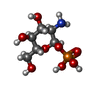








 Lauryldimethylamine oxide
Lauryldimethylamine oxide アセト酢酸
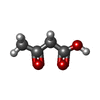
アセト酢酸 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 19-BM
/ ビームライン: 19-BM 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




