| 登録情報 | データベース: PDB / ID: 1qtp
|
|---|
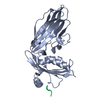
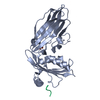
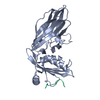
| タイトル | CRYSTAL STRUCTURE OF THE AP-2 CLATHRIN ADAPTOR ALPHA-APPENDAGE |
|---|
 要素 要素 | AP-2 CLATHRIN ADAPTOR ALPHA SUBUNIT (ALPHA-ADAPTIN C) |
|---|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / FOUR-WAVELENGTH MAD / MEMBRANE PROTEIN (膜タンパク質) / FOUR-WAVELENGTH MAD /  SELENOMETHIONINE (セレノメチオニン) SELENOMETHIONINE (セレノメチオニン) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
LDL clearance / WNT5A-dependent internalization of FZD2, FZD5 and ROR2 / WNT5A-dependent internalization of FZD4 / Trafficking of GluR2-containing AMPA receptors / Retrograde neurotrophin signalling / VLDLR internalisation and degradation / Recycling pathway of L1 / AP-2 adaptor complex / postsynaptic neurotransmitter receptor internalization / Cargo recognition for clathrin-mediated endocytosis ...LDL clearance / WNT5A-dependent internalization of FZD2, FZD5 and ROR2 / WNT5A-dependent internalization of FZD4 / Trafficking of GluR2-containing AMPA receptors / Retrograde neurotrophin signalling / VLDLR internalisation and degradation / Recycling pathway of L1 / AP-2 adaptor complex / postsynaptic neurotransmitter receptor internalization / Cargo recognition for clathrin-mediated endocytosis / clathrin adaptor activity /  Clathrin-mediated endocytosis / membrane coat / Clathrin-mediated endocytosis / membrane coat /  clathrin-dependent endocytosis / MHC class II antigen presentation / regulation of hematopoietic stem cell differentiation / protein serine/threonine kinase binding / vesicle-mediated transport / Neutrophil degranulation / clathrin-dependent endocytosis / MHC class II antigen presentation / regulation of hematopoietic stem cell differentiation / protein serine/threonine kinase binding / vesicle-mediated transport / Neutrophil degranulation /  phosphatidylinositol binding / phosphatidylinositol binding /  分泌 / 分泌 /  intracellular protein transport / cytoplasmic side of plasma membrane / intracellular protein transport / cytoplasmic side of plasma membrane /  kinase binding / disordered domain specific binding / kinase binding / disordered domain specific binding /  シナプス小胞 / cytoplasmic vesicle / postsynapse / protein domain specific binding / protein-containing complex binding / シナプス小胞 / cytoplasmic vesicle / postsynapse / protein domain specific binding / protein-containing complex binding /  protein kinase binding類似検索 - 分子機能 protein kinase binding類似検索 - 分子機能 Gamma-adaptin ear (GAE) domain / Clathrin adaptor, alpha-adaptin, appendage, C-terminal subdomain /  Adaptor protein complex AP-2, alpha subunit / Alpha adaptin AP2, C-terminal domain / Adaptor protein complex AP-2, alpha subunit / Alpha adaptin AP2, C-terminal domain /  TATA結合タンパク質 / Coatomer/calthrin adaptor appendage, C-terminal subdomain / Clathrin adaptor, alpha/beta/gamma-adaptin, appendage, Ig-like subdomain / Adaptin C-terminal domain / Adaptin C-terminal domain / Clathrin/coatomer adaptor, adaptin-like, N-terminal ...Gamma-adaptin ear (GAE) domain / Clathrin adaptor, alpha-adaptin, appendage, C-terminal subdomain / TATA結合タンパク質 / Coatomer/calthrin adaptor appendage, C-terminal subdomain / Clathrin adaptor, alpha/beta/gamma-adaptin, appendage, Ig-like subdomain / Adaptin C-terminal domain / Adaptin C-terminal domain / Clathrin/coatomer adaptor, adaptin-like, N-terminal ...Gamma-adaptin ear (GAE) domain / Clathrin adaptor, alpha-adaptin, appendage, C-terminal subdomain /  Adaptor protein complex AP-2, alpha subunit / Alpha adaptin AP2, C-terminal domain / Adaptor protein complex AP-2, alpha subunit / Alpha adaptin AP2, C-terminal domain /  TATA結合タンパク質 / Coatomer/calthrin adaptor appendage, C-terminal subdomain / Clathrin adaptor, alpha/beta/gamma-adaptin, appendage, Ig-like subdomain / Adaptin C-terminal domain / Adaptin C-terminal domain / Clathrin/coatomer adaptor, adaptin-like, N-terminal / Adaptin N terminal region / Clathrin adaptor, appendage, Ig-like subdomain superfamily / TATA結合タンパク質 / Coatomer/calthrin adaptor appendage, C-terminal subdomain / Clathrin adaptor, alpha/beta/gamma-adaptin, appendage, Ig-like subdomain / Adaptin C-terminal domain / Adaptin C-terminal domain / Clathrin/coatomer adaptor, adaptin-like, N-terminal / Adaptin N terminal region / Clathrin adaptor, appendage, Ig-like subdomain superfamily /  TATA結合タンパク質 / TBP domain superfamily / Armadillo-like helical / Armadillo-type fold / TATA結合タンパク質 / TBP domain superfamily / Armadillo-like helical / Armadillo-type fold /  Immunoglobulin-like / Immunoglobulin-like /  サンドイッチ / 2-Layer Sandwich / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 サンドイッチ / 2-Layer Sandwich / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |    Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.6 Å シンクロトロン / 解像度: 1.6 Å |
|---|
 データ登録者 データ登録者 | Traub, L.M. / Downs, M.A. / Westrich, J.L. / Fremont, D.H. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1999 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1999
タイトル: Crystal structure of the alpha appendage of AP-2 reveals a recruitment platform for clathrin-coat assembly.
著者: Traub, L.M. / Downs, M.A. / Westrich, J.L. / Fremont, D.H. |
|---|
| 履歴 | | 登録 | 1999年6月28日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 1999年7月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年12月21日 | Group: Database references / Derived calculations / カテゴリ: database_2 / struct_conn / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード MEMBRANE PROTEIN (膜タンパク質) / FOUR-WAVELENGTH MAD /
MEMBRANE PROTEIN (膜タンパク質) / FOUR-WAVELENGTH MAD /  SELENOMETHIONINE (セレノメチオニン)
SELENOMETHIONINE (セレノメチオニン) 機能・相同性情報
機能・相同性情報 Clathrin-mediated endocytosis / membrane coat /
Clathrin-mediated endocytosis / membrane coat /  clathrin-dependent endocytosis / MHC class II antigen presentation / regulation of hematopoietic stem cell differentiation / protein serine/threonine kinase binding / vesicle-mediated transport / Neutrophil degranulation /
clathrin-dependent endocytosis / MHC class II antigen presentation / regulation of hematopoietic stem cell differentiation / protein serine/threonine kinase binding / vesicle-mediated transport / Neutrophil degranulation /  phosphatidylinositol binding /
phosphatidylinositol binding /  分泌 /
分泌 /  intracellular protein transport / cytoplasmic side of plasma membrane /
intracellular protein transport / cytoplasmic side of plasma membrane /  kinase binding / disordered domain specific binding /
kinase binding / disordered domain specific binding /  シナプス小胞 / cytoplasmic vesicle / postsynapse / protein domain specific binding / protein-containing complex binding /
シナプス小胞 / cytoplasmic vesicle / postsynapse / protein domain specific binding / protein-containing complex binding /  protein kinase binding
protein kinase binding
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン / 解像度: 1.6 Å
シンクロトロン / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1999
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1999 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1qtp.cif.gz
1qtp.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1qtp.ent.gz
pdb1qtp.ent.gz PDB形式
PDB形式 1qtp.json.gz
1qtp.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/qt/1qtp
https://data.pdbj.org/pub/pdb/validation_reports/qt/1qtp ftp://data.pdbj.org/pub/pdb/validation_reports/qt/1qtp
ftp://data.pdbj.org/pub/pdb/validation_reports/qt/1qtp リンク
リンク 集合体
集合体
 要素
要素
 Mus musculus (ハツカネズミ) / 解説: MUS MUSCULUS / プラスミド: PGEX-2T / 発現宿主:
Mus musculus (ハツカネズミ) / 解説: MUS MUSCULUS / プラスミド: PGEX-2T / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL4 / 参照: UniProt: P17427
Escherichia coli (大腸菌) / 株 (発現宿主): BL4 / 参照: UniProt: P17427 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 解析
解析 X-PLOR / 分類: refinement
X-PLOR / 分類: refinement ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj














