登録構造単位
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子 概要 構成要素の詳細
分子量 (理論値) 分子数 合計 (水以外) 49,794 4 ポリマ- 49,136 2 非ポリマー 658 2 水 8,737 485
1
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 99,589 8 ポリマ- 98,272 4 非ポリマー 1,317 4 水 72 4
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_665 -x+1,-y+1,z 1 crystal symmetry operation 3_655 -y+1,x,z 1 crystal symmetry operation 4_565 y,-x+1,z 1
Buried area 11420 Å2 ΔGint -47 kcal/mol Surface area 35440 Å2 手法
2
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 24,897 2 ポリマ- 24,568 1 非ポリマー 329 1 水 18 1
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
3
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
A: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 199,177 16 ポリマ- 196,544 8 非ポリマー 2,634 8 水 144 8
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_665 -x+1,-y+1,z 1 crystal symmetry operation 3_655 -y+1,x,z 1 crystal symmetry operation 4_565 y,-x+1,z 1 crystal symmetry operation 5_554 x+1/2,y+1/2,z-1/2 1 crystal symmetry operation 6_554 -x+1/2,-y+1/2,z-1/2 1 crystal symmetry operation 7_554 -y+1/2,x+1/2,z-1/2 1 crystal symmetry operation 8_554 y+1/2,-x+1/2,z-1/2 1
Buried area 26440 Å2 ΔGint -140 kcal/mol Surface area 66150 Å2 手法
4
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子
B: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 2
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 99,589 8 ポリマ- 98,272 4 非ポリマー 1,317 4 水 72 4
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_555 -x,-y,z 1 crystal symmetry operation 3_555 -y,x,z 1 crystal symmetry operation 4_555 y,-x,z 1
単位格子 Length a, b, c (Å) 94.750, 94.750, 124.420 Angle α, β, γ (deg.) 90.00, 90.00, 90.00 Int Tables number 79 Space group name H-M I4
詳細 The biological assembly is a tetramer generated from the one protmer. There are two distinct protomers in the asymmetric unit labeled A & B. Each protomer associates with its like to form the tetramer. To generate the tetramer apply the following symmetry operations to the asymmetric unit: 1-y,x,z and y,1-x,z and 1-x,1-y,z
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSPORT PROTEIN (運搬体タンパク質) / CNBD / C-LINKER /
TRANSPORT PROTEIN (運搬体タンパク質) / CNBD / C-LINKER /  PACEMAKER (心臓ペースメーカー) / HCN /
PACEMAKER (心臓ペースメーカー) / HCN /  HCN2 / CHANNEL /
HCN2 / CHANNEL /  CYCLIC NUCLEOTIDE / CAP / PKA /
CYCLIC NUCLEOTIDE / CAP / PKA /  cAMP (CAMP) /
cAMP (CAMP) /  ION CHANNEL (イオンチャネル) /
ION CHANNEL (イオンチャネル) /  LIGAND (リガンド)
LIGAND (リガンド) 機能・相同性情報
機能・相同性情報 HCN channels /
HCN channels /  HCN channel complex / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / sodium ion import across plasma membrane /
HCN channel complex / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / sodium ion import across plasma membrane /  voltage-gated sodium channel activity /
voltage-gated sodium channel activity /  regulation of membrane depolarization / potassium ion import across plasma membrane /
regulation of membrane depolarization / potassium ion import across plasma membrane /  voltage-gated potassium channel activity / sodium ion transmembrane transport ...
voltage-gated potassium channel activity / sodium ion transmembrane transport ... HCN channels /
HCN channels /  HCN channel complex / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / sodium ion import across plasma membrane /
HCN channel complex / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / sodium ion import across plasma membrane /  voltage-gated sodium channel activity /
voltage-gated sodium channel activity /  regulation of membrane depolarization / potassium ion import across plasma membrane /
regulation of membrane depolarization / potassium ion import across plasma membrane /  voltage-gated potassium channel activity / sodium ion transmembrane transport /
voltage-gated potassium channel activity / sodium ion transmembrane transport /  cAMP binding / cellular response to cAMP / potassium ion transmembrane transport / somatodendritic compartment / dendrite membrane /
cAMP binding / cellular response to cAMP / potassium ion transmembrane transport / somatodendritic compartment / dendrite membrane /  regulation of membrane potential / dendritic shaft /
regulation of membrane potential / dendritic shaft /  PDZ domain binding / molecular adaptor activity /
PDZ domain binding / molecular adaptor activity /  神経繊維 / neuronal cell body /
神経繊維 / neuronal cell body /  樹状突起 / protein-containing complex binding /
樹状突起 / protein-containing complex binding /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  細胞膜
細胞膜
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 2 Å
多波長異常分散 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nature / 年: 2003
ジャーナル: Nature / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1q43.cif.gz
1q43.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1q43.ent.gz
pdb1q43.ent.gz PDB形式
PDB形式 1q43.json.gz
1q43.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/q4/1q43
https://data.pdbj.org/pub/pdb/validation_reports/q4/1q43 ftp://data.pdbj.org/pub/pdb/validation_reports/q4/1q43
ftp://data.pdbj.org/pub/pdb/validation_reports/q4/1q43 リンク
リンク 集合体
集合体




 要素
要素
 Mus musculus (ハツカネズミ) / 遺伝子: HCN2, BCNG2 OR HAC1 / 器官: brain
Mus musculus (ハツカネズミ) / 遺伝子: HCN2, BCNG2 OR HAC1 / 器官: brain 脳 / プラスミド: pETGQ / 生物種 (発現宿主): Escherichia coli / 発現宿主:
脳 / プラスミド: pETGQ / 生物種 (発現宿主): Escherichia coli / 発現宿主: 
 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL-21 (DE3) / 参照: UniProt: O88703
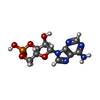
Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL-21 (DE3) / 参照: UniProt: O88703 環状アデノシン一リン酸
環状アデノシン一リン酸 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  CHESS
CHESS  / ビームライン: F2 / 波長: 0.979493,0.979184,0.934038
/ ビームライン: F2 / 波長: 0.979493,0.979184,0.934038 解析
解析 :
:  多波長異常分散 / 解像度: 2→19.81 Å / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
多波長異常分散 / 解像度: 2→19.81 Å / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber : 0.195
: 0.195  ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj