| 登録情報 | データベース: PDB / ID: 1nz6
|
|---|
| タイトル | Crystal Structure of Auxilin J-Domain |
|---|
 要素 要素 | Auxilin |
|---|
 キーワード キーワード | PROTEIN BINDING / Alpha Helix / Anti-Parallel Helix Hairpin |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of clathrin coat assembly / synaptic vesicle recycling / clathrin heavy chain binding / synaptic vesicle uncoating / clathrin coat disassembly / 加水分解酵素; エステル加水分解酵素; 1価のリン酸エステル加水分解酵素 / clathrin-dependent endocytosis / clathrin-coated vesicle / clathrin binding / intracellular transport ...regulation of clathrin coat assembly / synaptic vesicle recycling / clathrin heavy chain binding / synaptic vesicle uncoating / clathrin coat disassembly / 加水分解酵素; エステル加水分解酵素; 1価のリン酸エステル加水分解酵素 / clathrin-dependent endocytosis / clathrin-coated vesicle / clathrin binding / intracellular transport / heat shock protein binding / protein tyrosine phosphatase activity / SH3 domain binding / presynapse / vesicle / molecular adaptor activity / postsynaptic density / protein domain specific binding / intracellular membrane-bounded organelle / cytoplasm類似検索 - 分子機能 DnaJ domain / Tensin phosphatase, C2 domain / Tensin-type phosphatase domain / C2 domain of PTEN tumour-suppressor protein / Phosphatase tensin-type domain profile. / C2 tensin-type domain profile. / C2 domain of PTEN tumour-suppressor protein / DnaJ domain / DnaJ molecular chaperone homology domain / dnaJ domain profile. ...DnaJ domain / Tensin phosphatase, C2 domain / Tensin-type phosphatase domain / C2 domain of PTEN tumour-suppressor protein / Phosphatase tensin-type domain profile. / C2 tensin-type domain profile. / C2 domain of PTEN tumour-suppressor protein / DnaJ domain / DnaJ molecular chaperone homology domain / dnaJ domain profile. / Chaperone J-domain superfamily / DnaJ domain / Tyrosine-specific protein phosphatases domain / Tyrosine specific protein phosphatases domain profile. / Protein-tyrosine phosphatase-like / C2 domain superfamily / Helix Hairpins / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Bos taurus (ウシ) Bos taurus (ウシ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.5 Å 多波長異常分散 / 解像度: 2.5 Å |
|---|
 データ登録者 データ登録者 | Jiang, J. / Taylor, A.B. / Prasad, K. / Ishikawa-Brush, Y. / Hart, P.J. / Lafer, E.M. / Sousa, R. |
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2003 ジャーナル: Biochemistry / 年: 2003
タイトル: Structure-function analysis of the auxilin J-domain reveals an extended Hsc70 interaction interface.
著者: Jiang, J. / Taylor, A.B. / Prasad, K. / Ishikawa-Brush, Y. / Hart, P.J. / Lafer, E.M. / Sousa, R. |
|---|
| 履歴 | | 登録 | 2003年2月16日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2003年4月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月29日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2011年11月16日 | Group: Atomic model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 2.5 Å
多波長異常分散 / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2003
ジャーナル: Biochemistry / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1nz6.cif.gz
1nz6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1nz6.ent.gz
pdb1nz6.ent.gz PDB形式
PDB形式 1nz6.json.gz
1nz6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1nz6_validation.pdf.gz
1nz6_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1nz6_full_validation.pdf.gz
1nz6_full_validation.pdf.gz 1nz6_validation.xml.gz
1nz6_validation.xml.gz 1nz6_validation.cif.gz
1nz6_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/nz/1nz6
https://data.pdbj.org/pub/pdb/validation_reports/nz/1nz6 ftp://data.pdbj.org/pub/pdb/validation_reports/nz/1nz6
ftp://data.pdbj.org/pub/pdb/validation_reports/nz/1nz6 リンク
リンク 集合体
集合体
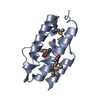

 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X8C / 波長: 0.9788, 0.9790, 0.9813
/ ビームライン: X8C / 波長: 0.9788, 0.9790, 0.9813 解析
解析 多波長異常分散 / 解像度: 2.5→50 Å / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
多波長異常分散 / 解像度: 2.5→50 Å / σ(F): 0 / 立体化学のターゲット値: Engh & Huber ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj








