+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ie1 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
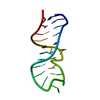
| タイトル | NMR Solution Structure of an In Vitro Selected RNA which is Sequence Specifically Recognized by Hamster Nucleolin RBD12. | ||||||||||||||||||
 要素 要素 | 5'-R(* キーワード キーワード RNA (リボ核酸) / eucaryotic loop E motif A-form helix flexible loop RNA (リボ核酸) / eucaryotic loop E motif A-form helix flexible loop機能・相同性 |  リボ核酸 / RNA (> 10) リボ核酸 / RNA (> 10) 機能・相同性情報 機能・相同性情報手法 |  溶液NMR / Simulated annealing starting from randomized templates. Base planarity restraints were added. 溶液NMR / Simulated annealing starting from randomized templates. Base planarity restraints were added.  データ登録者 データ登録者Bouvet, P. / Allain, F.H.-T. / Finger, L.D. / Dieckmann, T. / Feigon, J. |  引用 引用 ジャーナル: J.Mol.Biol. / 年: 2001 ジャーナル: J.Mol.Biol. / 年: 2001タイトル: Recognition of pre-formed and flexible elements of an RNA stem-loop by nucleolin. 著者: Bouvet, P. / Allain, F.H. / Finger, L.D. / Dieckmann, T. / Feigon, J. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ie1.cif.gz 1ie1.cif.gz | 250.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ie1.ent.gz pdb1ie1.ent.gz | 207.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ie1.json.gz 1ie1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ie/1ie1 https://data.pdbj.org/pub/pdb/validation_reports/ie/1ie1 ftp://data.pdbj.org/pub/pdb/validation_reports/ie/1ie1 ftp://data.pdbj.org/pub/pdb/validation_reports/ie/1ie1 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 7095.323 Da / 分子数: 1 / 由来タイプ: 合成 詳細: Made by in vitro transcription using Milligan templates. |
|---|
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: RNA was also assigned using U/C, A, and, G specifically labeled samples. Dieckmann, T., Feigon, J. J. Biomol. NMR 9 (1997) 259-272. |
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
| |||||||||||||||||||||||||
結晶化 | *PLUS 手法: その他 / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: Simulated annealing starting from randomized templates. Base planarity restraints were added. ソフトェア番号: 1 | ||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: all calculated structures submitted 計算したコンフォーマーの数: 18 / 登録したコンフォーマーの数: 18 |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj





























