+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dtt | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURE OF HIV-1 REVERSE TRANSCRIPTASE IN COMPLEX WITH PETT-2 (PETT130A94) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE/TRANSFERASE / HIV-1 reverse transcriptase AIDS / non-nucleoside inhibitor /  drug design (医薬品設計) / HYDROLASE-TRANSFERASE COMPLEX drug design (医薬品設計) / HYDROLASE-TRANSFERASE COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus ... integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus ... integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus / APOBEC3G mediated resistance to HIV-1 infection / Binding and entry of HIV virion / integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus / APOBEC3G mediated resistance to HIV-1 infection / Binding and entry of HIV virion /  ウイルスのライフサイクル / : / : / Assembly Of The HIV Virion / ウイルスのライフサイクル / : / : / Assembly Of The HIV Virion /  HIV-1 retropepsin / HIV-1 retropepsin /  retroviral ribonuclease H / Budding and maturation of HIV virion / retroviral ribonuclease H / Budding and maturation of HIV virion /  exoribonuclease H / exoribonuclease H /  exoribonuclease H activity / protein processing / host multivesicular body / exoribonuclease H activity / protein processing / host multivesicular body /  逆転写酵素 / viral genome integration into host DNA / viral penetration into host nucleus / establishment of integrated proviral latency / 逆転写酵素 / viral genome integration into host DNA / viral penetration into host nucleus / establishment of integrated proviral latency /  RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity /  転移酵素; リンを含む基を移すもの; 核酸を移すもの / 転移酵素; リンを含む基を移すもの; 核酸を移すもの /  peptidase activity / symbiont-mediated suppression of host gene expression / viral nucleocapsid / DNA recombination / peptidase activity / symbiont-mediated suppression of host gene expression / viral nucleocapsid / DNA recombination /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  DNAポリメラーゼ / aspartic-type endopeptidase activity / DNAポリメラーゼ / aspartic-type endopeptidase activity /  DNA-directed DNA polymerase activity / symbiont entry into host cell / DNA-directed DNA polymerase activity / symbiont entry into host cell /  lipid binding / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / lipid binding / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity /  DNA binding / DNA binding /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  生体膜 / identical protein binding 生体膜 / identical protein binding類似検索 - 分子機能 | ||||||
| 生物種 |    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 3 Å シンクロトロン / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Ren, J. / Diprose, J. / Warren, J. / Esnouf, R.M. / Bird, L.E. / Ikemizu, S. / Slater, M. / Milton, J. / Balzarini, J. / Stuart, D.I. / Stammers, D.K. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2000 ジャーナル: J.Biol.Chem. / 年: 2000タイトル: Phenylethylthiazolylthiourea (PETT) non-nucleoside inhibitors of HIV-1 and HIV-2 reverse transcriptases. Structural and biochemical analyses. 著者: Ren, J. / Diprose, J. / Warren, J. / Esnouf, R.M. / Bird, L.E. / Ikemizu, S. / Slater, M. / Milton, J. / Balzarini, J. / Stuart, D.I. / Stammers, D.K. #1:  ジャーナル: J.Med.Chem. / 年: 1999 ジャーナル: J.Med.Chem. / 年: 1999タイトル: Crystallographic analysis of the binding modes of thiazoloisoindolinone non-nucleoside inhibitors to HIV-1 reverse transcriptase and comparison with modeling studies. 著者: Ren, J. / Esnouf, R.M. / Hopkins, A.L. / Stuart, D.I. / Stammers, D.K. #2:  ジャーナル: J.Med.Chem. / 年: 1999 ジャーナル: J.Med.Chem. / 年: 1999タイトル: Design of MKC-442 (emivirine) analogues with improved activity against drug-resistant HIV mutants. 著者: Hopkins, A.L. / Ren, J. / Tanaka, H. / Baba, M. / Okamato, M. / Stuart, D.I. / Stammers, D.K. #3:  ジャーナル: Biochemistry / 年: 1998 ジャーナル: Biochemistry / 年: 1998タイトル: Crystal structures of HIV-1 reverse transcriptase in complex with carboxanilide derivatives. 著者: Ren, J. / Esnouf, R.M. / Hopkins, A.L. / Warren, J. / Balzarini, J. / Stuart, D.I. / Stammers, D.K. #4:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1998 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1998タイトル: 3'-Azido-3'-deoxythymidine drug resistance mutations in HIV-1 reverse transcriptase can induce long range conformational changes. 著者: Ren, J. / Esnouf, R.M. / Hopkins, A.L. / Jones, E.Y. / Kirby, I. / Keeling, J. / Ross, C.K. / Larder, B.A. / Stuart, D.I. / Stammers, D.K. #5: ジャーナル: Acta Crystallogr.,Sect.D / 年: 1998 タイトル: Continuous and discontinuous changes in the unit cell of HIV-1 reverse transcriptase crystals on dehydration. 著者: Esnouf, R.M. / Ren, J. / Garman, E.F. / Somers, D.O. / Ross, C.K. / Jones, E.Y. / Stammers, D.K. / Stuart, D.I. #6:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1997 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1997タイトル: Unique features in the structure of the complex between HIV-1 reverse transcriptase and the bis(heteroaryl)piperazine (BHAP) U-90152 explain resistance mutations for this nonnucleoside inhibitor. 著者: Esnouf, R.M. / Ren, J. / Hopkins, A.L. / Ross, C.K. / Jones, E.Y. / Stammers, D.K. / Stuart, D.I. #7:  ジャーナル: J.Med.Chem. / 年: 1996 ジャーナル: J.Med.Chem. / 年: 1996タイトル: Complexes of HIV-1 reverse transcriptase with inhibitors of the HEPT series reveal conformational changes relevant to the design of potent non-nucleoside inhibitors. 著者: Hopkins, A.L. / Ren, J. / Esnouf, R.M. / Willcox, B.E. / Jones, E.Y. / Ross, C. / Miyasaka, T. / Walker, R.T. / Tanaka, H. / Stammers, D.K. / Stuart, D.I. #8:  ジャーナル: Structure / 年: 1995 ジャーナル: Structure / 年: 1995タイトル: The structure of HIV-1 reverse transcriptase complexed with 9-chloro-TIBO: lessons for inhibitor design. 著者: Ren, J. / Esnouf, R. / Hopkins, A. / Ross, C. / Jones, Y. / Stammers, D. / Stuart, D. #9:  ジャーナル: Nat.Struct.Biol. / 年: 1995 ジャーナル: Nat.Struct.Biol. / 年: 1995タイトル: High resolution structures of HIV-1 RT from four RT-inhibitor complexes. 著者: Ren, J. / Esnouf, R. / Garman, E. / Somers, D. / Ross, C. / Kirby, I. / Keeling, J. / Darby, G. / Jones, Y. / Stuart, D. #10:  ジャーナル: Nat.Struct.Biol. / 年: 1995 ジャーナル: Nat.Struct.Biol. / 年: 1995タイトル: Mechanism of inhibition of HIV-1 reverse transcriptase by non-nucleoside inhibitors. 著者: Esnouf, R. / Ren, J. / Ross, C. / Jones, Y. / Stammers, D. / Stuart, D. #11: ジャーナル: J.Mol.Biol. / 年: 1994 タイトル: Crystals of HIV-1 reverse transcriptase diffracting to 2.2 A resolution. 著者: Stammers, D.K. / Somers, D.O. / Ross, C.K. / Kirby, I. / Ray, P.H. / Wilson, J.E. / Norman, M. / Ren, J.S. / Esnouf, R.M. / Garman, E.F. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dtt.cif.gz 1dtt.cif.gz | 201.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dtt.ent.gz pdb1dtt.ent.gz | 167.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dtt.json.gz 1dtt.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dt/1dtt https://data.pdbj.org/pub/pdb/validation_reports/dt/1dtt ftp://data.pdbj.org/pub/pdb/validation_reports/dt/1dtt ftp://data.pdbj.org/pub/pdb/validation_reports/dt/1dtt | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 64594.949 Da / 分子数: 1 / 断片: P66 / 由来タイプ: 組換発現 由来: (組換発現)    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)属: Lentivirus  レンチウイルス属 / 発現宿主: レンチウイルス属 / 発現宿主:  Bacteria (細菌) / 参照: UniProt: P04585 Bacteria (細菌) / 参照: UniProt: P04585 |
|---|---|
| #2: タンパク質 | 分子量: 51399.047 Da / 分子数: 1 / 断片: P51 / 由来タイプ: 組換発現 由来: (組換発現)    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)属: Lentivirus  レンチウイルス属 / 発現宿主: レンチウイルス属 / 発現宿主:  Bacteria (細菌) / 参照: UniProt: P04585 Bacteria (細菌) / 参照: UniProt: P04585 |
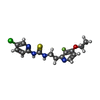
| #3: 化合物 | ChemComp-FTC / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 2 X線回折 / 使用した結晶の数: 2 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.46 Å3/Da / 溶媒含有率: 50.09 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5 詳細: see reference 11, pH 5, VAPOR DIFFUSION, SITTING DROP, temperature 277K | |||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / pH: 5 / 詳細: Stammers, D.K., (1994) J.Mol.Biol., 242, 586. | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| |||||||||||||||
| 検出器 |
| |||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 | |||||||||||||||
| 反射 | 解像度: 3→30 Å / Num. obs: 21409 / % possible obs: 89.2 % / Observed criterion σ(I): -1.5 / 冗長度: 4.2 % / Rmerge(I) obs: 0.095 / Net I/σ(I): 11.4 | |||||||||||||||
| 反射 シェル | 解像度: 3→3.11 Å / 冗長度: 3 % / Rmerge(I) obs: 0.592 / Num. unique all: 1793 / % possible all: 76.7 | |||||||||||||||
| 反射 | *PLUS 最低解像度: 30 Å / Num. measured all: 86903 | |||||||||||||||
| 反射 シェル | *PLUS % possible obs: 81.7 % / Num. unique obs: 1771 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 3→30 Å / σ(F): 0 / 立体化学のターゲット値: Engh & Huber 詳細: Due to the low ratio between the number of reflections and the number of parameters to be refined, positional restraints were applied to all atoms distant from the NNRTI-binding site (defined ...詳細: Due to the low ratio between the number of reflections and the number of parameters to be refined, positional restraints were applied to all atoms distant from the NNRTI-binding site (defined as greater than 25 anstrom from the CA atom of residue 188) throughout the refinement.
| ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3→30 Å
| ||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / 分類: refinement | ||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 3 Å / 最低解像度: 30 Å / σ(F): 0 / Rfactor obs: 0.199 | ||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: c_angle_deg / Dev ideal: 1.4 |
 ムービー
ムービー コントローラー
コントローラー










































 PDBj
PDBj