+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
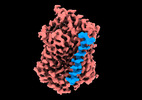
| タイトル | Structure of human SGLT2-MAP17 complex bound with substrate AMG in the occluded conformation | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  glucose transporter (グルコーストランスポーター) / glucose transporter (グルコーストランスポーター) /  SGLT / sodium glucose transporter / SGLT / sodium glucose transporter /  membrane protein (膜タンパク質) / membrane protein (膜タンパク質) /  PROTEIN TRANSPORT PROTEIN TRANSPORT | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報low-affinity glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / glucose:sodium symporter activity / alpha-glucoside transmembrane transporter activity / hexose transmembrane transport / D-glucose transmembrane transporter activity / Cellular hexose transport /  renal glucose absorption / glucose import across plasma membrane ...low-affinity glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / glucose:sodium symporter activity / alpha-glucoside transmembrane transporter activity / hexose transmembrane transport / D-glucose transmembrane transporter activity / Cellular hexose transport / renal glucose absorption / glucose import across plasma membrane ...low-affinity glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / glucose:sodium symporter activity / alpha-glucoside transmembrane transporter activity / hexose transmembrane transport / D-glucose transmembrane transporter activity / Cellular hexose transport /  renal glucose absorption / glucose import across plasma membrane / sodium ion import across plasma membrane / sodium ion transport / carbohydrate metabolic process / apical plasma membrane / extracellular exosome / renal glucose absorption / glucose import across plasma membrane / sodium ion import across plasma membrane / sodium ion transport / carbohydrate metabolic process / apical plasma membrane / extracellular exosome /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.33 Å クライオ電子顕微鏡法 / 解像度: 3.33 Å | ||||||||||||
 データ登録者 データ登録者 | Chen L / Niu Y / Cui W | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structures of human SGLT in the occluded state reveal conformational changes during sugar transport. 著者: Wenhao Cui / Yange Niu / Zejian Sun / Rui Liu / Lei Chen /  要旨: Sodium-Glucose Cotransporters (SGLT) mediate the uphill uptake of extracellular sugars and play fundamental roles in sugar metabolism. Although their structures in inward-open and outward-open ...Sodium-Glucose Cotransporters (SGLT) mediate the uphill uptake of extracellular sugars and play fundamental roles in sugar metabolism. Although their structures in inward-open and outward-open conformations are emerging from structural studies, the trajectory of how SGLTs transit from the outward-facing to the inward-facing conformation remains unknown. Here, we present the cryo-EM structures of human SGLT1 and SGLT2 in the substrate-bound state. Both structures show an occluded conformation, with not only the extracellular gate but also the intracellular gate tightly sealed. The sugar substrate are caged inside a cavity surrounded by TM1, TM2, TM3, TM6, TM7, and TM10. Further structural analysis reveals the conformational changes associated with the binding and release of substrates. These structures fill a gap in our understanding of the structural mechanisms of SGLT transporters. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33963.map.gz emd_33963.map.gz | 7.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33963-v30.xml emd-33963-v30.xml emd-33963.xml emd-33963.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33963_fsc.xml emd_33963_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33963.png emd_33963.png | 40.8 KB | ||
| マスクデータ |  emd_33963_msk_1.map emd_33963_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-33963.cif.gz emd-33963.cif.gz | 5.9 KB | ||
| その他 |  emd_33963_additional_1.map.gz emd_33963_additional_1.map.gz emd_33963_additional_2.map.gz emd_33963_additional_2.map.gz emd_33963_half_map_1.map.gz emd_33963_half_map_1.map.gz emd_33963_half_map_2.map.gz emd_33963_half_map_2.map.gz | 78.9 MB 41.3 MB 77.7 MB 77.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33963 http://ftp.pdbj.org/pub/emdb/structures/EMD-33963 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33963 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33963 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33963.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33963.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.821 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33963_msk_1.map emd_33963_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_33963_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_33963_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33963_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33963_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human SGLT2-MAP17 complex
| 全体 | 名称: human SGLT2-MAP17 complex |
|---|---|
| 要素 |
|
-超分子 #1: human SGLT2-MAP17 complex
| 超分子 | 名称: human SGLT2-MAP17 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Sodium/glucose cotransporter 2
| 分子 | 名称: Sodium/glucose cotransporter 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 72.949414 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEEHTEAGSA PEMGAQKALI DNPADILVIA AYFLLVIGVG LWSMCRTNRG TVGGYFLAGR SMVWWPVGAS LFASNIGSGH FVGLAGTGA ASGLAVAGFE WNALFVVLLL GWLFAPVYLT AGVITMPQYL RKRFGGRRIR LYLSVLSLFL YIFTKISVDM F SGAVFIQQ ...文字列: MEEHTEAGSA PEMGAQKALI DNPADILVIA AYFLLVIGVG LWSMCRTNRG TVGGYFLAGR SMVWWPVGAS LFASNIGSGH FVGLAGTGA ASGLAVAGFE WNALFVVLLL GWLFAPVYLT AGVITMPQYL RKRFGGRRIR LYLSVLSLFL YIFTKISVDM F SGAVFIQQ ALGWNIYASV IALLGITMIY TVTGGLAALM YTDTVQTFVI LGGACILMGY AFHEVGGYSG LFDKYLGAAT SL TVSEDPA VGNISSFCYR PRPDSYHLLR HPVTGDLPWP ALLLGLTIVS GWYWCSDQVI VQRCLAGKSL THIKAGCILC GYL KLTPMF LMVMPGMISR ILYPDEVACV VPEVCRRVCG TEVGCSNIAY PRLVVKLMPN GLRGLMLAVM LAALMSSLAS IFNS SSTLF TMDIYTRLRP RAGDRELLLV GRLWVVFIVV VSVAWLPVVQ AAQGGQLFDY IQAVSSYLAP PVSAVFVLAL FVPRV NEQG AFWGLIGGLL MGLARLIPEF SFGSGSCVQP SACPAFLCGV HYLYFAIVLF FCSGLLTLTV SLCTAPIPRK HLHRLV FSL RHSKEEREDL DADEQQGSSL PVQNGCPESA MEMNEPQAPA PSLFRQCLLW FCGMSRGGVG SPPPLTQEEA AAAARRL ED ISEDPSWARV VNLNALLMMA VAVFLWGFYA UniProtKB: Sodium/glucose cotransporter 2 |
-分子 #2: PDZK1-interacting protein 1
| 分子 | 名称: PDZK1-interacting protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.235 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSALSLLILG LLTAVPPASC QQGLGNLQPW MQGLIAVAVF LVLVAIAFAV NHFWCQEEPE PAHMILTVGN KADGVLVGTD GRYSSMAAS FRSSEHENAY ENVPEEEGKV RSTPM UniProtKB: PDZK1-interacting protein 1 |
-分子 #3: methyl alpha-D-glucopyranoside
| 分子 | 名称: methyl alpha-D-glucopyranoside / タイプ: ligand / ID: 3 / コピー数: 1 / 式: GYP |
|---|---|
| 分子量 | 理論値: 194.182 Da |
| Chemical component information |  ChemComp-GYP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X










































