+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of tissue-specific lipid scramblase ATG9B homotrimer, refined with C3 symmetry applied | ||||||||||||||||||
 マップデータ マップデータ | ATG9B C3 map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード |  membrane protein (膜タンパク質) / lipid scramblase / membrane protein (膜タンパク質) / lipid scramblase /  autophagy (オートファジー) / phagopore / lipid transporter / Atg9 / Atg9B / LIPID TRANSPORT autophagy (オートファジー) / phagopore / lipid transporter / Atg9 / Atg9B / LIPID TRANSPORT | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis / phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis /  オートファジー / オートファジー /  autophagosome assembly / autophagosome assembly /  オートファゴソーム ... オートファゴソーム ... phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis / phospholipid scramblase activity / programmed necrotic cell death / nucleophagy / protein localization to phagophore assembly site / phagophore assembly site membrane / phagophore assembly site / bone morphogenesis /  オートファジー / オートファジー /  autophagosome assembly / autophagosome assembly /  オートファゴソーム / オートファゴソーム /  ゴルジ体 / recycling endosome membrane / ゴルジ体 / recycling endosome membrane /  ゴルジ体 / endoplasmic reticulum membrane ゴルジ体 / endoplasmic reticulum membrane類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
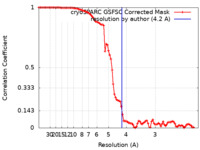
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.2 Å クライオ電子顕微鏡法 / 解像度: 4.2 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Chiduza GN / Pye VE / Tooze SA / Cherepanov P | ||||||||||||||||||
| 資金援助 |  英国, European Union, 5件 英国, European Union, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Autophagy / 年: 2024 ジャーナル: Autophagy / 年: 2024タイトル: ATG9B is a tissue-specific homotrimeric lipid scramblase that can compensate for ATG9A. 著者: George N Chiduza / Acely Garza-Garcia / Eugenia Almacellas / Stefano De Tito / Valerie E Pye / Alexander R van Vliet / Peter Cherepanov / Sharon A Tooze /  要旨: Macroautophagy/autophagy is a fundamental aspect of eukaryotic biology, and the autophay-related protein ATG9A is part of the core machinery facilitating this process. In addition to ATG9A ...Macroautophagy/autophagy is a fundamental aspect of eukaryotic biology, and the autophay-related protein ATG9A is part of the core machinery facilitating this process. In addition to ATG9A vertebrates encode ATG9B, a poorly characterized paralog expressed in a subset of tissues. Herein, we characterize the structure of human ATG9B revealing the conserved homotrimeric quaternary structure and explore the conformational dynamics of the protein. Consistent with the experimental structure and computational chemistry, we establish that ATG9B is a functional lipid scramblase. We show that ATG9B can compensate for the absence of ATG9A in starvation-induced autophagy displaying similar subcellular trafficking and steady-state localization. Finally, we demonstrate that ATG9B can form a heteromeric complex with ATG2A. By establishing the molecular structure and function of ATG9B, our results inform the exploration of niche roles for autophagy machinery in more complex eukaryotes and reveal insights relevant across species. ATG: autophagy related; CHS: cholesteryl hemisuccinate; cryo-EM: single-particle cryogenic electron microscopy; CTF: contrast transfer function: CTH: C- terminal α helix; FSC: fourier shell correlation; HDIR: HORMA domain interacting region; LMNG: lauryl maltose neopentyl glycol; MD: molecular dynamics simulations; MSA: multiple sequence alignment; NBD-PE: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(7-nitro-2-1,3-benzoxadiazol-4-yl ammonium salt); POPC: palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine; RBG: repeating beta groove domain; RMSD: root mean square deviation; SEC: size-exclusion chromatography; TMH: transmembrane helix. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17789.map.gz emd_17789.map.gz | 78.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17789-v30.xml emd-17789-v30.xml emd-17789.xml emd-17789.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17789_fsc.xml emd_17789_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17789.png emd_17789.png | 86.9 KB | ||
| マスクデータ |  emd_17789_msk_1.map emd_17789_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17789.cif.gz emd-17789.cif.gz | 6.7 KB | ||
| その他 |  emd_17789_additional_1.map.gz emd_17789_additional_1.map.gz emd_17789_half_map_1.map.gz emd_17789_half_map_1.map.gz emd_17789_half_map_2.map.gz emd_17789_half_map_2.map.gz | 74.9 MB 77.8 MB 77.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17789 http://ftp.pdbj.org/pub/emdb/structures/EMD-17789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17789 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8poeMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17789.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17789.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATG9B C3 map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17789_msk_1.map emd_17789_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: ATG9B C3 DeepEMhancer map
| ファイル | emd_17789_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATG9B C3 DeepEMhancer map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ATG9B C3 half map 2
| ファイル | emd_17789_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATG9B C3 half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ATG9B C3 half map 1
| ファイル | emd_17789_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATG9B C3 half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homotrimer of ATG9B
| 全体 | 名称: Homotrimer of ATG9B |
|---|---|
| 要素 |
|
-超分子 #1: Homotrimer of ATG9B
| 超分子 | 名称: Homotrimer of ATG9B / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 303 KDa |
-分子 #1: Autophagy-related protein 9B
| 分子 | 名称: Autophagy-related protein 9B / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 105.667008 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DYKDDDDKDY KDDDDKDYKD DDDKHHHHHH ENLYFQGVSR MGWGGRRRRL GRWGDLGPGS VPLLPMPLPP PPPPSCRGPG GGRISIFSL SPAPHTRSSP SSFSPPTAGP PCSVLQGTGA SQSCHSALPI PATPPTQAQP AMTPASASPS WGSHSTPPLA P ATPTPSQQ ...文字列: DYKDDDDKDY KDDDDKDYKD DDDKHHHHHH ENLYFQGVSR MGWGGRRRRL GRWGDLGPGS VPLLPMPLPP PPPPSCRGPG GGRISIFSL SPAPHTRSSP SSFSPPTAGP PCSVLQGTGA SQSCHSALPI PATPPTQAQP AMTPASASPS WGSHSTPPLA P ATPTPSQQ CPQDSPGLRV GPLIPEQDYE RLEDCDPEGS QDSPIHGEEQ QPLLHVPEGL RGSWHHIQNL DSFFTKIYSY HQ RNGFACI LLEDVFQLGQ FIFIVTFTTF LLRCVDYNVL FANQPSNHTR PGPFHSKVTL SDAILPSAQC AERIRSSPLL VLL LVLAAG FWLVQLLRSV CNLFSYWDIQ VFYREALHIP PEELSSVPWA EVQSRLLALQ RSGGLCVQPR PLTELDIHHR ILRY TNYQV ALANKGLLPA RCPLPWGGSA AFLSRGLALN VDLLLFRGPF SLFRGGWELP HAYKRSDQRG ALAARWGRTV LLLAA LNLA LSPLVLAWQV LHVFYSHVEL LRREPGALGA RGWSRLARLQ LRHFNELPHE LRARLARAYR PAAAFLRTAA PPAPLR TLL ARQLVFFAGA LFAALLVLTV YDEDVLAVEH VLTAMTALGV TATVARSFIP EEQCQGRAPQ LLLQTALAHM HYLPEEP GP GGRDRAYRQM AQLLQYRAVS LLEELLSPLL TPLFLLFWFR PRALEIIDFF HHFTVDVAGV GDICSFALMD VKRHGHPQ W LSAGQTEASL SQRAEDGKTE LSLMRFSLAH PLWRPPGHSS KFLGHLWGRV QQDAAAWGAT SARGPSTPGV LSNCTSPLP EAFLANLFVH PLLPPRDLSP TAPCPAAATA SLLASISRIA QDPSSVSPGG TGGQKLAQLP ELASAEMSLH VIYLHQLHQQ QQQQEPWGE AAASILSRPC SSPSQPPSPD EEKPSWSSDG SSPASSPRQQ WGTQKARNLF PGGFQVTTDT QKEPDRASCT D UniProtKB: Autophagy-related protein 9B |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 8.5 詳細: 20 mM Tris-HCl, pH 8.5; 200 mM NaCl; 1 mM TCEP; 10% glycerol supplemented with 0.001% w/v lauryl maltose neopentyl glycol and 0.0002% w/v cholesteryl hemisuccinate |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 50 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 46296 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 2 / 実像数: 37217 / 平均露光時間: 1.2 sec. / 平均電子線量: 48.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER 当てはまり具合の基準: Cross-correlation coeffecient |
| 得られたモデル |  PDB-8poe: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X