-検索条件
-検索結果
検索 (著者・登録者: yun & bai)の結果133件中、1から50件目までを表示しています

EMDB-36027: 
Human high-affinity choline transporter CHT1 in the HC-3-bound outward-facing open conformation, monomeric state

EMDB-36029: 
Human high-affinity choline transporter CHT1 in the inward-facing apo-open conformation

EMDB-36030: 
Human high-affinity choline transporter CHT1 in the choline-bound inward-facing occluded conformation

PDB-8j75: 
Human high-affinity choline transporter CHT1 in the HC-3-bound outward-facing open conformation, monomeric state

PDB-8j76: 
Human high-affinity choline transporter CHT1 in the inward-facing apo-open conformation

PDB-8j77: 
Human high-affinity choline transporter CHT1 in the choline-bound inward-facing occluded conformation

EMDB-36025: 
Human high-affinity choline transporter CHT1 in the HC-3-bound outward-facing open conformation, dimeric state

PDB-8j74: 
Human high-affinity choline transporter CHT1 in the HC-3-bound outward-facing open conformation, dimeric state

EMDB-35906: 
cryo-EM structure of human EMC
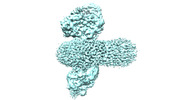
EMDB-35907: 
cryo-EM structure of human EMC and VDAC

PDB-8j0n: 
cryo-EM structure of human EMC

PDB-8j0o: 
cryo-EM structure of human EMC and VDAC

EMDB-42124: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab

PDB-8ucd: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab

EMDB-36855: 
S. cerevisiae Chs1 in complex with polyoxin B
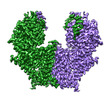
EMDB-36856: 
S. cerevisiae Chs1 in apo state

EMDB-36859: 
S. cerevisiae Chs1 in complex with UDP

EMDB-36862: 
S. cerevisiae Chs1 in complex with UDP-GlcNAc
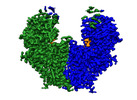
EMDB-36863: 
S. cerevisiae Chs1 in complex with UDP-GlcNAc and GlcNAc

EMDB-36864: 
S. cerevisiae Chs1 in complex with Nikkomycin Z
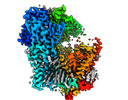
EMDB-36748: 
Structure of a fungal 1,3-beta-glucan synthase

PDB-8jzn: 
Structure of a fungal 1,3-beta-glucan synthase
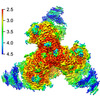
EMDB-28617: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB

EMDB-28618: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-COMBO1 FAB

EMDB-28619: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-MM28 FAB

PDB-8euu: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB

PDB-8euv: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-COMBO1 FAB

PDB-8euw: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-MM28 FAB

EMDB-36861: 
S. cerevisiae Chs1 in complex with UDP and GlcNAc

EMDB-36857: 
S. cerevisiae Chs1 in apo state incubated with GlcNAc
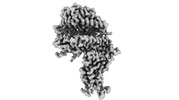
EMDB-33841: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.12.1 RBD in complex with human ACE2 (local refinement)
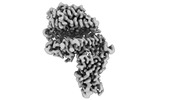
EMDB-33870: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with human ACE2 (local refinement)
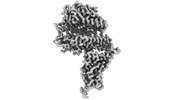
EMDB-34120: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with golden hamster ACE2 (local refinement)

EMDB-34138: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with mouse ACE2 (local refinement)

EMDB-34217: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with rat ACE2 (local refinement)

EMDB-34409: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4/5 RBD in complex with human ACE2 (local refinement)

EMDB-34494: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with human ACE2 (three-RBD-up conformation)

EMDB-34498: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2.12.1 spike trimer in complex with human ACE2 (three-RBD-up conformation)

EMDB-34499: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike protein in complex with mouse ACE2

EMDB-34506: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with rat ACE2

EMDB-34509: 
Cryo-EM map of SARS-CoV-2 Omicron BA.4/5 (N658S) spike trimer in complex with human ACE2 (three-RBD-up conformation)
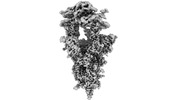
EMDB-34510: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike trimer in complex with golden hamster ACE2

PDB-7yhw: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2.12.1 RBD in complex with human ACE2 (local refinement)

PDB-7yj3: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with human ACE2 (local refinement)

PDB-7yv8: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with golden hamster ACE2 (local refinement)

PDB-7yvu: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with mouse ACE2 (local refinement)

PDB-8gry: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 RBD in complex with rat ACE2 (local refinement)

PDB-8h06: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4/5 RBD in complex with human ACE2 (local refinement)
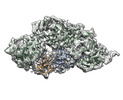
EMDB-40241: 
1:1:1 agrin/LRP4/MuSK complex

PDB-8s9p: 
1:1:1 agrin/LRP4/MuSK complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
