-検索条件
-検索結果
検索 (著者・登録者: wu & c)の結果3,527件中、1から50件目までを表示しています

EMDB-37130: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form

EMDB-37131: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in monomeric form

PDB-8kdb: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form

PDB-8kdc: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in monomeric form

PDB-8yy8: 
Fzd7 -Gs complex

EMDB-40180: 
MsbA bound to cerastecin C

EMDB-19861: 
Vertebrate microtubule-capping gamma-tubulin ring complex

EMDB-37362: 
CryoEM structure of human PI3K-alpha (P85/P110-H1047R) with QR-7909 binding at an allosteric site

EMDB-37363: 
CryoEM structure of human PI3K-alpha (P85/P110-H1047R) with QR-8557 binding at an allosteric site

PDB-8w9a: 
CryoEM structure of human PI3K-alpha (P85/P110-H1047R) with QR-7909 binding at an allosteric site

PDB-8w9b: 
CryoEM structure of human PI3K-alpha (P85/P110-H1047R) with QR-8557 binding at an allosteric site

EMDB-35323: 
Cryo-EM structure of the ISFba1 TnpB-reRNA-dsDNA complex

PDB-8iaz: 
Cryo-EM structure of the ISFba1 TnpB-reRNA-dsDNA complex

EMDB-34377: 
Cryo-EM structure of human Pannexin-3 R36S/F40R variant in pre-open state

PDB-8gyt: 
Cryo-EM structure of human Pannexin-3 R36S/F40R variant in pre-open state.

EMDB-35931: 
cryo-EM structures of Ufd4 in complex with Ubc4-Ub

PDB-8j1r: 
cryo-EM structures of Ufd4 in complex with Ubc4-Ub

EMDB-41363: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

PDB-8tl6: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

EMDB-35906: 
cryo-EM structure of human EMC
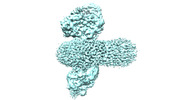
EMDB-35907: 
cryo-EM structure of human EMC and VDAC

PDB-8j0n: 
cryo-EM structure of human EMC

PDB-8j0o: 
cryo-EM structure of human EMC and VDAC
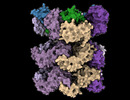
EMDB-19177: 
Structure of the 55LCC ATPase complex

PDB-8rhn: 
Structure of the 55LCC ATPase complex

EMDB-41877: 
Cryo-EM structure of long form insulin receptor (IR-B) in the apo state

EMDB-41878: 
Cryo-EM structure of long form insulin receptor (IR-B) with four IGF2 bound, symmetric conformation.

EMDB-41880: 
Cryo-EM structure of long form insulin receptor (IR-B) with three IGF2 bound, asymmetric conformation.

EMDB-43279: 
Cryo-EM structure of short form insulin receptor (IR-A) with four IGF2 bound, symmetric conformation.

EMDB-43280: 
Cryo-EM structure of short form insulin receptor (IR-A) with three IGF2 bound, asymmetric conformation.

PDB-8u4b: 
Cryo-EM structure of long form insulin receptor (IR-B) in the apo state

PDB-8u4c: 
Cryo-EM structure of long form insulin receptor (IR-B) with four IGF2 bound, symmetric conformation.

PDB-8u4e: 
Cryo-EM structure of long form insulin receptor (IR-B) with three IGF2 bound, asymmetric conformation.

PDB-8vjb: 
Cryo-EM structure of short form insulin receptor (IR-A) with four IGF2 bound, symmetric conformation.

PDB-8vjc: 
Cryo-EM structure of short form insulin receptor (IR-A) with three IGF2 bound, asymmetric conformation.

EMDB-38158: 
P/Q type calcium channel

EMDB-38159: 
P/Q type calcium channel in complex with omega-conotoxin MVIIC

EMDB-38160: 
P/Q type calcium channel in complex with omega-Agatoxin IVA

PDB-8x90: 
P/Q type calcium channel

PDB-8x91: 
P/Q type calcium channel in complex with omega-conotoxin MVIIC

PDB-8x93: 
P/Q type calcium channel in complex with omega-Agatoxin IVA

EMDB-33347: 
Cryo-EM structure of S glycoprotein encoded by the Covid-19 mRNA vaccine candidate RQ3013 (Postfusion state)

PDB-7xog: 
Cryo-EM structure of S glycoprotein encoded by the Covid-19 mRNA vaccine candidate RQ3013 (Postfusion state)

EMDB-35832: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

PDB-8iyx: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

EMDB-41625: 
Type IV pilus from Pseudomonas PAO1 strain

EMDB-41632: 
Asymmetric reconstruction of mature PP7 virions

EMDB-41633: 
Type IV pilus from Pseudomonas PAO1 strain with PP7 Maturation protein

EMDB-41675: 
The original consensus map of PP7 binding to T4P from PAO1

EMDB-41780: 
Composite map of PP7 binding to type IV pilus from PAO1
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
