-検索条件
-検索結果
検索 (著者・登録者: wong & w)の結果714件中、1から50件目までを表示しています

EMDB-29907: 
Structure of human NDS.1 Fab and 1G01 Fab in complex with influenza virus neuraminidase from A/Indiana/10/2011 (H3N2v); consensus map with only Fab 1G01 resolved

EMDB-29908: 
Structure of human NDS.1 Fab and 1G01 Fab in complex with influenza virus neuraminidase from A/Indiana/10/2011 (H3N2v), locally refined map

EMDB-29909: 
Structure of human NDS.3 Fab in complex with influenza virus neuraminidase from A/Darwin/09/2021 (H3N2)

PDB-8gat: 
Structure of human NDS.1 Fab and 1G01 Fab in complex with influenza virus neuraminidase from A/Indiana/10/2011 (H3N2v), based on consensus cryo-EM map with only Fab 1G01 resolved

PDB-8gau: 
Structure of human NDS.1 Fab and 1G01 Fab in complex with influenza virus neuraminidase from A/Indiana/10/2011 (H3N2v)

PDB-8gav: 
Structure of human NDS.3 Fab in complex with influenza virus neuraminidase from A/Darwin/09/2021 (H3N2)

EMDB-37631: 
Hepatitis B virus capsid (HBV core protein)

EMDB-37634: 
SR protein kinase 2 bound at 2-fold vertex of Hepatitis B virus capsid

EMDB-38062: 
Hepatitis B virus capsid in complex with SR protein kinase 2
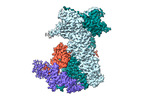
EMDB-18498: 
Cryo-EM structure of the benzo[a]pyrene-bound Hsp90-XAP2-AHR complex

PDB-8qmo: 
Cryo-EM structure of the benzo[a]pyrene-bound Hsp90-XAP2-AHR complex
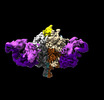
EMDB-41048: 
Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5

PDB-8t5c: 
Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5

EMDB-36887: 
Structure of GPR34-Gi complex

PDB-8k4n: 
Structure of GPR34-Gi complex
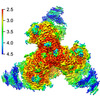
EMDB-28617: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB

EMDB-28618: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-COMBO1 FAB

EMDB-28619: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-MM28 FAB

PDB-8euu: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB

PDB-8euv: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-COMBO1 FAB

PDB-8euw: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-MM28 FAB

EMDB-28910: 
Glycan-Base ConC Env Trimer

PDB-8f7t: 
Glycan-Base ConC Env Trimer

EMDB-28115: 
Western Equine Encephalitis Virus-Like Particle in Complex with SKW19 Fab

EMDB-28116: 
Western Equine Encephalitis Virus-Like Particle in Complex with SKW24 Fab

EMDB-28117: 
Eastern Equine Encephalitis Virus-Like Particle in Complex with SKE26 Fab

EMDB-37096: 
Rpd3S histone deacetylase complex

EMDB-37122: 
Rpd3S in complex with 187bp nucleosome

EMDB-37123: 
Rpd3S in complex with nucleosome with H3K36MLA modification, H3K9Q mutation and 187bp DNA

EMDB-37124: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 187bp DNA, class1

EMDB-37125: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 187bp DNA, class2

EMDB-37126: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 187bp DNA, class3

EMDB-37127: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 167bp DNA

PDB-8kc7: 
Rpd3S histone deacetylase complex

PDB-8kd2: 
Rpd3S in complex with 187bp nucleosome

PDB-8kd3: 
Rpd3S in complex with nucleosome with H3K36MLA modification, H3K9Q mutation and 187bp DNA

PDB-8kd4: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 187bp DNA, class1

PDB-8kd5: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 187bp DNA, class2

PDB-8kd6: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 187bp DNA, class3

PDB-8kd7: 
Rpd3S in complex with nucleosome with H3K36MLA modification and 167bp DNA

EMDB-41302: 
Lassa GPC trimer in complex with Fab GP23

EMDB-28891: 
cryo-EM structure of a structurally designed Human metapneumovirus F protein in complex with antibody MPE8

PDB-8f6x: 
cryo-EM structure of a structurally designed Human metapneumovirus F protein in complex with antibody MPE8
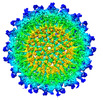
EMDB-28118: 
Cryo-EM structure of Antibody SKW11 in complex with Western Equine Encephalitis Virus-Like Particle
EMDB-28119: 
Cryo-EM structure of Antibody SKT05 in complex with Western Equine Encephalitis Virus-like Particle

EMDB-28187: 
Cryo-EM I1 reconstruction of antibody SKV09 in complex with VEEV alphavirus VLP

EMDB-28188: 
Cryo-EM I1 reconstruction of antibody SKV16 in complex with VEEV alphavirus VLP

EMDB-27757: 
Cryo-EM Structure of Eastern Equine Encephalitis Virus in complex with SKE26 Fab

EMDB-28056: 
Venezuelan equine encephalitis virus-like particle in complex with Fab SKT05

EMDB-28058: 
Venezuelan equine encephalitis virus-like particle in complex with Fab SKT05, local refinement
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
