-検索条件
-検索結果
検索 (著者・登録者: rak & a)の結果822件中、1から50件目までを表示しています

EMDB-41371: 
Cryo-EM structure of the Rev1-Polzeta-DNA-dCTP complex

EMDB-41372: 
Rev1(deltaN)-Polzeta-DNA-dCTP complex

EMDB-38453: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein in complex with ACE2 (1-up state)

EMDB-38454: 
Structure of the SARS-CoV-2 EG.5.1 spike RBD in complex with ACE2

PDB-8xlm: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein in complex with ACE2 (1-up state)

PDB-8xln: 
Structure of the SARS-CoV-2 EG.5.1 spike RBD in complex with ACE2

EMDB-37648: 
SARS-CoV-2 EG.5.1 spike glycoprotein (1-up state)

EMDB-37650: 
SARS-CoV-2 EG.5.1 spike glycoprotein (closed-2 state)

EMDB-37651: 
SARS-CoV-2 EG.5.1 spike glycoprotein (closed-1 state)

PDB-8wmd: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein (closed-2 state)

PDB-8wmf: 
Structure of the SARS-CoV-2 EG.5.1 spike glycoprotein (closed-1 state)

EMDB-37850: 
Cryo-EM structure of native H. thermoluteolus TH-1 GroEL

EMDB-37853: 
Cryo-EM structure of H. thermoluteolus GroEL-GroES2 football complex

EMDB-37862: 
Cryo-EM structure of H. thermophilus GroEL-GroES2 asymmetric football complex

EMDB-37863: 
Cryo-EM structure of H. thermophilus GroEL-GroES bullet complex

PDB-8wu4: 
Cryo-EM structure of native H. thermoluteolus TH-1 GroEL

PDB-8wuc: 
Cryo-EM structure of H. thermoluteolus GroEL-GroES2 football complex

PDB-8wuw: 
Cryo-EM structure of H. thermophilus GroEL-GroES2 asymmetric football complex

PDB-8wux: 
Cryo-EM structure of H. thermophilus GroEL-GroES bullet complex
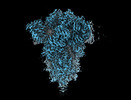
EMDB-18807: 
SD1-2 fab in complex with SARS-COV-2 BA.12.1 Spike Glycoprotein.
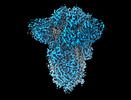
EMDB-18808: 
SD1-3 Fab in complex with SARS-CoV-2 BA.2.12.1 Spike Glycoprotein

PDB-8r1c: 
SD1-2 Fab in complex with SARS-CoV-2 BA.2.12.1 Spike Glycoprotein

PDB-8r1d: 
SD1-3 Fab in complex with SARS-CoV-2 BA.2.12.1 Spike Glycoprotein

EMDB-16680: 
BA.4/5-5 FAB IN COMPLEX WITH SARS-COV-2 BA.4 SPIKE GLYCOPROTEIN

PDB-8cin: 
BA.4/5-5 FAB IN COMPLEX WITH SARS-COV-2 BA.4 SPIKE GLYCOPROTEIN

EMDB-40811: 
Cryo-EM structure of NLRP3 open octamer
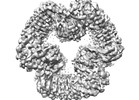
EMDB-40820: 
Cryo-EM structure of NLRP3 closed hexamer
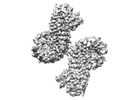
EMDB-40855: 
Structure of NLRP3 and NEK7 complex

PDB-8swf: 
Cryo-EM structure of NLRP3 open octamer

PDB-8swk: 
Cryo-EM structure of NLRP3 closed hexamer

PDB-8sxn: 
Structure of NLRP3 and NEK7 complex

EMDB-41725: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

EMDB-41727: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

PDB-8tyl: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

PDB-8tyo: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

EMDB-16279: 
Structure of CEACAM5 A3-B3 domain in Complex with Tusamitamab Fab

PDB-8bw0: 
Structure of CEACAM5 A3-B3 domain in Complex with Tusamitamab Fab

EMDB-36724: 
Structure of the SARS-CoV-2 XBB.1.5 spike glycoprotein (closed state 1)

EMDB-36726: 
Structure of the SARS-CoV-2 XBB.1.5 spike glycoprotein (closed state 2)

EMDB-36727: 
Structure of SARS-CoV-2 XBB.1.5 spike glycoprotein in complex with ACE2 (1-up state)

EMDB-36728: 
Structure of SARS-CoV-2 XBB.1.5 spike glycoprotein in complex with ACE2 (2-up state)

EMDB-36729: 
Structure of SARS-CoV-2 XBB.1.5 spike RBD in complex with ACE2

PDB-8jyk: 
Structure of the SARS-CoV-2 XBB.1.5 spike glycoprotein (closed state 1)

PDB-8jym: 
Structure of the SARS-CoV-2 XBB.1.5 spike glycoprotein (closed state 2)

PDB-8jyn: 
Structure of SARS-CoV-2 XBB.1.5 spike glycoprotein in complex with ACE2 (1-up state)

PDB-8jyo: 
Structure of SARS-CoV-2 XBB.1.5 spike glycoprotein in complex with ACE2 (2-up state)

PDB-8jyp: 
Structure of SARS-CoV-2 XBB.1.5 spike RBD in complex with ACE2

EMDB-42502: 
Cyanobacterial RNA polymerase elongation complex with NusG and CTP

PDB-8urw: 
Cyanobacterial RNA polymerase elongation complex with NusG and CTP

EMDB-36905: 
SARS-CoV-2 BA.1 RBD with UT28-RD
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
