-検索条件
-検索結果
検索 (著者・登録者: lei & m)の結果3,911件中、1から50件目までを表示しています

EMDB-43246: 
TehA from Haemophilus influenzae purified in DDM

EMDB-43247: 
TehA from Haemophilus influenzae purified in GDN

EMDB-43248: 
TehA from Haemophilus influenzae purified in LMNG

EMDB-43249: 
TehA from Haemophilus influenzae purified in OG

PDB-8vi2: 
TehA from Haemophilus influenzae purified in DDM

PDB-8vi3: 
TehA from Haemophilus influenzae purified in GDN

PDB-8vi4: 
TehA from Haemophilus influenzae purified in LMNG

PDB-8vi5: 
TehA from Haemophilus influenzae purified in OG

EMDB-36659: 
Structure of human TRPV4 with antagonist A1

EMDB-36660: 
Structure of human TRPV4 with antagonist GSK279

EMDB-36675: 
Structure of human TRPV4 with antagonist A2

EMDB-36676: 
Structure of human TRPV4 with antagonist A2 and RhoA

PDB-8ju5: 
Structure of human TRPV4 with antagonist A1

PDB-8ju6: 
Structure of human TRPV4 with antagonist GSK279

PDB-8jvi: 
Structure of human TRPV4 with antagonist A2

PDB-8jvj: 
Structure of human TRPV4 with antagonist A2 and RhoA

EMDB-42300: 
Structure of the C3bBb-albicin complex

EMDB-42244: 
Complex of C3b with the inhibitor albicin

PDB-8uh2: 
Complex of C3b with the inhibitor albicin
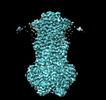
EMDB-40180: 
MsbA bound to cerastecin C

PDB-8gk7: 
MsbA bound to cerastecin C
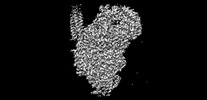
EMDB-37130: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form

EMDB-37131: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in monomeric form

PDB-8kdb: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form

PDB-8kdc: 
Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in monomeric form
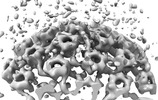
EMDB-18482: 
Herpes simplex virus 1 capsid (WT) vertices in perinuclear NEC-coated vesicles determined in situ

EMDB-18484: 
Herpes simplex virus 1 nuclear egress complex (WT) determined in situ from perinuclear vesicles

EMDB-17766: 
CryoEM structure of Nal1 protein, allele SPIKE, from Oryza sativa japonica group

EMDB-17768: 
CryoEM structure of Nal1 protein, allele IR64, from Oryza sativa indica cultivar

PDB-8pn1: 
CryoEM structure of Nal1 protein, allele SPIKE, from Oryza sativa japonica group

PDB-8pn2: 
CryoEM structure of Nal1 protein, allele IR64, from Oryza sativa indica cultivar
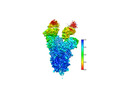
EMDB-37240: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab

EMDB-37241: 
The interface structure of Omicron RBD binding to 5817 Fab

PDB-8khc: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab

PDB-8khd: 
The interface structure of Omicron RBD binding to 5817 Fab

EMDB-17974: 
Pseudorabies virus cytosolic C-capsid (US3 KO) vertices determined in situ

EMDB-17975: 
Pseudorabies virus primary enveloped (perinuclear) C-capsid (US3 KO) vertices determined in situ

EMDB-17976: 
Pseudorabies nuclear C-capsids (US3 KO) vertices determined in situ

EMDB-18473: 
Subtomogram average of pseudorabies virus nuclear egress complex helical form (UL31/34) determined in situ

EMDB-18474: 
Subtomogram average of pseudorabies virus nuclear egress complex (UL31/34) determined in situ

EMDB-18479: 
Pseudorabies virus cytosolic C-capsid (WT) vertices determined in situ

EMDB-18480: 
Pseudorabies virus nuclear C-capsid (WT) vertices determined in situ

EMDB-18481: 
Herpes simplex virus 1 cytosolic C-capsid (WT) vertices determined in situ
EMDB-18483: 
Herpes simplex virus 1 nuclear C-capsid (WT) vertices determined in situ

EMDB-37389: 
cryo-EM structure of native mastigonemes isolated from Chlamydomonas reinhardtii at 3.0 angstrom resolution

PDB-8wa2: 
cryo-EM structure of native mastigonemes isolated from Chlamydomonas reinhardtii at 3.0 angstrom resolution

EMDB-19856: 
Focused map 1- K48-linked ubiquitin chain formation with a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB~acceptor UB-SIL1 peptide

EMDB-19857: 
Focused map 2 - K48-linked ubiquitin chain formation with a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB~acceptor UB-SIL1 peptide

EMDB-19858: 
Focused map 3 - K48-linked ubiquitin chain formation with a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB~acceptor UB-SIL1 peptide

EMDB-19859: 
Focused map 4 - K48-linked ubiquitin chain formation with a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB~acceptor UB-SIL1 peptide
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
