-検索条件
-検索結果
検索 (著者・登録者: kossiakoff & aa)の結果89件中、1から50件目までを表示しています
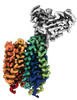
EMDB-43683: 
Cryo-EM structure of FLVCR2 in the inward-facing state with choline bound
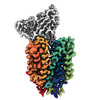
EMDB-43684: 
Cryo-EM structure of FLVCR2 in the outward-facing state with choline bound

EMDB-40622: 
Chlorella virus Hyaluronan Synthase bound to GlcA extended GlcNAc primer

EMDB-40623: 
Chlorella virus Hyaluronan Synthase bound to GlcNAc primer and UDP-GlcA

EMDB-40624: 
Chlorella virus Hyaluronan Synthase bound to GlcA extended GlcNAc primer and UDP

EMDB-40591: 
Xenopus laevis hyaluronan synthase 1

EMDB-40594: 
Xenopus laevis hyaluronan synthase 1, nascent HA polymer bound state
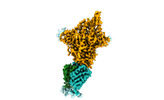
EMDB-40598: 
Xenopus laevis hyaluronan synthase 1, UDP-bound, gating loop inserted state

EMDB-19440: 
Cryo-EM structure of human NTCP-Bulevirtide complex

EMDB-41255: 
Cryo-EM structure of Pseudomonas aeruginosa TonB-dependent transporter PhuR in complex with synthetic antibody and heme

EMDB-17655: 
Human OATP1B3

EMDB-17677: 
Human OATP1B1

EMDB-27674: 
LRRC8A:C conformation 2 (oblong) top mask

EMDB-27675: 
LRRC8A:C conformation 2 (oblong) LRR mask

EMDB-27676: 
LRRC8A:C conformation 2 (oblong)

EMDB-27677: 
LRRC8A:C conformation 1 (round) top focus

EMDB-27678: 
LRRC8A:C conformation 1 (round) LRR focus 1

EMDB-27679: 
LRRC8A:C conformation 1 (round) LRR focus 2

EMDB-27681: 
LRRC8A:C conformation 1 (round) LRR focus 3

EMDB-27682: 
LRRC8A:C conformation 1 (round)

EMDB-27686: 
LRRC8A:C in MSPE3D1 nanodisc top focus

EMDB-27687: 
LRRC8A:C in MSP1E3D1 nanodisc

EMDB-28894: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28895: 
LRRC8A(T48D):C conformation 2 LRR focus

EMDB-28897: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28898: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28901: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28903: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28904: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-28905: 
LRRC8A(T48D):C conformation 2 top focus

EMDB-14779: 
Cryo-EM structure of C-mannosyltransferase CeDPY19, in complex with acceptor peptide and bound to CMT2-Fab and anti-Fab nanobody

EMDB-14780: 
Cryo-EM structure of C-mannosyltransferase CeDPY19, in apo state, bound to CMT2-Fab and anti-Fab nanobody

EMDB-14781: 
Cryo-EM structure of C-mannosyltransferase CeDPY19, in complex with Dol25-P-Man and bound to CMT2-Fab and anti-Fab nanobody

EMDB-14782: 
Cryo-EM structure of C-mannosyltransferase CeDPY19, in ternary complex with Dol25-P-C-Man and acceptor peptide, bound to CMT2-Fab and anti-Fab nanobody
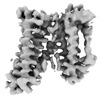
EMDB-24698: 
Cryo-EM structure of the HIV-1 restriction factor human SERINC3

EMDB-24705: 
Human SERINC3-DeltaICL4

EMDB-25836: 
CryoEM Structure of sFab COP-3 Complex with human claudin-4 and Clostridium perfringens enterotoxin C-terminal domain focused map
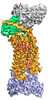
EMDB-25171: 
Cryo-EM structure of ACKR3 in complex with CXCL12, an intracellular Fab, and an extracellular Fab

EMDB-25172: 
Cryo-EM structure of ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ, an intracellular Fab, and an extracellular Fab
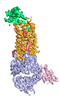
EMDB-25173: 
Cryo-EM structure of ACKR3 in complex with CXCL12 and an intracellular Fab
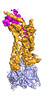
EMDB-25174: 
Cryo-EM structure of human ACKR3 in complex with chemokine N-terminal mutant CXCL12_LRHQ and an intracellular Fab
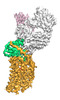
EMDB-25175: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab
EMDB-25176: 
Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, an extracellular Fab, and an intracellular Fab
EMDB-25177: 
Cryo-EM structure of human ACKR3 in complex with a small molecule partial agonist CCX662, and an intracellular Fab

EMDB-15024: 
Structure of the human sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody

EMDB-25750: 
Structure of the peroxisomal retro-translocon formed by a heterotrimeric ubiquitin ligase complex

PDB-7tbl: 
Composite structure of the human nuclear pore complex (NPC) cytoplasmic face generated with a 12A cryo-ET map of the purified HeLa cell NPC

PDB-7tbm: 
Composite structure of the dilated human nuclear pore complex (NPC) generated with a 37A in situ cryo-ET map of CD4+ T cell NPC
EMDB-26054: 
Single-Particle Cryo-EM Structure of the WaaL O-antigen ligase in its ligand bound state
EMDB-26057: 
Single-Particle Cryo-EM Structure of the WaaL O-antigen ligase in its apo state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
