-検索条件
-検索結果
検索 (著者・登録者: jones & ey)の結果76件中、1から50件目までを表示しています

EMDB-29857: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

PDB-8g8w: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

EMDB-29396: 
Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP
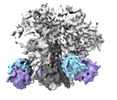
EMDB-29836: 
vFP52.02 Fab in complex with BG505 DS-SOSIP Env trimer

EMDB-29880: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 1)

EMDB-29881: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 2)

EMDB-29882: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 3)

EMDB-29905: 
vFP48.02 Fab in complex with BG505 DS-SOSIP Env trimer

PDB-8fr6: 
Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP

PDB-8g85: 
vFP52.02 Fab in complex with BG505 DS-SOSIP Env trimer

PDB-8g9w: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 1)

PDB-8g9x: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 2)

PDB-8g9y: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 3)

PDB-8gas: 
vFP48.02 Fab in complex with BG505 DS-SOSIP Env trimer

EMDB-27385: 
Oligomeric C9 in complex with aE11 Fab

EMDB-28863: 
PolyC9 in complex with aE11 Fab

PDB-8de6: 
Oligomeric C9 in complex with aE11 Fab
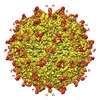
EMDB-25606: 
Zika Virus particle bound with IgM antibody DH1017 Fab fragment

PDB-7t17: 
Zika Virus asymmetric unit bound with IgM antibody DH1017 Fab fragment

EMDB-23521: 
Prefusion RSV F glycoprotein bound by neutralizing site V-directed antibody ADI-14442

PDB-7lue: 
Prefusion RSV F glycoprotein bound by neutralizing site V-directed antibody ADI-14442

EMDB-23520: 
Cryo-EM structure of RSV preF bound by Fabs 32.4K and 01.4B

PDB-7luc: 
Cryo-EM structure of RSV preF bound by Fabs 32.4K and 01.4B

PDB-7l6o: 
Cryo-EM structure of HIV-1 Env CH848.3.D0949.10.17chim.6R.DS.SOSIP.664

EMDB-23518: 
Cryo-EM map of DH851.3 bound to HIV-1 CH505 Env

PDB-7lu9: 
Cryo-EM structure of DH851.3 bound to HIV-1 CH505 Env

EMDB-23519: 
Cryo-EM map of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer

PDB-7lua: 
Cryo-EM structure of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer

EMDB-23152: 
Cryo-electron microscopy reconstruction of antibody DH898.1 Fab-dimer bound to glycans 332, 392, and 396 of HIV Env CH848 10.17 SOSIP trimer

EMDB-23153: 
Cryo-electron microscopy local refinement of antibody DH898.1 Fab-dimer bound to glycans 332, 392, and 396 of HIV Env CH848 10.17 SOSIP trimer

EMDB-23149: 
Cryo-electron microscopy reconstruction of antibody DH898.1 Fab-dimer bound near the CD4 binding site of HIV Env SOSIP trimer CH848 10.17

EMDB-11938: 
Ternary complex of full-length Caspase-8 and FADD

EMDB-11939: 
Central region of Caspase-8:FADD ternary complex

EMDB-11940: 
Ternary complex of Full length Caspase-8 with FADD and FLIPs

EMDB-11941: 
CryoEM analysis of ternary complex of full-length Caspase-8 with FADD and FLIPs

EMDB-23124: 
Cryo-electron microcospy reconstruction of CH848.3.D0949.10.17chim.6R.DS.SOSIP.664 HIV Env

EMDB-23145: 
Cryo-electron microscopy reconstruction of locally refined antibody DH898.1 Fab-dimer

PDB-7l6m: 
Cryo-EM structure of DH898.1 Fab-dimer from local refinement of the Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer

EMDB-23156: 
SARS-CoV 2 Spike Protein bound to LY-CoV555

PDB-7l3n: 
SARS-CoV 2 Spike Protein bound to LY-CoV555

EMDB-23094: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to one copy of domain-swapped antibody 2G12

EMDB-23095: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to two copies of domain-swapped antibody 2G12

EMDB-23097: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound domain-swapped antibody 2G12 from masked 3D refinement

PDB-7l02: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to one copy of domain-swapped antibody 2G12

PDB-7l06: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound to two copies of domain-swapped antibody 2G12

PDB-7l09: 
Cryo-EM structure of SARS-CoV-2 2P S ectodomain bound domain-swapped antibody 2G12 from masked 3D refinement

EMDB-10242: 
Structure of Coxsackievirus A10

EMDB-10256: 
Structure of Coxsackievirus A10 A-particle

EMDB-10263: 
Structure of Coxsackievirus A10 complexed with its receptor KREMEN1

EMDB-20616: 
EM structure of MPEG-1(w.t.) soluble pre-pore
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
