-検索条件
-検索結果
検索 (著者・登録者: elias & p)の結果65件中、1から50件目までを表示しています

EMDB-18214: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly
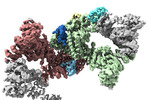
EMDB-18216: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer
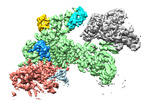
EMDB-18217: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused on E2-like density

EMDB-18218: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused dimeric core
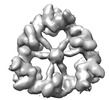
EMDB-18220: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain
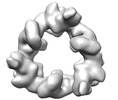
EMDB-18221: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 DOC domain
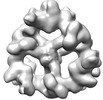
EMDB-18222: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARM9 domain
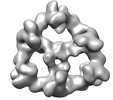
EMDB-18223: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 ARIH-RBR element

EMDB-19179: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated conformation - symmetry expanded unneddylated dimer

PDB-8q7e: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly

PDB-8q7h: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer

PDB-8rhz: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated conformation - symmetry expanded unneddylated dimer

EMDB-17014: 
Consensus map of HSV-1 DNA polymerase-processivity factor complex in pre-translocation state

EMDB-17013: 
HSV-1 DNA polymerase-processivity factor complex in halted elongation state consensus map

EMDB-17018: 
Consensus map of HSV-1 DNA polymerase-processivity factor complex in exonuclease state

EMDB-16918: 
Focused refinement map of HSV-1 DNA polymerase in pre-translocation state

EMDB-16919: 
Focused refinement map of HSV-1 DNA polymerase processivity factor in pre-translocation state

EMDB-16924: 
Focused refinement of HSV-1 DNA polymerase in halted elongation state

EMDB-16925: 
Focused refinement of HSV-1 DNA polymerase processivity factor in halted elongation state

EMDB-16927: 
Focused refinement map of HSV-1 DNA polymerase in exonuclease state

EMDB-16928: 
Focused refinement map of HSV-1 DNA polymerase processivity factor in exonuclease state

EMDB-16906: 
HSV-1 DNA polymerase-processivity factor complex in pre-translocation state

EMDB-16907: 
HSV-1 DNA polymerase-processivity factor complex in halted elongation state

EMDB-16909: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state

EMDB-16910: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state active site

EMDB-16911: 
HSV-1 DNA polymerase active site in alternative exonuclease state

EMDB-16912: 
HSV-1 DNA polymerase beta-hairpin loop

PDB-8oj6: 
HSV-1 DNA polymerase-processivity factor complex in pre-translocation state

PDB-8oj7: 
HSV-1 DNA polymerase-processivity factor complex in halted elongation state

PDB-8oja: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state

PDB-8ojb: 
HSV-1 DNA polymerase-processivity factor complex in exonuclease state active site

PDB-8ojc: 
HSV-1 DNA polymerase active site in alternative exonuclease state

PDB-8ojd: 
HSV-1 DNA polymerase beta-hairpin loop

EMDB-27382: 
Helical rods of far-red light-absorbing allophycocyanin in Synechococcus sp.

PDB-8ddy: 
Helical rods of far-red light-absorbing allophycocyanin in Synechococcus sp.
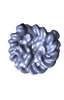
EMDB-35060: 
Clr4-H3K9 Nucleosome complex

EMDB-12142: 
ASCT2 in the presence of the inhibitor Lc-BPE in the outward-open conformation.

EMDB-12143: 
ASCT2 in the presence of the inhibitor ERA-21 in the outward-open conformation.

PDB-7bcq: 
ASCT2 in the presence of the inhibitor Lc-BPE (position "up") in the outward-open conformation.

PDB-7bcs: 
ASCT2 in the presence of the inhibitor Lc-BPE (position "down") in the outward-open conformation.

PDB-7bct: 
ASCT2 in the presence of the inhibitor ERA-21 in the outward-open conformation.

EMDB-24195: 
Complex structure of HIV superinfection Fab QA013.2 and BG505.SOSIP.664

PDB-7n65: 
Complex structure of HIV superinfection Fab QA013.2 and BG505.SOSIP.664

PDB-7ko8: 
Cryo-EM structure of the mature and infective Mayaro virus

EMDB-22961: 
Cryo-EM structure of the mature and infective Mayaro virus

EMDB-11050: 
Mec1-Ddc2 (F2244L mutant) in complex with Mg AMP-PNP

EMDB-11051: 
Mec1-Ddc2 (F2244L mutant) in complex with Mg AMP-PNP (State II)

EMDB-11055: 
Mec1-Ddc2 (wild-type) in complex with AMP-PNP

EMDB-11056: 
Mec1-Ddc2 (wild-type) apo

PDB-6z2w: 
Mec1-Ddc2 (F2244L mutant) in complex with Mg AMP-PNP
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
