-検索条件
-検索結果
検索 (著者・登録者: bueler & sa)の結果79件中、1から50件目までを表示しています

EMDB-27984: 
Yeast VO in complex with Vma12-22p

EMDB-27985: 
Yeast VO missing subunits a, e, and f in complex with Vma12-22p

EMDB-27986: 
Yeast VO in complex with Vma21p

EMDB-27987: 
YAR027W and YAR028W in complex with c subunits from yeast VO complex

EMDB-27988: 
Yeast VO in complex with Vma12-22p purified via Vma21p-3xFLAG

EMDB-27963: 
Mycobacterial respiratory complex I with both quinone positions modelled
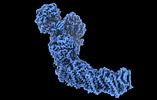
EMDB-27964: 
Mycobacterial respiratory complex I, fully-inserted quinone
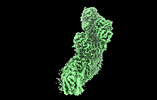
EMDB-27965: 
Mycobacterial respiratory complex I, semi-inserted quinone

EMDB-26385: 
Structure of porcine V-ATPase with mEAK7 and SidK, Rotary state 2

EMDB-26386: 
Structure of porcine kidney V-ATPase with SidK, Rotary State 1

EMDB-26387: 
Structure of porcine kidney V-ATPase with SidK, Rotary State 2

EMDB-26388: 
Structure of porcine kidney V-ATPase with SidK, Rotary State 3

EMDB-26002: 
V-ATPase from Saccharomyces cerevisiae, State 3

PDB-7tmt: 
V-ATPase from Saccharomyces cerevisiae, State 3

EMDB-26000: 
V-ATPase from Saccharomyces cerevisiae, State 1

EMDB-26001: 
V-ATPase from Saccharomyces cerevisiae, State 2

PDB-7tmr: 
V-ATPase from Saccharomyces cerevisiae, State 1

PDB-7tms: 
V-ATPase from Saccharomyces cerevisiae, State 2

EMDB-25996: 
Complete V1 Complex from Saccharomyces cerevisiae

EMDB-25997: 
V1 complex lacking subunit C from Saccharomyces cerevisiae, State 1

EMDB-25998: 
V1 complex lacking subunit C from Saccharomyces cerevisiae, State 2

EMDB-25999: 
V1 complex lacking subunit C from Saccharomyces cerevisiae, State 3

PDB-7tmm: 
Complete V1 Complex from Saccharomyces cerevisiae

PDB-7tmo: 
V1 complex lacking subunit C from Saccharomyces cerevisiae, State 1

PDB-7tmp: 
V1 complex lacking subunit C from Saccharomyces cerevisiae, State 2

PDB-7tmq: 
V1 complex lacking subunit C from Saccharomyces cerevisiae, State 3

EMDB-24455: 
Mycobacterial CIII2CIV2 supercomplex, Inhibitor free

EMDB-24456: 
Mycobacterial CIII2CIV2 supercomplex, inhibitor free, -Lpqe cyt cc open

EMDB-24457: 
Mycobacterial CIII2CIV2 supercomplex, Telacebec (Q203) bound

EMDB-22738: 
CryoEM reconstruction of SARS-CoV-2 receptor binding domain in complex with the Fab fragment of neutralizing antibody 46

EMDB-22739: 
CryoEM reconstruction of SARS-CoV-2 Spike in complex with the Fab fragment of neutralizing antibody 80.

EMDB-22740: 
CryoEM reconstruction of SARS-CoV-2 Spike in complex with the Fab fragment of neutralizing antibody 298

EMDB-22741: 
CryoEM reconstruction of SARS-CoV-2 Spike in complex with the Fab fragment of neutralizing antibody 324

EMDB-22311: 
Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase rotational state 1

EMDB-22312: 
Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase rotational state 2

EMDB-22313: 
Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase rotational state 3

EMDB-22314: 
Cryo-EM structure of bedaquiline-saturated Mycobacterium smegmatis ATP synthase rotational state 1

EMDB-22315: 
Cryo-EM structure of bedaquiline-saturated Mycobacterium smegmatis ATP synthase rotational state 2

EMDB-22316: 
Cryo-EM structure of bedaquiline-saturated Mycobacterium smegmatis ATP synthase rotational state 3

EMDB-22317: 
Cryo-EM structure of bedaquiline-washed Mycobacterium smegmatis ATP synthase rotational state 1

EMDB-22318: 
Cryo-EM structure of bedaquiline-washed Mycobacterium smegmatis ATP synthase rotational state 2

EMDB-22319: 
Cryo-EM structure of bedaquiline-washed Mycobacterium smegmatis ATP synthase rotational state 3
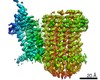
EMDB-22320: 
Cryo-EM structure of bedaquiline-free Mycobacterium smegmatis ATP synthase FO region

EMDB-22321: 
Cryo-EM structure of bedaquiline-saturated Mycobacterium smegmatis ATP synthase FO region

EMDB-22322: 
Cryo-EM structure of bedaquiline-washed Mycobacterium smegmatis ATP synthase FO region

EMDB-21317: 
Mammalian V-ATPase from rat brain with the Legionella pneumophila effector protein SidK - rotational state 1 non-uniform refinement

EMDB-21318: 
Mammalian V-ATPase from rat brain with the Legionella pneumophila effector protein SidK - rotational state 2 non-uniform refinement

EMDB-21319: 
Mammalian V-ATPase from rat brain with the Legionella pneumophila effector protein SidK - rotational state 3 non-uniform refinement

EMDB-21345: 
Mammalian V-ATPase from rat brain soluble V1 region rotational state 1 with SidK and ADP (from focused refinement)

EMDB-21346: 
Mammalian V-ATPase from rat brain soluble V1 region rotational state 2 with SidK and ADP (from focused refinement)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
