-検索条件
-検索結果
検索 (著者・登録者: allen & er)の結果395件中、1から50件目までを表示しています

EMDB-42301: 
Cryo-EM Structure of Human Ninjurin1 curved oligomer

PDB-8uip: 
Cryo-EM Structure of Human Ninjurin1 curved oligomer
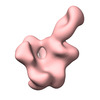
EMDB-42464: 
chEnv TTT protein in complex with 43A2 Fab

EMDB-42468: 
chEnv TTT protein in complex with CM01A Fab
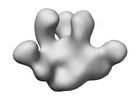
EMDB-43664: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines
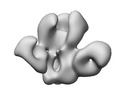
EMDB-43665: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (cH125 TTT)

EMDB-43666: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (H2/1 GCN4)

EMDB-43668: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines (H5/1 GCN4)

EMDB-43669: 
Triple tandem trimer immunogens for HIV-1 and influenza nucleic acid-based vaccines. H5 GCN4

EMDB-43482: 
Density map for gamma tubulin ring complex capped microtubule

EMDB-43483: 
Refined density map of gamma tubulin ring complex capped microtubule

EMDB-43519: 
Structure of the gamma tubulin ring complex nucleated microtubule protofilament.

PDB-8vrj: 
Rigid body fitted model for gamma tubulin ring complex capped microtubule

PDB-8vrk: 
Rigid body fitted model for refined density map of gamma tubulin ring complex capped microtubule

PDB-8vt7: 
Structure of the gamma tubulin ring complex nucleated microtubule protofilament.

EMDB-41711: 
Disulfide-stabilized HIV-1 CA hexamer in complex with PQBP1 Nt

PDB-8ty6: 
Disulfide-stabilized HIV-1 CA hexamer in complex with PQBP1 Nt

EMDB-18586: 
Cryo-EM reconstruction of crosslinked native Bacillus subtilis collided disome binding MutS2 SMR and KOW domains

EMDB-18585: 
Cryo-EM reconstruction of Bacillus subtilis collided disome binding MutS2 SMR and KOW domains

EMDB-18901: 
Bacillus subtilis MutS2-collided disome complex (stalled 70S)

PDB-8r55: 
Bacillus subtilis MutS2-collided disome complex (collided 70S)

EMDB-18558: 
Bacillus subtilis MutS2-collided disome complex (stalled 70S)

PDB-8qpp: 
Bacillus subtilis MutS2-collided disome complex (stalled 70S)

EMDB-29227: 
cryoEM structure of a broadly neutralizing antibody STI-9167

EMDB-29172: 
Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate

PDB-8fhw: 
Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate

EMDB-28537: 
cryoEM structure of a broadly neutralizing anti-SARS-CoV-2 antibody STI-9167

PDB-8eqf: 
cryoEM structure of a broadly neutralizing anti-SARS-CoV-2 antibody STI-9167

EMDB-40803: 
BG505 Boost2 SOSIP.664 in complex with NHP polyclonal antibody FP2

EMDB-40804: 
BG505 Boost2 SOSIP.664 in complex with NHP Polyclonal Antibody FP4

EMDB-40805: 
BG505 Boost 2 in complex with NHP antibody V1V2

EMDB-40806: 
BG505 Boost 2 in complex with NHP Polyclonal Antibody N625

EMDB-40807: 
BG505 Boost2 SOSIP.664 in complex with NHP polyclonal antibody Base1

EMDB-40808: 
Boost 2 SOSIP.664 in complex with NHP polyclonal antibody Base2

EMDB-40809: 
BG505 Boost2 SOSIP.664 in complex with NHP polyclonal antibody Base3

EMDB-40810: 
BG505 Boost2 SOSIP.664 in complex with NHP polyclonal antibody FP1

PDB-8sw7: 
BG505 Boost2 SOSIP.664 in complex with NHP polyclonal antibody FP1
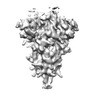
EMDB-16403: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C3 symmetry
EMDB-16404: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C1 symmetry

EMDB-16405: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E) in C3 symmetry

EMDB-16406: 
Subtomogram averaging of a coronavirus spike in situ (ChAdOx1 19E) in C1 symmetry

EMDB-16697: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C1 symmetry after 3D classification

EMDB-28540: 
BG505 UFO-E2p-L4P nanoparticle reconstructed by focused refinement with a mask around the nanoparticle core
EMDB-28541: 
BG505 UFO trimer map reconstructed from BG505 UFO-E2p-L4P nanoparticle by localized reconstruction

EMDB-28542: 
BG505 UFO-10GS-I3-01v9-L7P nanoparticle reconstructed by focused refinement with a mask around the nanoparticle core

EMDB-28543: 
BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction

PDB-8eqn: 
BG505 UFO-E2p-L4P nanoparticle reconstructed by focused refinement with a mask around the nanoparticle core

EMDB-14888: 
CM01B in complex with ConM SOSIP

EMDB-14889: 
CM05A1 in complex with conM SOSIP

EMDB-14890: 
CM02A in complex with conM SOSIP
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
