-検索条件
-検索結果
検索 (著者・登録者: alessandro & costa)の結果76件中、1から50件目までを表示しています

EMDB-18191: 
X. laevis CMG dimer bound to dimeric DONSON - without ATPase

EMDB-18192: 
X. laevis CMG dimer bound to dimeric DONSON - MCM ATPase

EMDB-18195: 
Single CMG purified from replicating Xenopus egg extracts

PDB-8q6o: 
X. laevis CMG dimer bound to dimeric DONSON - without ATPase

PDB-8q6p: 
X. laevis CMG dimer bound to dimeric DONSON - MCM ATPase
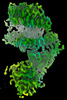
EMDB-29530: 
SARS-CoV-2 XBB.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment
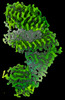
EMDB-29531: 
SARS-CoV-2 BQ.1.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment
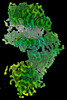
EMDB-40240: 
SARS-CoV-2 BN.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment

PDB-8fxb: 
SARS-CoV-2 XBB.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment

PDB-8fxc: 
SARS-CoV-2 BQ.1.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment

PDB-8s9g: 
SARS-CoV-2 BN.1 spike RBD bound to the human ACE2 ectodomain and the S309 neutralizing antibody Fab fragment

EMDB-13978: 
S. cerevisiae CMGE nucleating origin DNA melting

EMDB-13988: 
S. cerevisiae CMGE dimer nucleating origin DNA melting

EMDB-14439: 
S. cerevisiae CMGE dimer nucleating origin DNA melting

PDB-7qhs: 
S. cerevisiae CMGE nucleating origin DNA melting

PDB-7z13: 
S. cerevisiae CMGE dimer nucleating origin DNA melting

EMDB-14860: 
Cryo-EM structure of the MVV CSC intasome at 4.5A resolution

PDB-7zpp: 
Cryo-EM structure of the MVV CSC intasome at 4.5A resolution

EMDB-13176: 
3.0 A resolution structure of a DNA-loaded MCM double hexamer

EMDB-13211: 
Structure of a DNA-loaded MCM double hexamer engaged with the Dbf4-dependent kinase

PDB-7p30: 
3.0 A resolution structure of a DNA-loaded MCM double hexamer

PDB-7p5z: 
Structure of a DNA-loaded MCM double hexamer engaged with the Dbf4-dependent kinase

EMDB-11220: 
Cryo-EM structure of the regulatory linker of ALC1 bound to the nucleosome's acidic patch: nucleosome class.

EMDB-11221: 
Cryo-EM structure of the regulatory linker of ALC1 bound to the nucleosome's acidic patch: hexasome class.

PDB-6zhx: 
Cryo-EM structure of the regulatory linker of ALC1 bound to the nucleosome's acidic patch: nucleosome class.

PDB-6zhy: 
Cryo-EM structure of the regulatory linker of ALC1 bound to the nucleosome's acidic patch: hexasome class.
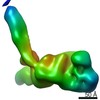
EMDB-10870: 
Cohesin complex with loader gripping DNA

EMDB-10930: 
Cohesin complex with loader gripping DNA

PDB-6yuf: 
Cohesin complex with loader gripping DNA

EMDB-10744: 
Microtubule Nucleation by Single Human gamma-TuRC in a Partly Open Asymmetric Conformation

EMDB-4980: 
Cryo-EM structure of an MCM loading intermediate

PDB-6rqc: 
Cryo-EM structure of an MCM loading intermediate

EMDB-4692: 
Human-D02 Nucleosome Core Particle with biotin-streptavidin label

EMDB-4693: 
Structure of a human nucleosome at 3.5 A resolution

EMDB-4960: 
PFV intasome - nucleosome strand transfer complex

PDB-6r0c: 
Human-D02 Nucleosome Core Particle with biotin-streptavidin label

PDB-6rny: 
PFV intasome - nucleosome strand transfer complex

EMDB-4788: 
D. melanogaster CMG-DNA with ATP, State 2B

PDB-6raz: 
D. melanogaster CMG-DNA, State 2B

EMDB-4785: 
D. melanogaster CMG-DNA with ATP, State 1A

EMDB-4786: 
D. melanogaster CMG-DNA with ATP, State 1B

EMDB-4787: 
D. melanogaster CMG-DNA with ATP, State 2A

PDB-6raw: 
D. melanogaster CMG-DNA, State 1A

PDB-6rax: 
D. melanogaster CMG-DNA, State 1B

PDB-6ray: 
D. melanogaster CMG-DNA, State 2A

EMDB-4958: 
Negative stain EM 3D reconstruction of the UvrA-UvrB-DNA complex.

EMDB-0287: 
Cryo-EM structure of S. cerevisiae Polymerase epsilon deltacat mutant

EMDB-0288: 
S. cerevisiae CMG-Pol epsilon-DNA

PDB-6hv8: 
Cryo-EM structure of S. cerevisiae Polymerase epsilon deltacat mutant

PDB-6hv9: 
S. cerevisiae CMG-Pol epsilon-DNA
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
