+ データを開く
データを開く
- 基本情報
基本情報
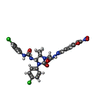
| 登録情報 | データベース: PDB / ID: 8do3 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
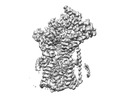
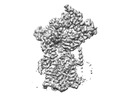
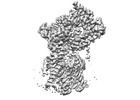
| タイトル | Cryo-EM structure of the human Sec61 complex inhibited by eeyarestatin I | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | PROTEIN TRANSPORT/INHIBITOR /  translocon (トランスロコン) / translocon (トランスロコン) /  inhibitor (酵素阻害剤) / inhibitor (酵素阻害剤) /  protein translocation / protein translocation /  PROTEIN TRANSPORT / PROTEIN TRANSPORT-INHIBITOR complex PROTEIN TRANSPORT / PROTEIN TRANSPORT-INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endoplasmic reticulum Sec complex / endoplasmic reticulum quality control compartment / pronephric nephron development / cotranslational protein targeting to membrane / Ssh1 translocon complex / Sec61 translocon complex / protein targeting to ER / protein insertion into ER membrane /  post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation ...endoplasmic reticulum Sec complex / endoplasmic reticulum quality control compartment / pronephric nephron development / cotranslational protein targeting to membrane / Ssh1 translocon complex / Sec61 translocon complex / protein targeting to ER / protein insertion into ER membrane / post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation ...endoplasmic reticulum Sec complex / endoplasmic reticulum quality control compartment / pronephric nephron development / cotranslational protein targeting to membrane / Ssh1 translocon complex / Sec61 translocon complex / protein targeting to ER / protein insertion into ER membrane /  post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation / post-translational protein targeting to endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane, translocation /  signal sequence binding / SRP-dependent cotranslational protein targeting to membrane / signal sequence binding / SRP-dependent cotranslational protein targeting to membrane /  post-translational protein targeting to membrane, translocation / endoplasmic reticulum organization / retrograde protein transport, ER to cytosol / post-translational protein targeting to membrane, translocation / endoplasmic reticulum organization / retrograde protein transport, ER to cytosol /  epidermal growth factor binding / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane / protein transmembrane transporter activity / : / epidermal growth factor binding / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / SRP-dependent cotranslational protein targeting to membrane / protein transmembrane transporter activity / : /  calcium channel activity / calcium channel activity /  ribosome binding / ER-Phagosome pathway / endoplasmic reticulum membrane / ribosome binding / ER-Phagosome pathway / endoplasmic reticulum membrane /  小胞体 / 小胞体 /  RNA binding / RNA binding /  生体膜 / 生体膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.22 Å クライオ電子顕微鏡法 / 解像度: 3.22 Å | |||||||||
 データ登録者 データ登録者 | Park, E. / Itskanov, S. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: A common mechanism of Sec61 translocon inhibition by small molecules. 著者: Samuel Itskanov / Laurie Wang / Tina Junne / Rumi Sherriff / Li Xiao / Nicolas Blanchard / Wei Q Shi / Craig Forsyth / Dominic Hoepfner / Martin Spiess / Eunyong Park /    要旨: The Sec61 complex forms a protein-conducting channel in the endoplasmic reticulum membrane that is required for secretion of soluble proteins and production of many membrane proteins. Several natural ...The Sec61 complex forms a protein-conducting channel in the endoplasmic reticulum membrane that is required for secretion of soluble proteins and production of many membrane proteins. Several natural and synthetic small molecules specifically inhibit Sec61, generating cellular effects that are useful for therapeutic purposes, but their inhibitory mechanisms remain unclear. Here we present near-atomic-resolution structures of human Sec61 inhibited by a comprehensive panel of structurally distinct small molecules-cotransin, decatransin, apratoxin, ipomoeassin, mycolactone, cyclotriazadisulfonamide and eeyarestatin. All inhibitors bind to a common lipid-exposed pocket formed by the partially open lateral gate and plug domain of Sec61. Mutations conferring resistance to the inhibitors are clustered at this binding pocket. The structures indicate that Sec61 inhibitors stabilize the plug domain in a closed state, thereby preventing the protein-translocation pore from opening. Our study provides the atomic details of Sec61-inhibitor interactions and the structural framework for further pharmacological studies and drug design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8do3.cif.gz 8do3.cif.gz | 111.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8do3.ent.gz pdb8do3.ent.gz | 80.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8do3.json.gz 8do3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/do/8do3 https://data.pdbj.org/pub/pdb/validation_reports/do/8do3 ftp://data.pdbj.org/pub/pdb/validation_reports/do/8do3 ftp://data.pdbj.org/pub/pdb/validation_reports/do/8do3 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  27589MC  8dnvC  8dnwC  8dnxC  8dnyC  8dnzC  8do0C  8do1C  8do2C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  Protein targeting Protein targeting分子量: 7752.325 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SEC61G / プラスミド: pFastBac / 細胞株 (発現宿主): Sf9 Homo sapiens (ヒト) / 遺伝子: SEC61G / プラスミド: pFastBac / 細胞株 (発現宿主): Sf9発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P60059 |
|---|---|
| #2: タンパク質 |  Protein targeting Protein targeting分子量: 9987.456 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SEC61B Homo sapiens (ヒト) / 遺伝子: SEC61B発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P60468 |
| #3: タンパク質 |  Protein targeting / Sec61 alpha-1 Protein targeting / Sec61 alpha-1分子量: 52202.438 Da / 分子数: 1 Mutation: V263L, D264E, K268R, A270T, R271K, Y272V, Y276I, N277G, T278I, L394F, E346D, Q348G, M401I, R402N, H404K, M409I, V410Y, H411R 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SEC61A1, SEC61A / プラスミド: pFastBac / 細胞株 (発現宿主): Sf9 Homo sapiens (ヒト) / 遺伝子: SEC61A1, SEC61A / プラスミド: pFastBac / 細胞株 (発現宿主): Sf9発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P61619 |
| #4: 化合物 | ChemComp-SWR / |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: A human-yeast chimeric Sec complex treated with eeyarestatin I タイプ: COMPLEX / Entity ID: #1-#3 / 由来: RECOMBINANT | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / Organelle Homo sapiens (ヒト) / Organelle : endoplasmic reticulum : endoplasmic reticulum | ||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)株: Sf9 / プラスミド  : pFastBac : pFastBac | ||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||
| 試料 | 濃度: 10 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: Reconsitituted into a peptidisc. Monodisperse peak from a Superose 6 column. | ||||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: blot for 4 seconds before plunging |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 1600 nm / 最小 デフォーカス(公称値): 800 nm / Cs Bright-field microscopy / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 1600 nm / 最小 デフォーカス(公称値): 800 nm / Cs : 2.7 mm / アライメント法: COMA FREE : 2.7 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
| 電子光学装置 | エネルギーフィルター名称 : GIF Quantum LS / エネルギーフィルタースリット幅: 20 eV : GIF Quantum LS / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 539081 | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.22 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 211735 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 61.04 Å2 | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj