+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8c5d | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Glutathione transferase P1-1 from Mus musculus | ||||||
 要素 要素 | Glutathione S-transferase P 1 | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  multidrug resistance (多剤耐性) / multidrug resistance (多剤耐性) /  pesticide (農薬) / pesticide (農薬) /  enzyme inhibition (酵素阻害剤) / enzyme inhibition (酵素阻害剤) /  drug design (医薬品設計) drug design (医薬品設計) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to cell-matrix adhesion / negative regulation of neutrophil aggregation / negative regulation of peroxidase activity / Paracetamol ADME /  薬物代謝 / negative regulation of smooth muscle cell chemotaxis / Detoxification of Reactive Oxygen Species / 薬物代謝 / negative regulation of smooth muscle cell chemotaxis / Detoxification of Reactive Oxygen Species /  S-nitrosoglutathione binding / kinase regulator activity / response to L-ascorbic acid ...cellular response to cell-matrix adhesion / negative regulation of neutrophil aggregation / negative regulation of peroxidase activity / Paracetamol ADME / S-nitrosoglutathione binding / kinase regulator activity / response to L-ascorbic acid ...cellular response to cell-matrix adhesion / negative regulation of neutrophil aggregation / negative regulation of peroxidase activity / Paracetamol ADME /  薬物代謝 / negative regulation of smooth muscle cell chemotaxis / Detoxification of Reactive Oxygen Species / 薬物代謝 / negative regulation of smooth muscle cell chemotaxis / Detoxification of Reactive Oxygen Species /  S-nitrosoglutathione binding / kinase regulator activity / response to L-ascorbic acid / negative regulation of biosynthetic process / TRAF2-GSTP1 complex / dinitrosyl-iron complex binding / common myeloid progenitor cell proliferation / S-nitrosoglutathione binding / kinase regulator activity / response to L-ascorbic acid / negative regulation of biosynthetic process / TRAF2-GSTP1 complex / dinitrosyl-iron complex binding / common myeloid progenitor cell proliferation /  organic cyclic compound binding / hepoxilin biosynthetic process / glutathione derivative biosynthetic process / oligodendrocyte development / negative regulation of nitric-oxide synthase biosynthetic process / negative regulation of JUN kinase activity / linoleic acid metabolic process / negative regulation of leukocyte proliferation / negative regulation of monocyte chemotactic protein-1 production / JUN kinase binding / organic cyclic compound binding / hepoxilin biosynthetic process / glutathione derivative biosynthetic process / oligodendrocyte development / negative regulation of nitric-oxide synthase biosynthetic process / negative regulation of JUN kinase activity / linoleic acid metabolic process / negative regulation of leukocyte proliferation / negative regulation of monocyte chemotactic protein-1 production / JUN kinase binding /  glutathione peroxidase activity / cellular response to glucocorticoid stimulus / negative regulation of stress-activated MAPK cascade / negative regulation of interleukin-1 beta production / regulation of stress-activated MAPK cascade / prostaglandin metabolic process / negative regulation of vascular associated smooth muscle cell proliferation / glutathione peroxidase activity / cellular response to glucocorticoid stimulus / negative regulation of stress-activated MAPK cascade / negative regulation of interleukin-1 beta production / regulation of stress-activated MAPK cascade / prostaglandin metabolic process / negative regulation of vascular associated smooth muscle cell proliferation /  グルタチオン-S-トランスフェラーゼ / negative regulation of acute inflammatory response / グルタチオン-S-トランスフェラーゼ / negative regulation of acute inflammatory response /  glutathione transferase activity / negative regulation of tumor necrosis factor production / glutathione transferase activity / negative regulation of tumor necrosis factor production /  toxic substance binding / response to amino acid / animal organ regeneration / glutathione metabolic process / negative regulation of canonical NF-kappaB signal transduction / negative regulation of fibroblast proliferation / cellular response to epidermal growth factor stimulus / Neutrophil degranulation / xenobiotic metabolic process / response to nutrient levels / regulation of ERK1 and ERK2 cascade / response to reactive oxygen species / positive regulation of superoxide anion generation / negative regulation of MAP kinase activity / toxic substance binding / response to amino acid / animal organ regeneration / glutathione metabolic process / negative regulation of canonical NF-kappaB signal transduction / negative regulation of fibroblast proliferation / cellular response to epidermal growth factor stimulus / Neutrophil degranulation / xenobiotic metabolic process / response to nutrient levels / regulation of ERK1 and ERK2 cascade / response to reactive oxygen species / positive regulation of superoxide anion generation / negative regulation of MAP kinase activity /  fatty acid binding / negative regulation of extrinsic apoptotic signaling pathway / negative regulation of ERK1 and ERK2 cascade / response to toxic substance / cellular response to insulin stimulus / response to estradiol / response to ethanol / cellular response to lipopolysaccharide / negative regulation of apoptotic process / fatty acid binding / negative regulation of extrinsic apoptotic signaling pathway / negative regulation of ERK1 and ERK2 cascade / response to toxic substance / cellular response to insulin stimulus / response to estradiol / response to ethanol / cellular response to lipopolysaccharide / negative regulation of apoptotic process /  protein kinase binding / protein-containing complex / protein kinase binding / protein-containing complex /  ミトコンドリア / ミトコンドリア /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.28 Å 分子置換 / 解像度: 1.28 Å | ||||||
 データ登録者 データ登録者 | Papageorgiou, A.C. | ||||||
| 資金援助 | European Union, 1件
| ||||||
 引用 引用 |  ジャーナル: Biomolecules / 年: 2023 ジャーナル: Biomolecules / 年: 2023タイトル: Inhibition Analysis and High-Resolution Crystal Structure of Mus musculus Glutathione Transferase P1-1. 著者: Kupreienko, O. / Pouliou, F. / Konstandinidis, K. / Axarli, I. / Douni, E. / Papageorgiou, A.C. / Labrou, N.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8c5d.cif.gz 8c5d.cif.gz | 321.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8c5d.ent.gz pdb8c5d.ent.gz | 220.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8c5d.json.gz 8c5d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c5/8c5d https://data.pdbj.org/pub/pdb/validation_reports/c5/8c5d ftp://data.pdbj.org/pub/pdb/validation_reports/c5/8c5d ftp://data.pdbj.org/pub/pdb/validation_reports/c5/8c5d | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
|---|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 23635.160 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) / 遺伝子: Gstp1, Gstpib / 発現宿主: Mus musculus (ハツカネズミ) / 遺伝子: Gstp1, Gstpib / 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)参照: UniProt: P19157,  グルタチオン-S-トランスフェラーゼ グルタチオン-S-トランスフェラーゼ |
|---|
-非ポリマー , 6種, 622分子 
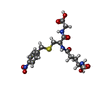









| #2: 化合物 |  グリセリン グリセリン#3: 化合物 | #4: 化合物 | ChemComp-NA / #5: 化合物 | ChemComp-CA / #6: 化合物 | ChemComp-CL / |  塩化物 塩化物#7: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.35 Å3/Da / 溶媒含有率: 47.66 % / 解説: Rectangular rods |
|---|---|
結晶化 | 温度: 289 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8 詳細: PEG 6000 20% (w/v), 0.2 M calcium chloride dihydrate, pH 8.0 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / ビームライン: P13 (MX1) / 波長: 1.033 Å / ビームライン: P13 (MX1) / 波長: 1.033 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2017年6月6日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.033 Å / 相対比: 1 : 1.033 Å / 相対比: 1 |
| 反射 | 解像度: 1.28→56.62 Å / Num. obs: 114851 / % possible obs: 99.7 % / 冗長度: 6.7 % / Biso Wilson estimate: 19.23 Å2 / CC1/2: 0.99 / Rrim(I) all: 0.059 / Net I/σ(I): 12.7 |
| 反射 シェル | 解像度: 1.28→1.3 Å / 冗長度: 6.3 % / Num. unique obs: 5532 / CC1/2: 0.35 / Rrim(I) all: 2.485 / % possible all: 98.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 / 解像度: 1.28→38.68 Å / SU ML: 0.1516 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 23.8494 分子置換 / 解像度: 1.28→38.68 Å / SU ML: 0.1516 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 23.8494 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.1 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 28.22 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.28→38.68 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj














