+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7xxk | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of SARS-CoV-2 N-CTD in complex with GMP | ||||||
 要素 要素 | Nucleoprotein 核タンパク質 核タンパク質 | ||||||
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  Coronavirus (オルトコロナウイルス亜科) / Coronavirus (オルトコロナウイルス亜科) /  C-terminal domain (C末端) / C-terminal domain (C末端) /  nucleocapsid (カプシド) / GMP nucleocapsid (カプシド) / GMP | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoplasmic capsid assembly / viral RNA genome packaging / response to host immune response / negative regulation of interferon-beta production / RNA stem-loop binding / Maturation of nucleoprotein / positive regulation of NLRP3 inflammasome complex assembly / intracellular non-membrane-bounded organelle / CD28 dependent PI3K/Akt signaling / MHC class I protein binding ...cytoplasmic capsid assembly / viral RNA genome packaging / response to host immune response / negative regulation of interferon-beta production / RNA stem-loop binding / Maturation of nucleoprotein / positive regulation of NLRP3 inflammasome complex assembly / intracellular non-membrane-bounded organelle / CD28 dependent PI3K/Akt signaling / MHC class I protein binding / SARS-CoV-2 targets host intracellular signalling and regulatory pathways / molecular condensate scaffold activity / protein sequestering activity / VEGFR2 mediated vascular permeability / TAK1-dependent IKK and NF-kappa-B activation / NOD1/2 Signaling Pathway / DDX58/IFIH1-mediated induction of interferon-alpha/beta / MHC class I protein complex / Interleukin-1 signaling /  カプシド / Interferon alpha/beta signaling / PIP3 activates AKT signaling / Transcription of SARS-CoV-2 sgRNAs / host cell endoplasmic reticulum-Golgi intermediate compartment / host cell Golgi apparatus / viral nucleocapsid / Translation of Structural Proteins / Virion Assembly and Release / host extracellular space / Induction of Cell-Cell Fusion / Attachment and Entry / host cell perinuclear region of cytoplasm / カプシド / Interferon alpha/beta signaling / PIP3 activates AKT signaling / Transcription of SARS-CoV-2 sgRNAs / host cell endoplasmic reticulum-Golgi intermediate compartment / host cell Golgi apparatus / viral nucleocapsid / Translation of Structural Proteins / Virion Assembly and Release / host extracellular space / Induction of Cell-Cell Fusion / Attachment and Entry / host cell perinuclear region of cytoplasm /  ribonucleoprotein complex / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / ribonucleoprotein complex / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity /  RNA binding / extracellular region / identical protein binding / RNA binding / extracellular region / identical protein binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Zhou, R.J. / Ni, X.C. / Lei, J. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Crystal structure of SARS-CoV-2 N-CTD in complex with GMP 著者: Zhou, R.J. / Ni, X.C. / Lei, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7xxk.cif.gz 7xxk.cif.gz | 164.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7xxk.ent.gz pdb7xxk.ent.gz | 127.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7xxk.json.gz 7xxk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xx/7xxk https://data.pdbj.org/pub/pdb/validation_reports/xx/7xxk ftp://data.pdbj.org/pub/pdb/validation_reports/xx/7xxk ftp://data.pdbj.org/pub/pdb/validation_reports/xx/7xxk | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  7c22S S: 精密化の開始モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 6分子 ABCDEF
| #1: タンパク質 |  核タンパク質 / Nucleocapsid protein / Protein N 核タンパク質 / Nucleocapsid protein / Protein N分子量: 13374.036 Da / 分子数: 6 / 断片: C-terminal domain / 由来タイプ: 組換発現 由来: (組換発現)   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2)発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P0DTC9 Escherichia coli (大腸菌) / 参照: UniProt: P0DTC9 |
|---|
-非ポリマー , 8種, 829分子 
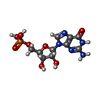











| #2: 化合物 | ChemComp-SCN /  チオシアン酸塩 チオシアン酸塩 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
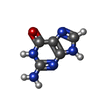
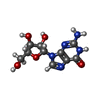
| #3: 化合物 |  グアニル酸 グアニル酸#4: 化合物 | ChemComp-K / #5: 化合物 | ChemComp-CL /  塩化物 塩化物#6: 化合物 | ChemComp-GUN / |  グアニン グアニン#7: 化合物 | #8: 化合物 | ChemComp-GMP / |  グアノシン グアノシン#9: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.03 Å3/Da / 溶媒含有率: 39.48 % |
|---|---|
結晶化 | 温度: 291 K / 手法: 蒸気拡散法, シッティングドロップ法 / 詳細: 0.15 M potassium thiocyanate, 30% w/v PEG MME 2000 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL19U1 / 波長: 0.97852 Å / ビームライン: BL19U1 / 波長: 0.97852 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2020年7月23日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97852 Å / 相対比: 1 : 0.97852 Å / 相対比: 1 |
| 反射 | 解像度: 2→19.91 Å / Num. obs: 45025 / % possible obs: 99.9 % / 冗長度: 13 % / CC1/2: 0.997 / Rmerge(I) obs: 0.184 / Net I/σ(I): 12 |
| 反射 シェル | 解像度: 2→2.05 Å / Rmerge(I) obs: 1.274 / Mean I/σ(I) obs: 2.1 / Num. unique obs: 3254 / CC1/2: 0.791 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 7C22 解像度: 2→19.91 Å / Cor.coef. Fo:Fc: 0.938 / Cor.coef. Fo:Fc free: 0.913 / SU R Cruickshank DPI: 0.196 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.23 / SU Rfree Blow DPI: 0.181 / SU Rfree Cruickshank DPI: 0.171
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 84.21 Å2 / Biso mean: 28.69 Å2 / Biso min: 13.72 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2→19.91 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2→2.01 Å / Rfactor Rfree error: 0 / Total num. of bins used: 51
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj















