+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7uin | ||||||
|---|---|---|---|---|---|---|---|
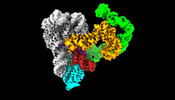
| タイトル | CryoEM Structure of an Group II Intron Retroelement | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | RNA BINDING PROTEIN/RNA/DNA / RNA / intron / group II / maturase / splicing / retrotransposition / RNA BINDING PROTEIN-RNA-DNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Chung, K. / Xu, L. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Structures of a mobile intron retroelement poised to attack its structured DNA target. 著者: Kevin Chung / Ling Xu / Pengxin Chai / Junhui Peng / Swapnil C Devarkar / Anna Marie Pyle /  要旨: Group II introns are ribozymes that catalyze their self-excision and function as retroelements that invade DNA. As retrotransposons, group II introns form ribonucleoprotein (RNP) complexes that roam ...Group II introns are ribozymes that catalyze their self-excision and function as retroelements that invade DNA. As retrotransposons, group II introns form ribonucleoprotein (RNP) complexes that roam the genome, integrating by reversal of forward splicing. Here we show that retrotransposition is achieved by a tertiary complex between a structurally elaborate ribozyme, its protein mobility factor, and a structured DNA substrate. We solved cryo-electron microscopy structures of an intact group IIC intron-maturase retroelement that was poised for integration into a DNA stem-loop motif. By visualizing the RNP before and after DNA targeting, we show that it is primed for attack and fits perfectly with its DNA target. This study reveals design principles of a prototypical retroelement and reinforces the hypothesis that group II introns are ancient elements of genetic diversification. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7uin.cif.gz 7uin.cif.gz | 409.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7uin.ent.gz pdb7uin.ent.gz | 309.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7uin.json.gz 7uin.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7uin_validation.pdf.gz 7uin_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7uin_full_validation.pdf.gz 7uin_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  7uin_validation.xml.gz 7uin_validation.xml.gz | 44.1 KB | 表示 | |
| CIF形式データ |  7uin_validation.cif.gz 7uin_validation.cif.gz | 67.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ui/7uin https://data.pdbj.org/pub/pdb/validation_reports/ui/7uin ftp://data.pdbj.org/pub/pdb/validation_reports/ui/7uin ftp://data.pdbj.org/pub/pdb/validation_reports/ui/7uin | HTTPS FTP |
-関連構造データ
| 関連構造データ | 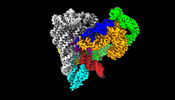 26550MC  7uimC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-RNA鎖 / タンパク質 / DNA鎖 , 3種, 3分子 BDA
| #1: RNA鎖 | 分子量: 206779.859 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) |
|---|---|
| #2: タンパク質 | 分子量: 49083.914 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア)遺伝子: ltrA_2, ltrA, ERS852417_00966, FYL37_05080 / 発現宿主:  |
| #3: DNA鎖 | 分子量: 11403.316 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) |
-非ポリマー , 3種, 54分子 




| #4: 化合物 | ChemComp-MG / #5: 化合物 | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Ternary complex of RNA, protein and DNA / タイプ: COMPLEX / Entity ID: #1-#3 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.260 MDa / 実験値: NO |
| 由来(天然) | 生物種:  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 297 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 500 nm |
| 試料ホルダ | 凍結剤: NITROGEN |
| 撮影 | 電子線照射量: 50.5 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 914099 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj

































































