| 登録情報 | データベース: PDB / ID: 6ph7
|
|---|
| タイトル | Crystal structure of bovine opsin with nerol bound |
|---|
 要素 要素 | - G protein CT2 peptide
- Rhodopsin
 ロドプシン ロドプシン
|
|---|
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  Olfactory Receptor (嗅覚受容体) / Olfactory Receptor (嗅覚受容体) /  Odorant (芳香化合物) Odorant (芳香化合物) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
detection of light stimulus involved in visual perception / retinal cone cell development /  Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane / Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane /  podosome assembly / absorption of visible light / podosome assembly / absorption of visible light /  opsin binding ...detection of light stimulus involved in visual perception / retinal cone cell development / opsin binding ...detection of light stimulus involved in visual perception / retinal cone cell development /  Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane / Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane /  podosome assembly / absorption of visible light / podosome assembly / absorption of visible light /  opsin binding / The canonical retinoid cycle in rods (twilight vision) / detection of chemical stimulus involved in sensory perception of bitter taste / : / G protein-coupled photoreceptor activity / photoreceptor inner segment membrane / G protein-coupled opsin signaling pathway / opsin binding / The canonical retinoid cycle in rods (twilight vision) / detection of chemical stimulus involved in sensory perception of bitter taste / : / G protein-coupled photoreceptor activity / photoreceptor inner segment membrane / G protein-coupled opsin signaling pathway /  11-cis retinal binding / cellular response to light stimulus / G protein-coupled receptor complex / Inactivation, recovery and regulation of the phototransduction cascade / 11-cis retinal binding / cellular response to light stimulus / G protein-coupled receptor complex / Inactivation, recovery and regulation of the phototransduction cascade /  phototransduction, visible light / phototransduction, visible light /  thermotaxis / Activation of the phototransduction cascade / outer membrane / detection of temperature stimulus involved in thermoception / arrestin family protein binding / thermotaxis / Activation of the phototransduction cascade / outer membrane / detection of temperature stimulus involved in thermoception / arrestin family protein binding /  photoreceptor cell maintenance / Ca2+ pathway / photoreceptor outer segment membrane / G alpha (i) signalling events / photoreceptor cell maintenance / Ca2+ pathway / photoreceptor outer segment membrane / G alpha (i) signalling events /  phototransduction / response to light stimulus / photoreceptor outer segment / G-protein alpha-subunit binding / sperm midpiece / photoreceptor inner segment / phototransduction / response to light stimulus / photoreceptor outer segment / G-protein alpha-subunit binding / sperm midpiece / photoreceptor inner segment /  視覚 / guanyl-nucleotide exchange factor activity / G protein-coupled receptor binding / G-protein beta/gamma-subunit complex binding / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / microtubule cytoskeleton organization / photoreceptor disc membrane / 視覚 / guanyl-nucleotide exchange factor activity / G protein-coupled receptor binding / G-protein beta/gamma-subunit complex binding / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / microtubule cytoskeleton organization / photoreceptor disc membrane /  heterotrimeric G-protein complex / cell-cell junction / heterotrimeric G-protein complex / cell-cell junction /  遺伝子発現 / G protein-coupled receptor signaling pathway / 遺伝子発現 / G protein-coupled receptor signaling pathway /  ゴルジ体 / ゴルジ体 /  GTPase activity / GTP binding / zinc ion binding / GTPase activity / GTP binding / zinc ion binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  metal ion binding / metal ion binding /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能  Rhodopsin, N-terminal / Amino terminal of the G-protein receptor rhodopsin / Rhodopsin, N-terminal / Amino terminal of the G-protein receptor rhodopsin /  ロドプシン / ロドプシン /  Opsin / Visual pigments (opsins) retinal binding site / Visual pigments (opsins) retinal binding site. / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / G-protein alpha subunit, group I / Serpentine type 7TM GPCR chemoreceptor Srsx ... Opsin / Visual pigments (opsins) retinal binding site / Visual pigments (opsins) retinal binding site. / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / G-protein alpha subunit, group I / Serpentine type 7TM GPCR chemoreceptor Srsx ... Rhodopsin, N-terminal / Amino terminal of the G-protein receptor rhodopsin / Rhodopsin, N-terminal / Amino terminal of the G-protein receptor rhodopsin /  ロドプシン / ロドプシン /  Opsin / Visual pigments (opsins) retinal binding site / Visual pigments (opsins) retinal binding site. / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / G-protein alpha subunit, group I / Serpentine type 7TM GPCR chemoreceptor Srsx / G-alpha domain profile. / Guanine nucleotide binding protein (G-protein), alpha subunit / G protein alpha subunit, helical insertion / G-protein alpha subunit / G protein alpha subunit / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like / Opsin / Visual pigments (opsins) retinal binding site / Visual pigments (opsins) retinal binding site. / Rhopdopsin 7-helix transmembrane proteins / Rhodopsin 7-helix transmembrane proteins / G-protein alpha subunit, group I / Serpentine type 7TM GPCR chemoreceptor Srsx / G-alpha domain profile. / Guanine nucleotide binding protein (G-protein), alpha subunit / G protein alpha subunit, helical insertion / G-protein alpha subunit / G protein alpha subunit / G-protein coupled receptors family 1 signature. / G protein-coupled receptor, rhodopsin-like /  GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family) / Up-down Bundle / P-loop containing nucleoside triphosphate hydrolase / Mainly Alpha類似検索 - ドメイン・相同性 GPCR, rhodopsin-like, 7TM / G-protein coupled receptors family 1 profile. / 7 transmembrane receptor (rhodopsin family) / Up-down Bundle / P-loop containing nucleoside triphosphate hydrolase / Mainly Alpha類似検索 - ドメイン・相同性(2Z)-3,7-dimethylocta-2,6-dien-1-ol /  パルミチン酸 / パルミチン酸 /  ロドプシン / Guanine nucleotide-binding protein G(t) subunit alpha-2類似検索 - 構成要素 ロドプシン / Guanine nucleotide-binding protein G(t) subunit alpha-2類似検索 - 構成要素 |
|---|
| 生物種 |    Bos taurus (ウシ) Bos taurus (ウシ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.902 Å 分子置換 / 解像度: 2.902 Å |
|---|
 データ登録者 データ登録者 | Eger, B.T. / Morizumi, T. / Ernst, O.P. |
|---|
| 資金援助 |  カナダ, 1件 カナダ, 1件 | 組織 | 認可番号 | 国 |
|---|
| Canadian Institutes of Health Research (CIHR) | Canada Research Excellence Chair |  カナダ カナダ |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Odorant-binding site in visual opsin
著者: Morizumi, T. / Kuroi, K. / Eger, B.T. / Ou, W.L. / Van Eps, N. / Tsukamoto, H. / Furutani, Y. / Ernst, O.P. |
|---|
| 履歴 | | 登録 | 2019年6月25日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2020年7月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / database_PDB_caveat / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_unobs_or_zero_occ_atoms / pdbx_validate_chiral / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _database_PDB_caveat.text / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_unobs_or_zero_occ_atoms.label_asym_id / _pdbx_validate_chiral.auth_asym_id / _pdbx_validate_chiral.auth_seq_id / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2024年3月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / software / struct_conn
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _software.name / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード SIGNALING PROTEIN /
SIGNALING PROTEIN /  Olfactory Receptor (嗅覚受容体) /
Olfactory Receptor (嗅覚受容体) /  Odorant (芳香化合物)
Odorant (芳香化合物) 機能・相同性情報
機能・相同性情報 Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane /
Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane /  podosome assembly / absorption of visible light /
podosome assembly / absorption of visible light /  opsin binding ...detection of light stimulus involved in visual perception / retinal cone cell development /
opsin binding ...detection of light stimulus involved in visual perception / retinal cone cell development /  Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane /
Opsins / VxPx cargo-targeting to cilium / rod bipolar cell differentiation / rod photoreceptor outer segment / sperm head plasma membrane /  podosome assembly / absorption of visible light /
podosome assembly / absorption of visible light /  opsin binding / The canonical retinoid cycle in rods (twilight vision) / detection of chemical stimulus involved in sensory perception of bitter taste / : / G protein-coupled photoreceptor activity / photoreceptor inner segment membrane / G protein-coupled opsin signaling pathway /
opsin binding / The canonical retinoid cycle in rods (twilight vision) / detection of chemical stimulus involved in sensory perception of bitter taste / : / G protein-coupled photoreceptor activity / photoreceptor inner segment membrane / G protein-coupled opsin signaling pathway /  11-cis retinal binding / cellular response to light stimulus / G protein-coupled receptor complex / Inactivation, recovery and regulation of the phototransduction cascade /
11-cis retinal binding / cellular response to light stimulus / G protein-coupled receptor complex / Inactivation, recovery and regulation of the phototransduction cascade /  phototransduction, visible light /
phototransduction, visible light /  thermotaxis / Activation of the phototransduction cascade / outer membrane / detection of temperature stimulus involved in thermoception / arrestin family protein binding /
thermotaxis / Activation of the phototransduction cascade / outer membrane / detection of temperature stimulus involved in thermoception / arrestin family protein binding /  photoreceptor cell maintenance / Ca2+ pathway / photoreceptor outer segment membrane / G alpha (i) signalling events /
photoreceptor cell maintenance / Ca2+ pathway / photoreceptor outer segment membrane / G alpha (i) signalling events /  phototransduction / response to light stimulus / photoreceptor outer segment / G-protein alpha-subunit binding / sperm midpiece / photoreceptor inner segment /
phototransduction / response to light stimulus / photoreceptor outer segment / G-protein alpha-subunit binding / sperm midpiece / photoreceptor inner segment /  視覚 / guanyl-nucleotide exchange factor activity / G protein-coupled receptor binding / G-protein beta/gamma-subunit complex binding / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / microtubule cytoskeleton organization / photoreceptor disc membrane /
視覚 / guanyl-nucleotide exchange factor activity / G protein-coupled receptor binding / G-protein beta/gamma-subunit complex binding / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / microtubule cytoskeleton organization / photoreceptor disc membrane /  heterotrimeric G-protein complex / cell-cell junction /
heterotrimeric G-protein complex / cell-cell junction /  遺伝子発現 / G protein-coupled receptor signaling pathway /
遺伝子発現 / G protein-coupled receptor signaling pathway /  ゴルジ体 /
ゴルジ体 /  GTPase activity / GTP binding / zinc ion binding /
GTPase activity / GTP binding / zinc ion binding /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  metal ion binding /
metal ion binding /  細胞膜
細胞膜
 Bos taurus (ウシ)
Bos taurus (ウシ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.902 Å
分子置換 / 解像度: 2.902 Å  データ登録者
データ登録者 カナダ, 1件
カナダ, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6ph7.cif.gz
6ph7.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6ph7.ent.gz
pdb6ph7.ent.gz PDB形式
PDB形式 6ph7.json.gz
6ph7.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ph/6ph7
https://data.pdbj.org/pub/pdb/validation_reports/ph/6ph7 ftp://data.pdbj.org/pub/pdb/validation_reports/ph/6ph7
ftp://data.pdbj.org/pub/pdb/validation_reports/ph/6ph7 リンク
リンク 集合体
集合体

 要素
要素 ロドプシン
ロドプシン
 Bos taurus (ウシ) / 参照: UniProt: P02699
Bos taurus (ウシ) / 参照: UniProt: P02699
 Bos taurus (ウシ) / 参照: UniProt: P04696*PLUS
Bos taurus (ウシ) / 参照: UniProt: P04696*PLUS
 オリゴ糖 / 分子量: 748.682 Da / 分子数: 1 / 由来タイプ: 組換発現
オリゴ糖 / 分子量: 748.682 Da / 分子数: 1 / 由来タイプ: 組換発現 Octyl glucoside
Octyl glucoside
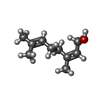

 パルミチン酸
パルミチン酸 ネロール
ネロール X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 23-ID-B / 波長: 1.033 Å
/ ビームライン: 23-ID-B / 波長: 1.033 Å : 1.033 Å / 相対比: 1
: 1.033 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 2.902→39.256 Å / SU ML: 0.36 / 交差検証法: THROUGHOUT / σ(F): 1.35 / 位相誤差: 29.21
分子置換 / 解像度: 2.902→39.256 Å / SU ML: 0.36 / 交差検証法: THROUGHOUT / σ(F): 1.35 / 位相誤差: 29.21  ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj













