+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6bfn | ||||||
|---|---|---|---|---|---|---|---|
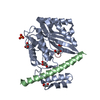
| タイトル | Crystal structure of human IRAK1 | ||||||
 要素 要素 | Interleukin-1 receptor-associated kinase 1 IRAK1 IRAK1 | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / Interleukin-1 (IL-1) receptor-associated kinases TRANSFERASE (転移酵素) / Interleukin-1 (IL-1) receptor-associated kinases | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / regulation of cytokine-mediated signaling pathway / : / protein serine/threonine kinase activity => GO:0004674 / toll-like receptor 2 signaling pathway / : / : / MyD88 dependent cascade initiated on endosome / Transcriptional Regulation by MECP2 / TRAF6 mediated induction of NFkB and MAP kinases upon TLR7/8 or 9 activation ...: / regulation of cytokine-mediated signaling pathway / : / protein serine/threonine kinase activity => GO:0004674 / toll-like receptor 2 signaling pathway / : / : / MyD88 dependent cascade initiated on endosome / Transcriptional Regulation by MECP2 / TRAF6 mediated induction of NFkB and MAP kinases upon TLR7/8 or 9 activation / MyD88 cascade initiated on plasma membrane / toll-like receptor 9 signaling pathway / activation of NF-kappaB-inducing kinase activity / MyD88-dependent toll-like receptor signaling pathway / interleukin-1-mediated signaling pathway / positive regulation of leukocyte adhesion to vascular endothelial cell / MyD88:MAL(TIRAP) cascade initiated on plasma membrane / toll-like receptor 4 signaling pathway /  Toll様受容体 / type I interferon-mediated signaling pathway / negative regulation of NF-kappaB transcription factor activity / positive regulation of type I interferon production / JNK cascade / lipopolysaccharide-mediated signaling pathway / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Toll様受容体 / type I interferon-mediated signaling pathway / negative regulation of NF-kappaB transcription factor activity / positive regulation of type I interferon production / JNK cascade / lipopolysaccharide-mediated signaling pathway / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation /  heat shock protein binding / p75NTR recruits signalling complexes / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / viral process / NF-kB is activated and signals survival / heat shock protein binding / p75NTR recruits signalling complexes / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / viral process / NF-kB is activated and signals survival /  lipid droplet / response to interleukin-1 / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / activated TAK1 mediates p38 MAPK activation / positive regulation of smooth muscle cell proliferation / TAK1-dependent IKK and NF-kappa-B activation / NOD1/2 Signaling Pathway / positive regulation of MAP kinase activity / Interleukin-1 signaling / cytokine-mediated signaling pathway / positive regulation of non-canonical NF-kappaB signal transduction / PIP3 activates AKT signaling / cellular response to heat / positive regulation of NF-kappaB transcription factor activity / lipid droplet / response to interleukin-1 / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / activated TAK1 mediates p38 MAPK activation / positive regulation of smooth muscle cell proliferation / TAK1-dependent IKK and NF-kappa-B activation / NOD1/2 Signaling Pathway / positive regulation of MAP kinase activity / Interleukin-1 signaling / cytokine-mediated signaling pathway / positive regulation of non-canonical NF-kappaB signal transduction / PIP3 activates AKT signaling / cellular response to heat / positive regulation of NF-kappaB transcription factor activity /  kinase activity / cellular response to hypoxia / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / positive regulation of canonical NF-kappaB signal transduction / cellular response to lipopolysaccharide / response to lipopolysaccharide / protein autophosphorylation / endosome membrane / kinase activity / cellular response to hypoxia / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / positive regulation of canonical NF-kappaB signal transduction / cellular response to lipopolysaccharide / response to lipopolysaccharide / protein autophosphorylation / endosome membrane /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / intracellular signal transduction / protein heterodimerization activity / protein kinase activity / intracellular signal transduction / protein heterodimerization activity /  protein phosphorylation / protein serine kinase activity / protein phosphorylation / protein serine kinase activity /  自然免疫系 / protein serine/threonine kinase activity / negative regulation of apoptotic process / protein homodimerization activity / protein-containing complex / 自然免疫系 / protein serine/threonine kinase activity / negative regulation of apoptotic process / protein homodimerization activity / protein-containing complex /  核質 / 核質 /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.26 Å シンクロトロン / 解像度: 2.26 Å | ||||||
 データ登録者 データ登録者 | Wang, L. / Qiao, Q. / Wu, H. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017タイトル: Crystal structure of human IRAK1. 著者: Wang, L. / Qiao, Q. / Ferrao, R. / Shen, C. / Hatcher, J.M. / Buhrlage, S.J. / Gray, N.S. / Wu, H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6bfn.cif.gz 6bfn.cif.gz | 256.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6bfn.ent.gz pdb6bfn.ent.gz | 205.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6bfn.json.gz 6bfn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bf/6bfn https://data.pdbj.org/pub/pdb/validation_reports/bf/6bfn ftp://data.pdbj.org/pub/pdb/validation_reports/bf/6bfn ftp://data.pdbj.org/pub/pdb/validation_reports/bf/6bfn | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  IRAK1 / IRAK-1 IRAK1 / IRAK-1分子量: 37777.289 Da / 分子数: 2 / 断片: UNP residues 194-530 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: IRAK1, IRAK Homo sapiens (ヒト) / 遺伝子: IRAK1, IRAK発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P51617,  non-specific serine/threonine protein kinase non-specific serine/threonine protein kinase#2: 化合物 | #3: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.27 Å3/Da / 溶媒含有率: 45.84 % |
|---|---|
結晶化 | 温度: 289 K / 手法: 蒸発脱水法 詳細: 20% PEG3350, 0.2 M calcium chloride, 0.1 M HEPES, pH 7.0 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 24-ID-E / 波長: 0.979 Å / ビームライン: 24-ID-E / 波長: 0.979 Å |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2016年6月21日 |
| 放射 | モノクロメーター: single crystal Si(220) side bounce プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.979 Å / 相対比: 1 : 0.979 Å / 相対比: 1 |
| 反射 | 解像度: 2.26→88.06 Å / Num. obs: 33471 / % possible obs: 100 % / 冗長度: 12.4 % / Net I/σ(I): 13.3 |
| 反射 シェル | 最高解像度: 2.26 Å |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.26→78.844 Å / SU ML: 0.28 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 22.28
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.26→78.844 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -30.1412 Å / Origin y: 8.9839 Å / Origin z: 17.1108 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj